Как создать сайт в блокноте HTML — пример и инструкция
1.Создайте текстовый документ.
2.По желанию переименуйте файл (у меня он будет называться first-page) и обязательно задайте ему расширение .html вместо .txt).
3.Откройте документ программой Блокнот, Notepad, Notepad++ или другом текстовом редакторе, желательно с подсветкой синтаксиса.
4.Скопируйте и вставьте в него следующий текст.
<!DOCTYPE html> <html> <head> <title>Моя первая страничка</title> </head> <body> Тело HTML-документа (отображается на экране) </body> </html>
5. Сохраните изменения в файле.
6. Откройте first-page.html, только уже не в Блокноте, а в любом доступном вам браузере. На экране должно отобразиться примерно следующее:
Взгляните на html-код своей первой веб-страницы. Посмотрите на результат его работы (страницу в браузере). Постарайтесь понять, за что какая строчка кода отвечает. Читать продолжайте только после попытки, которая обязательно увенчается успехом хотя бы частично.
Теперь проверьте, правильно ли вы этот код поняли. Рассмотрим его.
Это тег, с которого должен начинаться любой html-документ. Он даёт понять интернет-обозревателю, с какой версией языка разметки тот имеет дело. Без этой строки браузер может начать неправильно обрабатывать код. Написав , мы идентифицировали файл как документ стандарта HTML 5, и браузер, проанализировав первую строку, будет обрабатывать остальной код в соответствии с заданным стандартом.
Даёт понять интернет-обозревателю, что всё, находящееся между открывающим и закрывающим тегами — html-код. Вообще использовать эти теги не обязательно, но принято.
«Head» с английского — «Голова». Этот контейнер содержит теги со вспомогательной информацией для веб-браузеров и поисковых систем. Здесь можно задать файлы скриптов, таблицы стилей, ключевые слова и
<title>Моя первая страничка</title>
Заголовок страницы.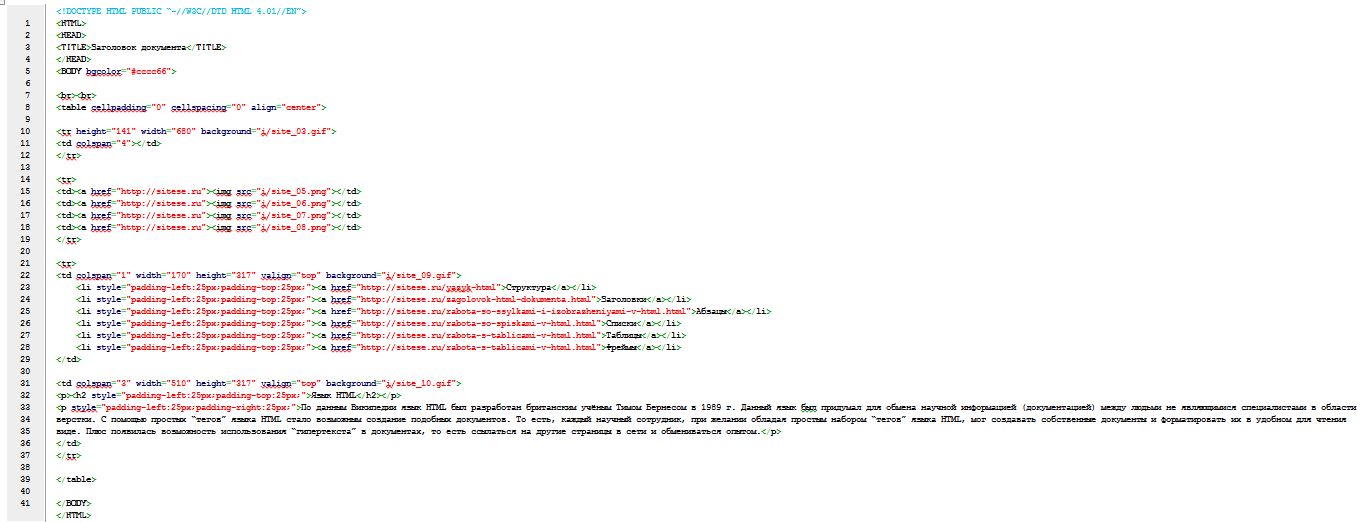 Открыв документ в браузере, вы наверняка обратили внимание на имя вкладки. Браузер вывел его на экран. Заголовок важен не только для поисковых систем, но и для людей, ведь он помогает понять, о чём страница.
Открыв документ в браузере, вы наверняка обратили внимание на имя вкладки. Браузер вывел его на экран. Заголовок важен не только для поисковых систем, но и для людей, ведь он помогает понять, о чём страница.
«Body» — тело. Таким образом у любого html-документа есть голова, содержащая вспомогательную информацию, и тело, внутри которого находится основной код документа. Так как наша страничка — первая, она не должна быть сложной, и поэтому тело документа содержит только текст «Тело HTML-документа (отображается на экране)»
Вы можете поменять надпись на любую другую, и она отобразится в браузере. На экран будет выведен любой находящийся между тегами
и
текст (если только это не теги), так что поздравляю, простые html-странички вы создавать уже научились. Внимательный читатель мог задуматься, почему теги расположены именно так:идёт перед, но закрывающий находится перед закрывающим. Чтобы это понять, рассмотрим предложение: Сайт полезный (называется «Яндекс» (кажется так)).
Кавычки расположены внутри скобок, вторые скобки закрываются перед первыми. Правила вложенности в HTML совпадают с правилами вложенности в русском языке. На основе рассмотренного html-шаблона можно создавать другие страницы, просто меняя информацию внутри контейнеров.
Это был простой пример создания сайта через блокнот, более сложные проекты можно делать с помощью визуального редактора HTML.
Полезные ссылки:
Создаем текстовый веб документ в Блокноте
Неграмотный текстовый документ
В программе Windows Блокнот или аналогичной создайте любой текст, например, «Мой сайт» (рис. 1).
Рис. 1. Создание в Блокноте текстового документа
Сохраните документ командой Файл-Сохранить Как дайте этому документу название index.html (рис. 2). Тип файла – все файлы (это важно).
Рис. 2. Даем документу название
Рис. 3. Открываем файл в блокноте для редактирования
Откройте сохраненный файл с помощью браузера, например, Internet Explorer и просмотрите результат. Так создается простейший веб документ для Интернет. Еще примеры:
Создаем правильный (грамотный) одностраничный html-документ
Разметка элементов языка HTML сопровождаются специальными скобками: открывающая скобка отмечает начало части страницы, а закрывающая отмечает конец этой части страницы. При этом, как правило, документ состоит из двух частей – заголовка и тела.
— заголовок документа.
— тело документа.
В заголовке , используя теги <title> title>, можно прописать название документа.
Для примера создадим простейшую текстовую HTML-страницу в программе Блокнот. Для этого надо создать текстовый файл (*.txt) и сохранить его с расширением *.htm (или *.html). Переименовать *.txt в *.htm удобно в любом файловом менеджере (коммандере), например, в NC или ТС, а просмотреть готовую страницу можно будет в любом браузере. Итак, листинг 1:
Листинг 1. Простейший html — документ
<HTML>
<HEAD>
<title>НАЗВАНИЕ ДОКУМЕНТАtitle>
HEAD>
<BODY>СОДЕРЖАНИЕ ДОКУМЕНТАBODY>
HTML>
Совет
Писать любой тег можно в любом регистре (не важно большими или маленькими буквами). Теги в HTML файле выполняются по порядку, столбиком: сверху — вниз.
Теперь сохраняем и меняем расширение TXT на HTM (рис. 4 и 5).
Рис. 4. Меняем расширение TXT на HTM
Рис. 5. Вид в браузере простейшего HTML-документа, созданного в программе Блокнот
5. Вид в браузере простейшего HTML-документа, созданного в программе Блокнот
Итак, создать одностраничный текстовый документ очень просто. У текстового файла, созданного в программе Блокнот и имеющего по умолчанию расширение для файлов *.txt необходимо изменить расширение для сохраненных на диск файлов на на *.htm или *.html. Имя такого файла для главной страницы сайта принято писать, как index. Таким образом, здесь и далее сохраняем все наши однастаничные веб документы как index.html.
Hyper Text Markup Language (HTML) — стандартный язык гипертекстовой разметки документов, образующих веб-ресурсы. Файлы, написанные на html, обычно имеют расширение имени *.HTM или *.HTML. Разница между html и htm в единственной лишней букве расширения имени файла. Больше распространено именно html, но ничто не мешает сокращать по своему усмотрению. Такое расширение происходит от полного названия языка гипертекстовой разметки – Hyper Text Markup Language. Последнее слово опускается, и получается htm. Ранее это было продиктовано необходимостью ограничения на количество букв в расширении имени файла: старые операционные системы понимали только три знака в расширении файла. Сегодня таких условий нет и от наличия или отсутствия одной последней буквы в веб документе ничего принципиально не меняется. Однако при работе в старых операционных системах и программах под них пользоваться придется расширение *.htm. Итак, буква «l», образуется от последнего слова аббревиатуры HyperText Markup Language. Файлы с расширением имени html более современны и распространены сегодня. Расширение htm устарело.
История HTML
Язык HTML был создан Тимом Бернерсом-Ли в 90-х годах прошлого века. Изначально главной функцией первой версии HTML было корректное, без каких-либо структурных искажений, воспроизведение текста на различных устройствах вывода информации. За прошедшее время HTML претерпел существенные изменения и значительно расширил возможности верстки документов. Язык имеет несколько версий:
Язык имеет несколько версий:
HTML 1.0 — начало 90-х годов. Спецификаций не существует, так как единого официального стандарта языка на тот момент не было. Язык был ориентирован только на обработку текста и применение к нему атрибутов — жирный текст, курсив etc.
HTML 2.0 — появилась поддержка форм.
HTML 3.2 — появилась возможность создания таблиц, отображение математических формул, эффект обтекания изображений.
HTML 4.0 — были отменены некоторые элементы HTML, взамен которых было предложено использовать таблицы стилей CSS. Была добавлена поддержка скриптов и фреймов.
HTML 4.01 — доработанная версия 4.0.
HTML 5 — 2010-й год — нынешнее время.
Правильный DOCTYPE в HTML5 это /SPAN>>
DOCTYPE указывает тип документа (веб документ). Это необходимо, чтобы браузер понимал, согласно какому стандарту отображать текущую страницу. Валидация сайта (проверка сайта на ошибки кода) позволяет следить за правильным отображением сайта в разных браузерах. Валидаторы (сервисы для проверки сайтов на наличие ошибок в структуре документа) проверяют HTML-код на соответствие определенному стандарту, который указан в самом начале любой HTML-страницы первой строчкой.
Элемент предназначен для указания типа текущего документа — DTD (document type definition, описание типа документа). Это необходимо, чтобы браузер понимал, как следует интерпретировать текущую веб-страницу, поскольку HTML существует в нескольких версиях, кроме того, имеется XHTML (EXtensible HyperText Markup Language, расширенный язык разметки гипертекста), похожий на HTML, но различающийся с ним по синтаксису. Чтобы браузер «не путался» и понимал, согласно какому стандарту отображать веб-страницу и необходимо в первой строке кода задавать . Кроме того, перед тегом обычно пишется конструкция doctype, которая указывает версию языка HTML, на которой сделан сайт. Актуальная сейчас версия языка HTML имеет номер пять и доктайп для нее должен выглядеть так — .
Актуальная сейчас версия языка HTML имеет номер пять и доктайп для нее должен выглядеть так — .
Пример грамотного написания элементарной веб страницы с наиболее популярной сегодня кодировкой:
Это заголовок тайтл
Это основное содержимое страницы.
Здесь тег <meta charset=»utf-8″> задает правильное отображение русского языка на веб странице. Как выглядит этот пример в браузере смотрите по данной ссылке.
Еще пример:
Ниже рассматриваются основные теги, применимые к тексту документа.
Заголовки
В языке HTML определены 6 уровней заголовка, нумеруемых по убыванию их размера:
Весь текст внутри таких скобок считается заголовком. Размер h2 соответствует самому крупному шрифту, а размер H6 — самому мелкому. Вот пример документа с использованием заголовков первого (h2) и второго (h3) уровней (Листинг 2):
Листинг 2. Меняем кегль (размер) шрифта
<HTML>
<HEAD>
HEAD>
<BODY>
<h2> Заголовок первого уровняh2>
<h3> Заголовок второго уровняh3>
BODY>
HTML>
Этот код в браузере выглядит так:
Абзацы
Одним из основных элементов страницы являются абзацы. Их можно сравнить с абзацами в книге — каждый абзац начинается с новой «красной» строки. Абзац создается с помощью тега
таким образом:
Это заголовок
Это абзац.
Это еще один абзац.
И еще один абзац.
Так код будет выглядеть в браузере:
Разрыв строк, наподобие абзаца, можно задать записью вида
(этот тег задает переход на новую строку). При этом задавать закрывающие скобки нет необходимости. Но, если закрывающий тег задан, то это не считается ошибкой. Код:
При этом задавать закрывающие скобки нет необходимости. Но, если закрывающий тег задан, то это не считается ошибкой. Код:
Этот код в браузере:
Параграфы (выравнивание текста)
Параграфы вводятся тегом
. С помощью параграфов можно выравнивать текст по центру, по левому (правому) краю, или по ширине. Примеры:текст
текст выровнен по центру.текст
текст выровнен по левому краю.текст
текст выровнен по правому краю.<p align=»justify»> текстp> текст выровнен по ширине (по обоим краям).
Примечание
Если не задавать параграфы, то текст в документе выравнивается по левому краю.
Теперь давайте изучим листинг 3.
Листинг 3. Выравнивание текста документа
<html>
<head>
<title>Выравнивание текстаtitle>
head>
<BR>
<P ALIGN=RIGHT> Текст по правому краюP>
body>
html>
Как видим, здесь имеется два выравнивания: по центру и по правому краю. Остальные теги вам также уже знакомы.
Еще пример:
Цвет фона и текста. Размер и гарнитура шрифта
Цвет фона и текста определяется шестнадцатиричным числом вида #hhhhhh, где первая пара символов является значением насыщенности для красного (Red) цвета, вторая — для зеленого (Green), и третья пара — для синего (Blue) цвета. #00 — минимальная насыщенность, #FF — максимальная насыщенность цвета. Пример изменения насыщенности красного цвета:
Подобрать нужную насыщенность цвета вы можете с помощью таблиц кода цвета HTML, которая есть в Интернет:
На веб страницах можно также использовать текстовое определение цвета: BLUE, RED и т. п., например, чтобы сделать цвет фона страницы серым, можно написать
п., например, чтобы сделать цвет фона страницы серым, можно написать
или
<BODY BGCOLOR=»GRAY»>.
Эти записи равноценны.
Вот несколько примеров цветов в их текстовом и шестнадцатеричном коде:
Для изменения цвета и размера шрифта используется метка . Ее атрибуты:COLOR=»#hhhhhh» – цвет шрифта;
SIZE=»n» — размер шрифта;
FACE=»имя_шрифта» – гарнитура шрифта (arial, verdana, helvetica, sans-serif и другие).
Пример использования метки <FONT> приведен в листинге 4:
Листинг 4. Цвет фона и текста. Размер и гарнитура шрифта
<html>
Текст, размер, гарнитура шрифта
12345
body>
html>
Как видим по тегам, здесь на сером фоне крупным шрифтом красного цвета и без засечек написаны цифры 12345.
Что еще нужно знать для создания сайтов?
Не обольщайтесь тем, что знание HTML для разметки текста – это все, что нужно знать для создания сайтов. Для того, чтобы научиться самостоятельно создавать с нуля сайты, качественные современные сайты вам понадобятся знания следующих технологий:
HTML и CSS
JavaScript, jQuery
PHP и MySQL
любого популярного сайтового движка (WordPress, Joomla, Drupal)
понимание того, что такое хостинг и домен
Чем лучше вы разбираетесь в этих технологиях, тем более востребованным специалистом являетесь.
HTML и CSS
База. То, без чего немыслимо создание сайтов полноценным образом. Именно с языка html начинается создание сайта.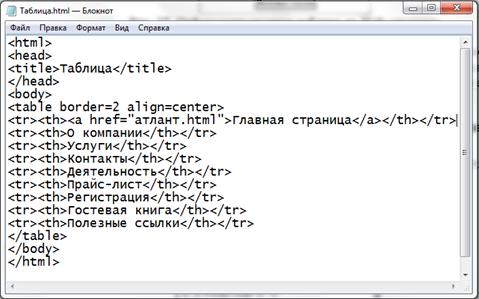 Html — это язык разметки гипертекста, с его помощью создается каркас сайта, его структура, разметка. С помощью же языка CSS все эти блоки получают тот внешний вид, который необходимо. Если сравнить это с работой художника, то html — это набросок рисунка, а css — уже раскрашенный в цвета и полностью готовый рисунок. Но нужны обе технологии, потому что пустоту не оформишь, а без оформления сайты выглядели бы совсем невзрачно. Иными словами, html без css может существовать, а наоборот быть не может. HTML — основа всего и база, именно с его помощью формируется структура страниц.
Html — это язык разметки гипертекста, с его помощью создается каркас сайта, его структура, разметка. С помощью же языка CSS все эти блоки получают тот внешний вид, который необходимо. Если сравнить это с работой художника, то html — это набросок рисунка, а css — уже раскрашенный в цвета и полностью готовый рисунок. Но нужны обе технологии, потому что пустоту не оформишь, а без оформления сайты выглядели бы совсем невзрачно. Иными словами, html без css может существовать, а наоборот быть не может. HTML — основа всего и база, именно с его помощью формируется структура страниц.
JavaScript
JavaScript — с этим языком любой сайт оживает, начинает реагировать на действия посетителя. Что можно сделать на JavaScript? Массу всего. Например, проверку введенных значений в форму, переключатели, слайдеры, всплывающие окна и много-много другого. Для языка JavaScript написана библиотека jQuery, которая во многом значительно упрощает работу по написанию скриптов и управлению элементами.
PHP и MySQL
Это так называемая back-end разработка, а html, css и js — front-end. Бэк-эндом ее назвали потому, что на внешний вид сайтов php код почти никак не влияет, зато для работоспособности ресурса он имеет просто огромное значение. Почти все движки сайтов написаны на нем. Комментирование, регистрация и авторизация, отзывы, корзина товаров, поиск, галерея, загрузка файлов, обработка форм — все это лишь малая часть того, что реализовывается с помощью php. Ну, а MySQl? Это язык, с помощью которого происходят запросы к базе данных. База данных — одна из самых важных вещей в сайтостроении. Как вы думаете, где хранятся все ваши отзывы и комментарии на многочисленных форумах, профили в социальных сетях и статьи на блогах? Все это хранится в базах данных.
CMS
CMS — это сайтовые движки. Это разработанные специалистами веб-программы, которые позволяют управлять сайтом в удобной форме через панель управления. Почти все они написаны на PHP.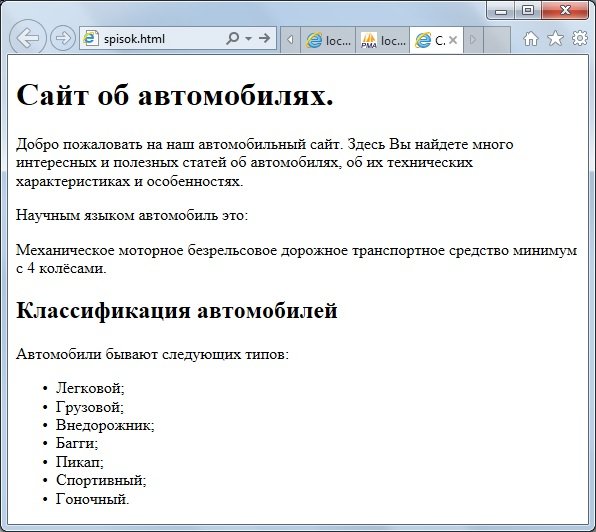 Соответственно, приступать к изучению работы с движком лучше уже после того, как вы постигли хотя бы основы php. Нет смысла изучать много движков, в большинстве случаев вы просто не сможете одновременно усваивать столько информации. Лучше стать специалистом по какому-то одному движку, чем кое-как разбираться во всех. И я рекомендую выбрать движок WordPress. Позднее вы сможете попробовать и другие, когда у вас будет больше опыта.
Соответственно, приступать к изучению работы с движком лучше уже после того, как вы постигли хотя бы основы php. Нет смысла изучать много движков, в большинстве случаев вы просто не сможете одновременно усваивать столько информации. Лучше стать специалистом по какому-то одному движку, чем кое-как разбираться во всех. И я рекомендую выбрать движок WordPress. Позднее вы сможете попробовать и другие, когда у вас будет больше опыта.
Время изучения: От пары дней до многих месяцев, потому что движки имеют разную сложность и порог вхождения. Например, cms Drupal не рекомендуется для новичков ввиду своей сложности.
Хостинг и домен
Вы, как будущий специалист в области сайтостроения, должны прекрасно понимать, как работать с хостингами и доменами и разбираться в терминах, близких к этим понятиям.
Фреймворки и другие инструменты для упрощения разработки
Если вы всегда будете создавать сайты с нуля, используя только сами языки и написание кода, то времени и сил у вас на это уйдет гораздо больше. Реализовать адаптивный дизайн для сайта — это далеко не самая простая задача, особенно если сайт сложный по макету. Но, с css-фреймворком, вроде Bootstrap вы сможете очень легко и быстро делать адаптивные шаблоны, все потому, что в фреймворк вложена система сетки. Это позволяет вам реализовать полную адаптивность, порой не написав ни одной строки css-кода. Параллельный пример можно привести и с javascript. На «голом» javascript порой будет достаточно трудно и попросту неудобно писать какой-то скрипт. Другое дело — использовать библиотеку Jquery, которая упрощает написание скриптов в несколько раз.
Задание 1.
Создайте в Блокноте правильный (грамотный) одностраничный html-документ с любым веб текст про своего домашнего питомца (кошку, собаку, рыбок в аквариуме или хомячка…). Можно написать про своего друга-приятеля (или про подругу). Документ должен иметь следующие параметры своего форматирования:
Фон (BGCOLOR) — серый,
Шрифт (COLOR) — синий,
Текст выровнять (ALIGN) по центру,
Размер шрифта (size) – 16,
Гарнитура (face) – ARIAL,
Название документа (title) – МОЙ КОТ (собака, хомяк и др…).
 Или, «Мой приятель Вася, одноклассница Маша» и т.д и т.п.
Или, «Мой приятель Вася, одноклассница Маша» и т.д и т.п.
Задание 2.
В созданном вами одностраничном текстовом сайте поменяйте атрибуты одного (любого) слова:
Шрифт – зеленый
Размер шрифта – 24
Гарнитура – Times
Кодировка UTF-8
Моя собака
Моя собака очень пугливая, если побежит за котом, то потом наверняка будет от него убегать. Он очень боится громких звукой (Хлопушек, выстрелов и тд). Его порода Йоркширский терьер, маленькая собака которая гоняет больших собак. Он очень предан нашей семье, ждёт нас всегда никуда не уходя, поэтому мы гуляем без поводка. Мы любим его, а он любит нас.
Что такое:
HTML, CSS, JavaScript, PHP, MySQL, CMS, Хостинг, домен, Фреймворки?
В чем разница между веб документами с расширениями html и htm?
Опишите HTML элементы форматирования веб текста — Заголовки h2-H6, Абзацы
.
, ПараграфыКакими командами можно произвести выравнивание HTML текста?
Как задать Цвет фона и текста, какой это цвет BLACK #000000?
Для чего нужен ?
Какое правильное название имени и расширения для сохраненного веб документа?
Как задать размер и гарнитуру (семейство) шрифта?
Поясните следующий код:
12345
Что мы увидим в браузере?
Как сделать веб страницу html с картинкой для чайников
для чайников
Вас заинтересовал вопрос: “Как сделать html страницу?”, то просмотрев данную статью, вы сможете 
HTML (HyperText Markup Language — «язык разметки гипертекста») не является языком программирования, он предназначен для разметки текстовых документов (т.е. с помощью него мы указываем браузеру где будет размещен текст, картинка, таблица и т.п. ).
То, как будут выглядеть картинки, текст и другие элементы на вашей странице, и как они будут располагаться относительно друг друга, определяют метки (тэги).
При помощи тэгов ваша страница (файл) общается с браузером, то есть она ему говорит, что здесь находиться текст определенного шрифта, размера, цвета и т.д. а браузер в свою очередь размещает этот текст, в своем окне, исключительно таким образом каким вы указали при создании страницы.
Теперь мы перейдем к практическому осуществлению нашей задачи “Как сделать html страницу?”. Для этого нам понадобится любой текстовый редактор. Я вам посоветую «Notepad++». Либо можете воспользоваться стандартным блокнотом, только будьте внимательны, когда будете сохранять файл, обязательно сохраняйте его с расширением «html». Название обязательно пропишите латинскими буквами.
Это будет выглядеть так:
«page.html»
И так, открываем наш редактор, и пишем следующий код:
|
<html> |
Сохраняем созданный текст.
Для просмотра созданной страницы html в любом браузере, нужно просто кликнуть на пиктограмме «page.html».
Вставить картинку в html страницу
Чтоб вставить картинку в html страницу, её необходимо предварительно подготовить.
Выбираем нужное изображение, редактируем его либо в документе Word (простой вариант), либо в Photoshop.

Пример:
название папки: user_page
в этой папке:
страница: page.html
картинка: kartinka.jpg
Для вставки картинки в страницу применяется тег <img>.
Для указания пути к картинке в теге используется параметр src=
Вот так будет выглядеть код html, при помощи которого можно вставить картинку в страницу html:
<img src=»/kartinka.jpg»> (при условии: страница и картинка находятся в одной папке).
А это код страницы html с картинкой
|
<html> <img src=»/kartinka.jpg»> |
Если вы выполнили все так, то в браузере будет отражен результат вашей работы.
Оказывается, сделать html страницу совсем не сложно!
Ниже приведены атрибуты для тега <img>, и их предназначение:
|
<img src=»/kartinka.jpg»> |
Без атрибутов — текст по умолчанию находится снизу картинки |
|
<img src=»/kartinka.jpg» align=»right»> |
align=»right» — картинка справа, текст слева |
|
<img src=»/kartinka.jpg» align=»left»> |
align=»left» — картинка слева, текст обтекает справа |
|
<img src=»/kartinka.jpg» align=»bottom»> |
align=»bottom» — как и по умолчанию, текст внизу картинки |
|
<img src=»/kartinka.jpg» align=»middle»> |
align=»middle» — текст посередине картинки |
|
<img src=»/kartinka. |
align=»top» — текст вверху картинки |
|
<img src=»/kartinka.jpg» vspace=»10″> |
vspace — задаёт расстояние между текстом и рисунком (по вертикали). Расстояние задается в пикселях. Pixel — самая маленькая единица изображения (точка). Например, если экран Вашего монитора 800х600, это значит, что он имеет 800 на 600 пикселей (точек). В примере расстояние равно 10 пикселям (точкам). |
|
|
hspace — задаёт горизонтальное расстояние между текстом и рисунком. Расстояние задаётся в пикселях. В примере отступ текста от картинки равен 20 пикселям (точкам) |
|
<img src=»/kartinka.jpg» alt=»Сайт для сайтостроителей»> |
alt — задаёт название (описание) картинки (действует не во всех браузерах. В Internet Explorer – работает!). Работает, если ваш посетитель наведёт мышку на картинку и подержит несколько секунд. В этом конкретном примере появится надпись «Сайт для сайтостроителей». Я рекомендую Вам задавать атрибут alt, особенно тогда, если Ваша картинка является ссылкой (кнопкой). |
|
|
width — задаёт ширину картинки в пикселях. Если же Вы не поставите этот атрибут, то картинка будет иметь реальный размер по ширине. (Рекомендую всегда задавать размеры!!!) |
|
<img src=»/uzeron_pc.jpg»> |
height — задаёт высоту картинки в пикселях. Если же Вы не поставите этот атрибут, то картинка будет иметь реальный размер по высоте. (Рекомендую всегда задавать размеры!!!) |
|
<img src=»/uzeron_pc.jpg» border=»0″> |
border — задаёт рамку вокруг картинки. |
|
<p align=»center»><img src=»/uzeron_pc.jpg»> </p> |
<p></p> — размещает саму картинку туда, куда нам необходимо. Размещение зависит от атрибута align. Есть, например: |
|
<body bgcolor=»#000000″ background= «uzeron_pc.jpg»></body> |
background=»Ваш_фон.jpg»- Ваша картинка будет фоном HTML странички. В атрибуте background=»Ваш_фон.jpg» нужно прописывать путь к Вашей картинке так же, как и в размещении обычной картинки. Я рекомендую оставлять bgcolor для того случая, если не загрузится Ваша картинка. |
Урок информатики на тему «Создание простейшей Web-страницы с использованием текстового редактора»
Тема: «Создание простейшей Web-страницы с использованием текстового редактора»
Класс: 11
Дата проведения:
Тип урока: урок приобретения новых навыков
Цель урока:
— Научиться создавать простые Web-страницы с использованием текстового редактора.
Планируемые образовательные результаты:
Предметные – формирование навыков создания Web-страниц, изучение команд языка HTML.
Метапредметные – развитие умения планировать результаты учебной деятельности, понимания востребованности Web-технологий в современном мире.
Личностные – развитие навыков концентрации внимания, развитие системного мышление, усидчивости.
Задачи:
Познакомиться со структурой html-документа.
Изучить команды для установления расположение текста на экране.
Изучить команды форматирования текста.
Отработать приёмы размещения графики на Web-страницах.
Научиться создавать таблицы.
Изучить построение гиперсвязей.
Материалы урока: персональный компьютер ученика, текст практической работы с пошаговой инструкцией, программа Блокнот, браузер.
Ход урока
I. Организационный момент.
II. Актуализация знаний.
Деловая игра по теме «Анализ наиболее успешных сайтов»
Роли: Руководитель группы аналитиков, докладчик.
Функции руководителя группы аналитиков – организация работы по анализу причин успеха сайтов, имеющих наибольший рейтинг, докладчика – представление отсчёта о проделанной работе.
Игровая цель: выявление причин успеха Web-сайтов, имеющих наибольший рейтинг.
Дидактические цели:
Формирование у учащихся знаний об основных принципах дизайна сайтов.
Выработка умения анализировать Web-сайты с точки зрения их содержательной ценности.
Умение представлять результаты анализа в вербальной форме и при помощи компьютерных программ MS Word, MS PowerPoint.
Совершенствование навыков выступления в аудитории.
Воспитательные цели:
Развитие творческого мышления.
Выработка установки на практическое использование полученных знаний, умений и навыков.
Воспитание индивидуального стиля поведения в процессе взаимодействия с людьми.
Сценарий игры. В ходе проведения деловой игры по данной теме, каждой группе учащихся предлагается проанализировать определенное количество сайтов, занимающих лидирующие позиции в рейтинге, например Rambler’s Top 100.
В качестве примерных вопросов, на которые должны ответить участники игры, можно предложить следующие:
Соответствует ли анализируемый сайт принципам Web-дизайна, как их понимают участники игры? Если нет, то в чём заключается расхождение?
В чём причина популярности того или иного сайта? Что определяет его успех в большей степени?
Какие недостатки можно выделить по каждому из рассмотренных сайтов?
После того как проведён анализ предложенный сайтов и по его результатам создан отчёт, докладчик выступает с сообщением. В данной игре возможно назначение участникам ролей сторонника докладчика, оппонента, критика и конформиста.
Игровой конфликт – противоречие, которое обусловлено разностью позиций игроков.
Способ генерирования событий – смешанный.
Выводы: обосновать основные правила Web-дизайна.
Профессиональный сайт должен корректно отображаться на экране.
Web-страница должна идентично отображаться в различных браузерах (Internet Explorer, Google Chrome, Opera, Yandex и др.).
Все страницы сайта и интегрированные в них графические элементы должны быть минимальными по объёму.
Web-страница обязательно должна включать навигационные элементы.
Весь проект должен быть выдержан в одном дизайнерском стиле.
Использовать только корректные цветовые схемы.
III. Постановка цели и задач
На прошлом уроке мы познакомились с тэгами языка HTML. В ходе деловой игры, рассмотрели правила оформления Web-страниц. Сегодня нам предстоит научиться применять наши знания на практике.
Давайте поставим цели и задачи нашего урока.
Учащиеся ставят цели и задачи урока. Учитель корректирует.
Учитель корректирует.
IV. Практическая работа
Учащиеся получают задание практической работы с пошаговой инструкцией. В качестве справочного материала по языку HTML учащиеся могут использовать сайт htmlbook.ru
Справка:
HTML (от англ. HyperText Markup Language) – язык гипертекстовой разметки, при помощи которого создаются Web-страницы.
Задание № 1. Создание простейшего файла HTML
1. Создайте личную папку, куда вы будете сохранять все файлы своего сайта.
2. Запустите программу Блокнот (Notepad).
3. Наберите в окне программы простейший файл HTML.
Учебный файл HTML
Расписание занятий на вторник
4. Сохраните файл под именем INDEX.HTML (обязательно укажите тип файла HTML при сохранении) в личной папке.
5. Для просмотра Web-страницы используйте любой браузер (Internet Explorer, Google Chrome, Opera, Mozilla Firefox, Yandex или другую). Для этого, не покидая программу Блокнот (сверните окно на панель задач), откройте личную папку и двойным кликом по файлу INDEX.HTML откройте окно браузера.
Результат работы показан на рисунке 1.
Рис. 1 Результат работы
Задание № 2. Управление расположением текста на экране
1. При необходимости откройте текст Web-страницы в Блокноте (1 щелчок правой клавишей мыши по файлу INDEX.HTML, в контекстном меню выбрать команду Открыть с помощью… и выбрать программу Блокнот). При необходимости открыть файл в браузере – двойной клик по значку файла левой клавишей мыши.
2. Внести изменения в файл INDEX.HTML, расположив слова Расписание, занятий, на вторник на разных строках.
Внести изменения в файл INDEX.HTML, расположив слова Расписание, занятий, на вторник на разных строках.
Учебный файл HTML
Расписание
занятий
на вторник
3. Сохраните текст с внесенными изменениями в файле INDEX.HTML (меню Файл | Сохранить). Если у вас уже отображается Web-страница, то вам достаточно переключиться на панели задач на программу браузера и обновить эту страницу (кнопка). Изменилось ли отображение текста на экране? Не удивляйтесь тому, что внешний вид вашей Web-страницы не изменился.
Не забывайте каждый раз сохранять текст Web-страницы при ее корректировке в программе Блокнот и обновлять страницу при ее просмотре в программе браузера.
Задание № 3. Некоторые специальные команды форматирования текста
Существуют специальные команды, выполняющие перевод строки и задающие начало нового абзаца. Кроме того существует команда, запрещающая программе браузера изменять каким-либо образом изменять форматирование текста и позволяет точно воспроизвести на экране заданный
фрагмент текстового файла. Тег перевода строки
отделяет строку от последующего текста или графики. Тег абзаца
тоже отделяет строку, но еще добавляет пустую строку, которая зрительно выделяет абзац.
Оба тега являются одноэлементными, тег
– двойной, т.е. требуется закрывающий тег.
1. Внесите изменения в файл INDEX.HTML
Учебный файл HTML
Расписание
занятий
на вторник
2. Сохраните внесенные изменения, переключитесь на панели задач на программу браузера, обновите Web-страницу.
Как изменилось отображение текста на экране?
Задание № 4. Выделение фрагментов текста
Выделение фрагментов текста
1. Внести изменения в текст файла INDEX.HTML
Учебный файл HTML
Расписание
занятий
на вторник
2. Посмотрите полученную Web-страницу.
Возможно использование комбинированных выделений текста.
Расписание занятий на вторник
Но при этом необходимо помнить следующее правило использования комбинированных тегов:
<Тег_1><Тег_2> … – правильная запись.
<Тег_1><Тег_2> … – ошибочная запись.
Обратите внимание на «вложенность» тегов, она напоминает «вложенность» скобок.
Задание № 5. Задание размеров символов Web-страницы
Существует два способа управления размером текста, отображаемого браузером:
использование стилей заголовка,
задание размера шрифта основного документа или размера текущего шрифта.
Используется шесть тегов заголовков: от
(тег двойной, т.е. требует закрытия). Каждому тегу соответствует конкретный стиль, заданный параметрами настройки браузера.
1. Внесите изменения в файл INDEX.HTML
Учебный файл HTML
занятий на вторник
В результате выполнения, должен получиться результат, представленный на рисунке 2.
Рис. 2 Результаты работы
Задание № 6. Установка размера текущего шрифта
Тег шрифта позволяет задавать размер текущего шрифта в отдельных местах текста в диапазоне от 1 до 7.
1. Внесите изменения в текст INDEX.HTML
Учебный файл HTML
Расписание
занятий на вторник
2. Самостоятельно измените размер текста «занятий на вторник», используя тег .
3. Измените оформление текста HTML-документа, используя тег выделения фрагментов и тег перевода строки и абзаца.
Задание № 7. Установка гарнитуры и цвета шрифта
Тег предоставляет возможности управления гарнитурой, цветом и размером текста. Изменение гарнитуры текста выполняется простым добавлением к тегу атрибута FACE. Например, для отображения текста шрифтом Arial необходимо записать:
Для изменения цвета шрифта можно использовать в теге атрибут COLOR=”X”. Вместо “X” надо подставить английское название цвета в кавычках (“ ”), либо его шестнадцатеричное значение. При задании цвета шестнадцатеричным числом необходимо представить этот цвет разложенным на три составляющие: красную (R – Red), зелѐную (G – Green), синюю (B – blue), каждая из которых имеет значение от 00 до FF. В этом случае мы имеем дело с так называемым форматом RGB.
1. Внесите изменения в файл INDEX.HTML
Учебный файл HTML
Расписание занятий на вторник
Задание № 8. Выравнивание текста по горизонтали
1. Внесите изменения в файл INDEX.HTML
Учебный файл HTML
Расписание
занятий на вторник
Задание № 9. Задание цвета фона и текста
При изображении фона и цвета браузеры используют цвета, установленные по умолчанию, – они заданы параметрами настройки браузера. Если вы хотите задать другие цвета, то это надо сделать в начале файла HTML в теге
Если вы хотите задать другие цвета, то это надо сделать в начале файла HTML в теге
. Атрибут BGCOLOR= определяет цвет фона страницы, атрибут TEXT= задает цвет текста для всей страницы, атрибуты LINK= и VLINK= определяют соответственно цвета непросмотренных и просмотренных ссылок.
1. Внесите изменения в файл INDEX.HTML
Учебный файл HTML
Расписание
занятий на вторник
2. Просмотрите изменения Web-страницы в браузере.
Таблица 1. Коды основных цветов в HTML
Задание № 10. Размещение графики на Web-странице
Тег позволяет вставить изображение на Web-страницу. Оно появится в том месте документа, где находится этот тег. Тег является одиночным. Необходимо помнить, что графические файлы должны находиться в той же папке, что и файл HTML, описывающий страницу. Графика в Web, как правило, распространяется в трех форматах: GIF, JPG, PNG.
Для выполнения следующего задания поместите файл с именем CLOCK.JPG (или другим именем) в рабочую папку.
Следует помнить, что для браузера важно, в каком регистре вы задаете описание имени и типа файла. Выработайте для себя определенное правило и строго следуйте ему. Если вы размещаете файл графического изображения во вложенной папке, то при описании изображения необходимо указывать путь доступа к файлу изображения, отображая вложенность папок.
1. Внесите изменения в файл INDEX.HTML
Учебный файл HTML
Расписание
занятий на вторник
2. Просмотрите изменения вашей Web-страницы в браузере.
На экране вы увидите, что показано на рисунке 3.
Рис.3 Результат работы
Задание № 11. Создание таблицы
Таблица является частью HTML-документа. Она представляет собой прямоугольную сетку, состоящую из вертикальных столбцов и горизонтальных строк. Пересечение строки и столбца называется ячейкой таблицы. Ячейка может содержать в себе текст, графику или другую
таблицу.
Таблица состоит из трех основных частей:
названия таблицы,
заголовков столбцов,
ячеек таблицы.
Таблица в Web-документе заполняется по строкам (слева направо по строке, затем переход на новую строку). Каждая ячейка таблицы должна быть заполнена (хотя бы пробелом, которые используются для создания пустых ячеек).
1. Запустите программу Блокнот и наберите текст следующей Web-страницы. Применяйте приемы копирования при создании таблицы, работая в программе Блокнот.
5 класс
Понедельник
| Урок | 5 «А» | 5 «Б» | 5 «В» |
| 1 | Русский язык | Литература | История |
| 2 | Математика | Информатика | Английский язык |
| 3 | История | Математика | Информатика |
2. Сохраните файл в личной рабочей папке под именем 5.HTML
3. Для просмотра созданной Web-страницы в окне личной рабочей папки двойным щелчком левой клавиши мыши загрузите браузер.
На экране вы увидите то, что показано на рисунке 4.
Рис. 4 Результат работы
Задание № 12. Создание ссылки на другой HTML-документ
Ссылки позволяют щелчком на выделенном слове или фразе перейти к другому файлу.
Опишем ссылку:
5 класс
После имени файла (5.HTML) между символами «>» и «<» располагается текст («5 класс»), на котором должен быть произведен щелчок для перехода к этому файлу.
1. Внесите изменения в файл INDEX.HTML
Учебный файл HTML
Расписание
занятий на вторник
| 5 класс | 6 класс |
| 7 класс | 8 класс |
| 9 класс | 10 класс |
| 11 класс |
2. Сохраните файл INDEX.HTML
3. Просмотрите полученную Web-страницу.
На экране вы увидите то, что изображено на рисунке 5.
Рис. 5 Результаты работы
В ходе выполнения практической работы, учитель прерывает деятельность учащихся для проведения физкультминутки.
V. Подведение итогов урока
Проверка выполненной работы. Оценивание работы учащихся с выставлением оценок.
VI. Домашнее задание
1) Читать по учебнику Семакин Информатика 11 класс (базовый уровень) § 14-15.
2) Создать простой Web-сайт на тему «Моя семья».
VII. Рефлексия
Коллективная оценка эмоционального состояния класса:
Учитель просит поднять руку всех, кто считает, что его эмоциональное состояние: 1) бодрое; 2) усталое.
Индивидуальная самооценка:
На бланках учащиеся заполняют таблицу
Достижения | Затруднения | Предложения |
|
|
|
Примеры создания простейших Web-страниц. Создание элементов диалога (кнопок и др.)
В Интернете есть сайты, размещающие домашние web-странички бесплатно,
например http://www.chat.ru/ (до 20 Мб), http://www.narod.ru/ (до 100 Мб),
http://www.boom.ru/ (до 50 Мб), http://www.fortunecity.com/ (до 100 Мб).
Для размещения страничек используют броузеры Internet Explorer 4.0-5.5, Netscape
Communicator 3.0 — 4.7, Opera 3.60-5.0, а также ftp-броузеры CuteFTP или LeechFTP.
Есть сайты, например http://www.da.ru/, http://www.jump.ru/, позволяющие бесплатно
получать удобные короткие адреса страниц.
Вопросы создания элементов диалога (кнопок и других) рассмотены в
Примере 6 и 7.
Рассмотрим примеры создания простейших Web-страниц.
Пример 1.
<HTML>
<HEAD>
<TITLE>Название документа</TITLE>
</HEAD>
<BODY bgcolor=»teal» text=»aqua»>
Здесь расположен сам Web-документ.
<CENTER><h2><font color=»yellow»>Всем привет!</h2></CENTER></font><P>
<CENTER><font color=»red» size=4>Здравствуй, мир!</CENTER></font>
</BODY>
</HTML>
Здесь атрибут size=4 задает размер шрифта.
Пример 2.
<HTML>
<HEAD>
<TITLE>Поисковые системы Internet.</TITLE>
</HEAD><BODY bgcolor=»navy» text=»yellow»>
<big><i><U>Поисковые системы в Интернете</i></big></U></CENTER>
<hr color=»aqua»>
<font color=»white»>Ниже приведены некоторые популярные зарубежные
поисковые системы и отечественная система Rambler. Для поиска
информации используется окно Search, в которое вводится ключевое
слово и нажимается кнопка Search.<br></font>
Просмотрите примеры:
<a href=»altavist.HTM»>ALTAVIST.HTM</a>,
<a href=»EXCITE.HTM»>EXCITE.HTM</a>,
<a href=»YAHOO.HTM»>YAHOO.HTM</a>!
<hr color=»red»>
1.<IMG SRC=»altavist.gif»>
2.<IMG SRC=»yahoo.gif»>
3.<IMG SRC=»excite.gif»>
4.<IMG SRC=»lycos.gif»><P>
5.<IMG SRC=»infoseek.gif»>
6.<IMG SRC=»rambler.gif»><hr color=»lime»>
<tt><big><U><font color=»aqua»>Используйте поисковые системы для
поиска информации в Интернете!</tt></big></U></font>
</BODY>
</HTML>
Пример 3.
<HTML>
<HEAD>
<TITLE>Эхо Москвы.</TITLE>
</HEAD>
<BODY bgcolor=»purple» text=»lime» link=»yellow»>
<CENTER><u><font color=»yellow»>24 часа в сутки!</u></font><br>
Информация на любые темы!
<font color=»aqua»>Частота на УКВ 73,82 МГц или 91,2 МГц FM.<p></font>
<IMG SRC=»echomsk. gif»><p>
gif»><p>
<i><big><font color=»white»>Слушайте Эхо Москвы!
Остальное видимость!<br></i></big></font>
Адрес в Интернете:
<a href=»http://www.echo.msk.ru»>http://www.echo.msk.ru</a></CENTER>
</BODY>
</HTML>
Пример 4.
<HTML>
<HEAD>
<TITLE>Упорядоченные и неупорядоченные списки</TITLE>
</HEAD>
<BODY BGCOLOR=»navy» text=»yellow»>
<h4><U>Неупорядоченный список</h4></U>
<UL>
<LI>Элемент 1.
<LI>Элемент 2.
<LI>Элемент 3.
</UL>
<HR color=»lime»>
<h4><U>Упорядоченный нумерованный список</h4></U>
<OL>
<LI>Элемент 1.
<LI>Элемент 2.
<LI>Элемент 3.
</OL>
</BODY>
</HTML>
Пример 5.
<HTML>
<HEAD>
<TITLE>Списки определений</TITLE>
</HEAD>
<BODY BGCOLOR=»purple» text=»yellow»>
<h4><u>Списки определений имеют вид:</h4></u>
<DL>
<DT>Название термина 1
<DD>Определение термина 1
<DD>Другое определение термина 1
<DT>Название термина 2
<DD>Определение термина 2
<DD>Другое определение термина 2
<DT>Название термина 3
<DD>Определение термина 3
<DD>Другое определение термина 3
</DL>
<HR color=»lime»>
<address>
Петров И. C., E-mail: [email protected]
C., E-mail: [email protected]
</address>
</BODY>
</HTML>
Тег <address> используется для введения адреса электронной почты
E-mail.
Пример 6.
Элементы диалога (кнопки, области для ввода текста).
<HTML>
<HEAD>
<TITLE>Формы</TITLE></HEAD>
<BASE>
<BODY bgcolor=»silver»>
<FORM>
<CENTER><FONT size=6>Элементы диалога</font></center>
<HR color=»blue»>
<Н2>Элемент ISINDEX</h3>
<ISINDEX prompt=»Cтpoкa для ввода критерия поиска»>
<HR color=»blue»>
<Н2>Элементы INPUT</h3>
<h4> Ввод текстовой строки </h4>
<INPUT type=»text» size=50>
<h4> Ввод пароля </h4>
<INPUT type=»password»>
<h4> Флажки </h4>
<INPUT type=»checkbox» name=»F001″ checked>
<INPUT type=»checkbox» name=»F001″ checked>
<h4> Переключатели </h4>
<INPUT type=»radio» name=»S001″ vаluе=»Первый»>
<INPUT type=»radio» name=»S001″ value=»Второй»>
<INPUT type=»radio» name=»S001″ value=»Третий» checked>
<h4> Кнопка подтверждения ввода </h4>
<INPUT type=»submit» value=»Подтверждение»>
<h4> Кнопка с изображением </h4>
<INPUT type=»image» src=»lycos.gif»>
<h4> Кнопка очистки формы </h4>
<INPUT type=»reset» value=»0чистка»>
<h4> Файл </h4>
<INPUT type=»file» name=»photo» accept=»image/*»>
<HR color=»blue»>
<Н2>Элемент SELECT
<SELECT multiple>
<OPTION value=а>Первый
<OPTION value=Ь>Второй
<OPTION value=с>Третий
<OPTION value=d>Четвертый
</select></h3>
<HR color=»blue»>
<Н2>Элемент TEXTAREA
<TEXTAREA rows=5 cols=30>
Область для ввода текста
</textarea></h3>
<HR color=»blue»>
</FORM>
</BODY></HTML>
Пример 6 с элементамт диалога.![]()
Элементы диалога
<Н2>Элемент ISINDEX
<Н2>Элементы INPUT
Ввод текстовой строки
Ввод пароля
Флажки
Переключатели
Кнопка подтверждения ввода
Кнопка с изображением
Кнопка очистки формы
Файл
<Н2>Элемент SELECT
<Н2>Элемент TEXTAREA Область для ввода текста
Пример 7.
Скрипт.
Программа на языке JavaScript позволяет осуществлять запуск
любой программы или переход к любому файлу при нажатии кнопки.
Здесь mark1() — метка. Для разных кнопок и разных команд надо
указывать разные метки. В данном примере при нажатии кнопки
запускается калькулятор.
<input type=»submit» value=»Калькулятор»>
<script language=»JavaScript»>
<!—
function mark1(){
window.location.href=»/windows/calc.exe»;
}
//—>
</script>
В данном примере при нажатии кнопки просматривается картинка lycos.gif (для Internet Explorer).
Пример 8.
Три вертикальных фрейма.
<html>
<frameset cols=»33%,33%,*» frameborder=»2″>
<frame name=»one» src=»1.htm» frameborder=»2″ scrolling=»yes»>
<frame name=»two» src=»2.htm» frameborder=»2″ scrolling=»yes»>
<frame name=»three» src=»3.htm» frameborder=»2″ scrolling=»yes»>
<noframes>
</noframes>
</frameset>
</html>
Пример 9.
Три горизонтальных фрейма.
<html>
<frameset rows=»33%,33%,*» frameborder=»2″>
<frame name=»one» src=»1.htm» frameborder=»2″ scrolling=»yes»>
<frame name=»two» src=»2.htm» frameborder=»2″ scrolling=»yes»>
<frame name=»three» src=»3.htm» frameborder=»2″ scrolling=»yes»>
<noframes>
<!— For browsers that don’t support frames —>
</noframes>
</frameset>
</html>
Пример 10.
Два вертикальных и два горизонтальных фрейма.
<html>
<frameset cols=»50%,*», rows=»50%,*» frameborder=»2″>
<frame name=»one» src=»1.htm» frameborder=»2″ scrolling=»yes»>
<frame name=»two» src=»2.htm» frameborder=»2″ scrolling=»yes»>
<frame name=»three» src=»3.htm» frameborder=»2″ scrolling=»yes»>
<frame name=»four» src=»4.htm» frameborder=»2″ scrolling=»yes»>
<noframes>
</noframes>
</frameset>
</html>
Создание Web-сайта на языке HTML. Учебное пособие
В примерной программе по информатике и ИКТ на тему “Коммуникационные технологии” отводится всего 12 часов и предлагается создать Web-страничку с использованием шаблонов. На тему “Мультимедийные технологии” выделяется 8 часов, но если в образовательном учреждении информатика изучается на пропедевтическом уровне уже в 5, 6 и 7 классах, то целесообразно перенести изучение мультимедийных технологий в пропедевтический курс и тогда можно увеличить время на изучение коммуникационных технологий, а точнее, можно добавить тему “Создание Web-сайтов на языке HTML” в 8 классе.
Освоение технологии создания сайтов
рекомендуется начать с языка разметки
гипертекста HTML в программе Блокнот, что является
первоосновой в данном направлении. Конечно, за 8
часов невозможно изучить весь язык HTML, но можно
показать назначение и применение основных тегов
языка. Если ученик заинтересуется технологией
создания сайтов, то сможет продолжить изучение
материала самостоятельно или на соответствующих
курсах, кружках или факультативах.
Конечно, за 8
часов невозможно изучить весь язык HTML, но можно
показать назначение и применение основных тегов
языка. Если ученик заинтересуется технологией
создания сайтов, то сможет продолжить изучение
материала самостоятельно или на соответствующих
курсах, кружках или факультативах.
Представленное методическое пособие прошло неоднократную опрабацию в 8-х классах гимназии №441 Фрунзенского р-на Санкт-Петербурга и включает материал для проведения теоретических и практических занятий, выполнение которых сначала демонстрируется через проектор, а затем учащиеся выполняют самостоятельно на компьютере, используя раздаточный материал к уроку.
Для учащихся, которые бысторо выполнили запланированную работу на уроке предусмотрены дополнительные задания.
Эффективно осваивается материал, когда учащиеся вместе с учителем выполняют общий проект, например, по теме из истории Санк-Петербурга “Драматические театры Санкт-Петербурга”, а затем в качестве итоговой работы создают собственный небольшой проект, используя в качестве шаблона сайт, созданный совместно с учителем.
В качестве зачетной работы ученики отвечают на вопросы теста на знание тегов HTML и представляют созданный самостоятельно сайт.
Основные цели обучения: формирование познавательного интереса, развитие интеллектуальных и творческих способностей в области Web-технологий.
Задачи.
Обучающие:
- сформировать систему знаний по технологии создания Web-сайтов;
- обучить языку разметки гипертекста HTML для создания сайтов;
- познакомить с этапами проектной деятельности.
Развивающие:
- развить творческие способности к самовыражению, посредством создания сайтов;
- сформировать умение сопоставлять, искать аналог и осуществлять перенос знаний в новую предметную область Web-технологий;
- развить навыки работы на компьютере
Воспитательные:
- воспитать добросовестное отношение к работе;
- воспитать чувства товарищества и личной ответственности за созданный сайт;
- воспитать художественный и эстетический вкус;
- воспитать грамотного и корректного
пользователя сети Интернет.

Урок 1
1. Общие сведения о Web-сайтах и языке HTML
Публикации во Всемирной паутине (World Wide Web) реализуются в форме Web-сайтов. Web-сайт по своей структуре напоминает журнал, который содержит информацию, посвящённую какой-либо теме или проблеме. Как журнал состоит из печатных страниц, так и Web-сайт состоит из компьютерных Web-страниц, объединённых гиперссылками. Web-страницы могут содержать текст, рисунки, таблицы, мультимедийные и динамические объекты. Создание Web-сайтов можно осуществлять с помощью языка HTML.
HTML – Hyper Text Markup Language — язык разметки гипертекста. HTML ни в коей мере не является языком программирования, он отвечает только за расположение элементов (текста, рисунков) в окне браузера. HTML является языком для создания Web-сайтов во Всемирной паутине. Язык HTML состоит из простых команд – тегов. Теги управляют представлением информации на экране при отображении HTML-документа. Теги заключаются в угловые скобки <>…</> и бывают парные и непарные (одиночные <>).
Документ HTML – это текстовый файл с расширением .html или .htm, содержащий набор тегов.
Браузер – (browser) – программа для просмотра Web-страниц. Широко используют бразеры Microsoft Internet Explorer, Opera и др. Браузер при получении документа HTML выполняет его анализ, строит объектную модель документа, затем результат отображает на мониторе.
2. Структура HTML-документа
<HTML>Секция заголовка Записываются meta-теги, содержащие информацию о
названии страницы, об авторе и др. </HEAD> <BODY> Тело документа Содержит непосредственно информацию страницы: тексты, рисунки, таблицы </BODY> </HTML> |
3. Форматирование символов
Символы, заключенные между следующими тегами отображают:
| <B>….</B> — полужирный
шрифт <I>……</I> — курсивный шрифт <U>……</U> — подчеркнутый шрифт |
<SUB>…</SUB> — нижний
индекс <SUP>…</SUP> — верхний индекс |
Параметры шрифта
Текст, заключенный между тегами <FONT….> ……..</FONT> имеет заданный размер, цвет и гарнитуру. Для этого используются атрибуты: SIZE = значение от 1 до 7 и COLOR = цвет (aqua, black, blue, fuchsia, gray, green, lime, maroon, navy, olive, purple, red, silver, teal, yellow, white), каждому цвету соответствует свой шестнадцатеричный код от 000000 до FFFFFF.
Если в тексте имеется несколько пробелов между словами или символы табуляции, то браузер на экран выводит всего один пробел. Если необходимы дополнительные пробелы то между словами надо добавить — символьный примитив.
Одиночный тег <BR> разрывает текстовый поток и вставляет пустую строку. Несколько таких тегов добавляют несколько пустых строк. Межстрочный интервал – одинарный.
4. Практическое задание №1.
Создание первого HTML-документа “Драматические
театры Санкт-Петербурга”, работа со шрифтами,
задание цвета и размера шрифта. Освоение
технологии работы.
Освоение
технологии работы.
Урок №2
1. Форматирование текста по абзацам
Тег <P> …</P> — начинает абзац с новой строки. Новый абзац отделяется от предыдущего двойным межстрочным интервалом.
Выравнивание текста по абзацам:
| <P ALIGN=CENTER> …</P> — по
центру <P ALIGN=JUSTIFY>…</P> — по ширине |
<P ALIGN=LEFT>… </P> — по
левому краю <P ALIGN=RIGHT>…</P> — по правому краю |
2. Задание цвета всего текста и фона документа
Описываются в начальном теге тела документа <BODY>
<BODY BGCOLOR=цвет фона документа TEXT=цвет текста >.
3. Заголовки разных уровней
Тегами <Hn>….</Hn> оформляют заключенный в них текст. Значения n меняются от 1 до 6, при этом текст выводится от более крупного к более мелкому. Теги <Hn>….</Hn> могут иметь атрибуты ALIGN=CENTER, LEFT, RIGHT.
4. Простые списки
| <UL>……</UL> -
неупорядоченный (ненумерованный) список <OL>……</OL> — упорядоченный (нумерованный) список Часть текста, которая будет оформляться как список, заключается в соответствующие теги, а каждый элемент списка помечается одиночным тегом <LI> |
Виды нумераций списков: <OL TYPE=»Square»> <OL TYPE=»Circle»> <OL TYPE=»Disk»> <OL TYPE=»A»> <OL TYPE=»I»> |
5. Практичекое задание №2
Практичекое задание №2
Форматирование текста по абзацам, цвет фона, заголовки разных уровней, списки.
Урок №3
1. Вставка графических изображений
Всеми браузерами поддерживаются форматы .gif, .jpg. Эти форматы являются растровыми. GIF – поддерживается прозрачность и анимацияю, хорошо подходит для рисованных изображений. JPG – для полноцветных изображений, хорошо подходит для отсканированных изображений и фотографий, анимацию не поддерживает.
Одиночный тег <IMG> вставляет графические изображения в текстовый поток в любом месте:
<IMG SRC=’имя графического файла’>
Необязательные атрибуты тега <img>:
| ALT = альтернативный
текст BORDER = толщина обрамляющей рамки в пикс., 0 нет рамки HEIGHT = высота изображения в пикселах или % WIDTH = ширина изображения в пикселах или % |
HSPACE = свободное
пространство слева и справа от изображения в
пикселах или % VSPACE =- свободное пространство сверху и снизу от изображения в пикселах или % ALIGN = left, right, middle выравнивание изображения |
Чтобы рисунок был по центру, можно использовать тег <CENTER>…….<CENTER>/
2. Практическое задание №3
Вставка и форматирование графических изображений. Самостятельное создание Web-страниц драматических театров.
Урок №4
1. Гиперссылки
Связь с другими документами организуется
тегами <A>…</A>.
<A href=”имя файла на который надо перейти” > текст гиперссылки </A>.
Рисунок так же можно сделать, как гиперссылку, написав:
<A HREF =’имя файла на который переходим’>< IMG SRC =’имя графического файла’></a>
2. Практическое задание №4
Оформление списка театров на главной странице glavn.htm, как гиперссылки на соотвествующие Web-страницы театров.
Урок №5
1. Таблицы
Используют не только для того, чтобы располагать данные в ячейках, сколько с целью позицирования фрагментов текста и изображений друг относительно друга.
С помощью таблиц удобно создавать навигацию по сайту.
| <TABLE>…</TABLE> — вся
таблица. <TR>…</TR> — строка. <TD>…</TD> — ячейка в ряду. |
Пример таблицы из двух строк (рядов), содержащих по две ячейки:
Текст ячейки 1, 1 |
Текст ячейки 1 2 |
Текст ячейки 2, 1 |
Текст ячейки 2, 2 |
| <TABLE> таблица <TR> <TD> текст ячейки 1,1</TD> <TD> текст ячейки 1,2</TD> </TR> первая строка <TR> <TD> текст 2,1 ячейки </TD> <TD> текст 2,2 ячейки </TD> </TR> вторая строка </TABLE> |
Ячейки таблицы могут содержать текст или
изображения, а также текст с HTML-тегами и
гиперссылки. Не следует оставлять ячейки таблицы
незаполненными, надо поместить хотя бы
неразрывный пробел
Не следует оставлять ячейки таблицы
незаполненными, надо поместить хотя бы
неразрывный пробел
Основные атрибуты тегов <TABLE> <TR> и <TD> задают параметры таблицы, строки или ячейки:
ALIGN=left, right, center – выравнивание (<table>, <tr>, <td>)
BGCOLOR=’цвет’ – фоновый цвет (<table>, <tr>, <td>)
HSPACE=значение – свободное пространство слева и справа от таблицы в пикселах (<table>)
VSPACE=значение — свободное пространство сверху и снизу от таблицы в пикселах (<table>)
WIDTH=значение – ширина таблицы (ячейки) – в пикселах, или в процентах (<table>, <td>)
HEIGHT= значение – высоты таблицы (ячейки, строки) – в пикселах, или в процентах (<table>, <td>, <tr>)
BORDER= значение – толщина рамки вокруг таблицы и ее ячеек, по умолчанию значение=1, если значение=0, то рамки нет (<table>, <td>)
BORDECOLOR=’цвет’ – цет рамки (<table,<td>>)
VALIGH=bottom, middle, top – выравнивание содержимого по вертикали (<tr>, <td>)
2. Практичекое задание №5
Создание навигации по сайту в форме таблицы из одной строки
Уроки №6 и №7
1. Секция заголовка <HEAD>, мета-теги
В секции заголовка на каждой странице указывается информация о документе, которая используется при его отображении. Текст, заключенный между тегами <TITLE> … </TITLE>, отображается в заголовке окна браузера.
В секции заголовка обычно помещается и ряд тегов <META> с различными атрибутами, предоставляющими дополнительную информацию (метаинформацию) о Web-сайте:
<HEAD>
<title>Драматические театры Санкт-Петербурга</title>
<meta HTTP-EQUIV=»Content-Type» CONTENT=»text/html;
charset=windows-1251″> — — (указывается тип кодовой
таблицы (windows-1251, Koi8-R и другие),
использованной при подготовке текстовой части
документа.
<meta name=»author» CONTENT=»Смирнова Татьяна, учитель 441 гимназии СПб»> — информация об
<meta name=»Keywords» content=»драматические, театры, Товстоногов, Комиссаржевская, Европа, ул.Рубинштейна”> (указывается через запятую набор ключевых слов, которые могут быть использованы рядом поисковых систем)
</HEAD>
2. Самостоятельная работа над своим проектом
Примерные темы проекта: “Мосты через Неву”, “Реки и каналы Санкт-Петербурга”, “Мосты через каналы Санкт-Петербурга”, “Необычные музеи Санкт-Петербурга”, “Музыкальные театры”, “Филармонии и капелла”, “Технические ВУЗ-ы Санкт-Петербурга”, “Детские театры Санкт-Петербурга”, “Гуманитарные ВУЗ-ы Санкт-Петербурга”, “Пригороды Санкт-Петербурга”, “Музеи Санкт-Петербурга”, “Военные учебные заведения в Санкт-Петербурге” и др.
Необходимо оформить главную страницу сайта по образцу файла glavn.htm, выбрав в качестве навигации по сайту таблицу или список, и 2 — 3 страницы, раскрывающие содержание сайта и содержащие: заголовок, текст и рисунок.
Урок №8
1. Контрольный тест на знание тегов HTML – 15 минут.
2. Рефлексия. Представление проекта и оценивание его учениками класса и учителем – 30 минут.
Практические задания – Приложение 1.
Вопросы контрольного теста – Приложение 2.
Литература для учителя
- Усенков Д.“Уроки Web-мастера”, Москва, БИНОМ, 2004 г.
- Смирнова И. “Начала Web-дизайна, Санкт-Петербург, БХВ, 2004г.
- Томас А.Пауэл “Web-дизайн. Наиболее полное руководство. В подлинике”, 2-е издание, БХВ-СПБ, 2005 г.
- Браун М. “HTML 3.2. Наиболее полное руководство. В подлиннике”, БХВ-СПб, 1999
- Захаркина В.
 В. “Основы создания Web-страниц”,
методическое пособие, СПБ, 2000 г.
В. “Основы создания Web-страниц”,
методическое пособие, СПБ, 2000 г. - К.Ахметов, “Microsoft Internet Explorer 4.0 для всех”, изд-во Компьютер, Москва, 1997 г.
Литература для ученика
- Усенков Д.“Уроки Web-мастера”, Москва, БИНОМ, 2004 г.
- Смирнова И. “Начала Web-дизайна, Санкт-Петербург, БХВ, 2004г.
Перечень Internet-ресурсов
- http://htmlbook.ru — Мержевич Влад. Краткий, но информационно насыщенный учебник по технологии создания сайтов, HTML, CSS, дизайну, графике и др.
- http://html.manual.ru — Городулин Владимир. HTML-справочник.
- http://winchanger.narod.ru — А. Климов. Краткий справочник по тегам HTML-языка.
Создание сайта The Beatles — Создание web-страниц на HTML — Лабораторный практикум — К уроку
| Практикум по созданию веб-сайта средствами HTML Тема: «Сайт «The Beatles». Класс: 8+ Теория и задания (.pdf) Куклина И. Д. Практикум по созданию веб-сайта средствами HTML // Информатика в школе. — М. — 2012. — № 2. — С. 94 — 96. |
Лабораторная работа. Создание web-страницы The Beatles
Предварительная подготовка
Задание.Найдите в Интернете и сохраните в своей папке следующие материалы:
- фотографию группы The Beatles;
- историю создания группы;
- дискографию;
- фильмографию (графические файлы с афишами объедините в папку IMAGES).
Технологическая карта № 1. Первая страница
Задание 1. Создайте пустую страницу с титулом The Beatles.
Теги и параметры:
- тег начала документа в формате HTML <HTML>;
- тег заголовка документа <HEAD>;
- тег заголовка (титула) страницы в окне браузера <TITLE>;
- тег содержимого страницы <BODY>.

Комментарии.
- В своей папке создайте папку Site, в которой будут располагаться все файлы проекта.
- В текстовом редакторе Блокнот создайте типовую структуру HTML-документа, добавив между тегами заголовок главной страницы The Beatles:
<HTML>
<HEAD>
<TITLE> The Beatles </TITLE>
</HEAD>
<BODY>
</BODY>
</HTML> - Сохраните файл под именем index.html в папку Site.
Важно. При сохранении файла в качестве типа файла укажите Все файлы. - Откройте файл index.html в браузере (как правило, двойной щелчок). Появится пустое окно браузера с заголовком.
Задание 2. Добавьте заголовком первого уровня текст (параметры: курсив, выравнивание по центру):
Добро пожаловать на сайт,
посвященный легендарной группе «The Beatles»
- тег заголовка первого уровня <h2>;
Параметр выравнивания align (добавляется внутрь открывающего тега заголовка, абзаца), по центру align=center; - тег курсива <EM>;
- тег перехода на новую строку <BR>.
- Откройте файл index.html в Блокноте для редактирования.
- Между тегами <BODY> </BODY> запишите текст Добро пожаловать на сайт, посвященный легендарной группе «The Beatles».
- Добавьте теги и параметры. Сравните свой текст с образцом:
<HTML>
<HEAD> <TITLE> O Beatles </TITLE> </HEAD>
<BODY>
<h2 align=center><EM>Добро пожаловать на сайт, <BR>посвященный легендарной группе «The Beatles» </EM></h2>
</BODY>
</HTML> - Сохраните файл. В браузере выполните команду Обновить (как правило, клавиша F5).
Задание 3. Добавьте на страницу текст (параметры: выравнивание по ширине; часть слов выделена курсивом):
Об этой группе написано столько, что вряд ли нам удалось бы добавить что-либо новое. На нашем сайте вы найдете историю создания группы, биографии участников, дискографию и фильмографию группы. Мы предлагаем вашему вниманию только достоверную информацию.
На нашем сайте вы найдете историю создания группы, биографии участников, дискографию и фильмографию группы. Мы предлагаем вашему вниманию только достоверную информацию.
Это учебный сайт демонстрационного содержания. Его цель — отработка технологии разметки гипертекстовых документов с помощью языка HTML. Более полную информацию о группе The Beatles вы найдете на других сайтах. Приносим свои извинения.
Теги и параметры:
- тег начала абзаца <P>;
параметр выравнивания абзаца по ширине align=justify
например, <P align=justify> Об этой группе написано столько…</P> - тег курсива <EM>.
Задание 4. По центру добавьте разделительную горизонтальную линию (толщина – 1 пиксель, ширина – 50% рабочего поля, цвет — зеленый).
Теги, параметры:
- тег вставки горизонтальной линии <HR>, закрывающего тега нет;
выравнивание — align; ширина — width; толщина, размер – size; цвет – color;
например, <HR align=center width=50% size=1 color=green>
Задание 5. Добавьте информацию об авторских правах (параметры: размер шрифта 1; шрифт – Arial; форматирование по центру).
© Все права защищены. Инициалы и фамилия, класс. Год.
Теги, параметры и спецсимволы:
- тег параметров шрифта <FONT>;
тип – face, размер – size, цвет – color; - спецсимвол © — ©
например, <P align=center> <FONT face=Arial size=1 > © Все права защищены. </FONT></P>
Задание 6. После заголовка 1-го уровня добавьте фотографию beatles.jpg, предварительно скопировав файл в папку Site (параметры: размер – 50%; выравнивание по центру пустой строки).
Теги и параметры:
- тег изображения <IMG>, закрывающего тега нет;
обязательный параметр – адрес файла с изображением – src;
выравнивание — align, ширина – width;
например, <IMG src=beatles. jpg width=50%>
jpg width=50%>
Самостоятельная работа
| Файл | Заголовок страницы | Временное содержание |
| Info.html | Общая информация | Раздел находится в стадии разработки |
| History.html | История | Раздел находится в стадии разработки |
| Disc.html | Дискография | Раздел находится в стадии разработки |
| Film.html | Фильмография | Раздел находится в стадии разработки |
| Links.html | Источники | Раздел находится в стадии разработки |
Технологическая карта № 2. Использование гиперссылок
Задание. Соедините все страницы сайта гиперссылками.
Задание 1. На главную страницу (файл index.html) добавить строку меню с гиперссылками на страницы сайта (файлы Info.html, History.html, Disc.html, Film.html, Links.html). Расположить меню в первой строке страницы.
Теги и параметры:
- код гиперссылки на внешний ресурс:
<A href=Адрес гиперссылки> Описание web-ресурса </А>
адрес может быть абсолютным, например,
<A href= http://ya.ru> Поиск в Яндексе</А>
или
относительным, например ссылка на страницу, находящуюся в корневой папке сайта:
<A href= history.html> История</А>
Главная | Общая информация
Задание 2. Добавьте меню на остальные страницы сайта (файлы Info.html, History.html, Disc.html, Film.html, Links.html). Обратите внимание, что на каждой из страниц должны быть ссылки на все страницы ресурса, кроме текущей. Например, на странице История должны быть ссылки на все страницы, включая Главную, кроме самой страницы История.
Протестируйте сайт.
Технологическая карта № 3. Создание списков
Задание 1. Оформите страницу Общая информация (файл Info.html) по образцу.
Теги и параметры:
- тег полужирного начертания <strong>;
- тег нумерованного списка <OL><LI> Текст </LI><LI> Текст </LI></OL>;
- параметр start меняет номер начала списка, например, <OL start=«5»>
Задание 2. Оформите страницу История группы (файл History.html) по образцу.
Теги и параметры:
- тег полужирного начертания <STRONG >;|
- тег маркированного списка <UL><LI> Текст </LI><LI> Текст </LI></UL>;
- коды для создания внутренней ссылки:
1) сделать закладку в соответствующем месте страницы при помощи параметра name:
<A name=Имя_закладки> </А>
Например,
<A name= top> </А>2) добавить ссылку на закладку:
<A href=#Имя_закладки>Описание web-ресурса </А>
Например,
<A href=#top> ВВЕРХ </А>
Фрагмент кода:
<h2 align=center><EM>История группы <A name=top></EM></h2>
<h3><FONT color=green>Содержание</FONT></h2>
<UL>
<LI> <A href=#c1>Начало</A>
<LI> <A href=#c2>Название</A>
…
</UL>
<STRONG> НАЧАЛО (1956—1960) <A name=c1></STRONG>
<P align=justify>Весной 1956 года Джон Леннон вместе с товарищами по школе основал группу «The Blackjacks» …
<P><A href=#top>Содержание</A>
Самостоятельная работа
Задание 3. Оформите страницу Дискография (файл Disco.html) по образцу. Протестируйте сайт.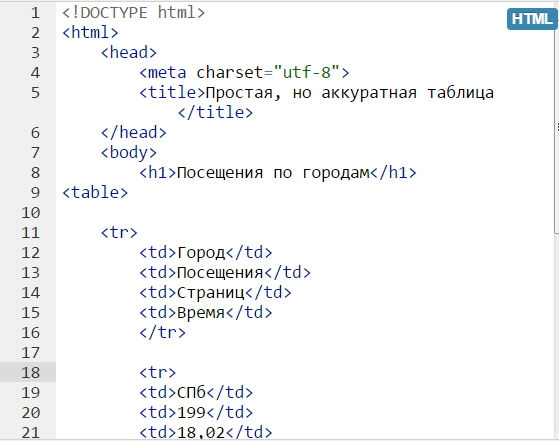
Информация:
ДИСКОГРАФИЯ
1963 Please Please Me, With The Beatles
1964 A Hard Day’s Night, Beatles For Sale
1965 Help!, Rubber Soul
1966 Revolver
1967 Sgt. Pepper’s Lonely Hearts Club Band, Magical Mystery Tour
1968 The Beatles
1969 Yellow Submarine, Abbey Road
1970 Let It Be
Образец (фрагмент):
Фрагмент кода:
<h2 align=center><EM>Дискография</EM></h2>
<P>1963
<UL>
<LI>Please Please Me
<LI>With The Beatles
</UL>
Технологическая карта № 4. Создание таблиц
Задание 1. Оформите страницу Фильмография (файл Film.html) по образцу.Информация:
Теги и параметры:ФИЛЬМОГРАФИЯ
Художественный фильм «A Hard Day’s Night» («Вечер трудного дня»), 1964
Художественный фильм «Help!» (На помощь!), 1965
Художественный фильм «Magical Mystery Tour» («Волшебное таинственное путешествие»), 1967
Мультипликационный фильм «Yellow Submarine» («Жёлтая подводная лодка»), 1968.
Документальный фильм «Let It Be» («Пусть будет так»), 1970
- теги для построения таблиц:
тег начала таблицы <TABLE>,
тег начала строки <TR>,
открывающий тег столбца таблицы <td>; - толщина рамки таблицы в пикселах – параметр border=Число;
- тег изображения <IMG>, закрывающего тега нет;
обязательный параметр – адрес файла с изображением – src;
выравнивание — align; ширина — width
Фрагмент кода:
<TABLE border=1 align=center>
<TR align=center> <TD> Год <TD> Русское название <TD> Оригинальное название <TD> Афиша <TD> Примечание </TR>
<TR align=center> <TD> 1964 <TD> Вечер трудного дня <TD> A Hard Day’s Night <TD> <IMG src=»IMAGES/Film1.
jpg» width=150> <TD> Х/ф </TR>
</TABLE>
Самостоятельная работа
Задание 3. Оформите страницу Источники (файл Links.html), разместив на ней в виде нумерованного списка все использованные информационные ресурсы. При указании Интернет-ресурса используйте внешние ссылки.Информационные источники
- Городулин В. HTML справочник [Электронный ресурс]: перс. сайт. Москва, 2008. URL: http://html.manual.ru/ (дата обращения: 20.09.2011)
- Комолова Н.В. Самоучитель HTML. – СПб.: Питер, 2009. – 272 с.: ил.
- ВикипедиЯ. Свободная энциклопедия [Электронный ресурс]: офиц. сайт.. URL: http://ru.wikipedia.org (дата обращения: 20.09.2011)
Setup — Программатическая информатика материалов
Короче говоря, вам понадобится:
- Файлы упражнений (Скачать)
- Python и Jupyter (рекомендуется: Anaconda)
- Tabula (Бесплатное программное обеспечение)
- API-ключ (создать учетную запись Citrination)
Если у вас есть какие-либо проблемы с этими шагами : Пожалуйста, спросите TA! Мы с радостью поможет вам настроить вашу среду.
Файлы упражнений
На этом семинаре мы выполним ряд упражнений по программированию.У нас есть собрали тетради с упражнениями и установочные файлы в один zip-архив; загрузите следующее и разархивируйте.
Учебное пособие по: использование терминала
Доступ ко многим программным инструментам проще всего получить через программный интерфейс. позвонил на терминал . Этот раздел представляет собой краткое руководство по использованию терминала. Мы будет минимально использовать терминал в этом семинаре, но он будет абсолютно необходимо для настройки вашего компьютера в мастерской. Пожалуйста, спросите ТА, если у вас возникли проблемы с использованием терминала.
Открытие терминала
- Mac:
terminal.— это ваш терминал — вы можете найти его через Spotlight поиск (Command + пробел). app
app - Windows: После того, как вы установили Anaconda python, у вас будет доступ к
Подсказка Anaconda— используйте это как терминал для мастерской. - Linux: если вы работаете в Linux, вы, вероятно, уже регулярно пользуетесь терминалом. На
В Ubuntu ярлык для открытия терминала —
Ctrl + Alt + T.
После открытия терминала вы должны увидеть окно, которое выглядит примерно так: последующий.
Чтобы использовать терминал, вы вручную вводите команды, а затем нажимаете Enter для выполнения.
Смена каталога
При поиске файлов (Mac: Finder, Windows: Explorer, Linux: например, Nautilus),
мы используем программу для просмотра каталогов . Ваш терминал делает нечто подобное
— в любой момент это , расположенный в конкретном каталоге .Последующий
снимок экрана показывает c , висящие d директории ( cd ) в мою папку Downloads ,
затем l i s ting ( ls ) содержимое указанного каталога. Здесь я разместил
архив sizes.zip для семинара. (Примечание: в Windows необходимо использовать
команда dir , а не ls .)
Распаковываю архив plays.zip из терминала командой unzip
упражнения.zip -d plays , что дает следующее:
Затем я могу cd в распакованную папку и установить необходимый Python
пакеты с pip install -r requirements.txt . Однако я могу сделать только это
после того, как я установил Anaconda!
Python и Jupyter
Мы рекомендуем Anaconda
питон, который будет
Обеспечьте почти всем необходимым. Обязательно установите python версии > =
3.0 .
Установка Windows
Если вы работаете в Windows, вам может потребоваться установить Visual C ++. Студия.Щелкните значок
по указанной выше ссылке и установите инструменты.
Студия.Щелкните значок
по указанной выше ссылке и установите инструменты.
Модули Python
После того, как вы установили python, вам нужно будет установить несколько python.
модули для выполнения всех упражнений семинара. Anaconda предоставляет пакет
установщик вызвал pip ; из командной строки вы можете просто выполнить
следующие команды.
Установить модули
В вашем терминале перейдите в каталог, в который вы распаковали упражнения, и выполните команду:
pip install -r требования.текстЭто установит требования для мастерской.
Открытие блокнотов Jupyter
Блокноты Jupyter — это особый способ работы с кодом Python. Они есть
Обозначается расширением файла .ipynb . К сожалению, вы не можете просто
дважды щелкните блокнот Jupyter, чтобы открыть его. Вместо этого вы должны запустить
Клиент Jupyter с вашего терминала. Перейдите в каталог, в котором хотите
Jupyter для запуска (для семинара здесь вы разархивировали содержимое упражнений.zip ) и выполните команду jupyter notebook .
Это откроет веб-страницу в вашем браузере в виде файлового проводника; ты можешь
затем щелкните записные книжки в этом каталоге, чтобы открыть их. Давай и открой check_install.ipynb , который вы запустите в конце установки, чтобы проверить,
все настроено правильно.
Табула
Tabula — это бесплатная программа для освобождения данных из PDF-файлы. Пожалуйста, перейдите по предыдущей ссылке и установите Tabula до семинара.
Настройка ключа API
Чтобы получить доступ к Citrination, вам необходимо предоставить свой ключ API. Однако мы настоятельно не рекомендую вам копировать этот ключ в скрипты, которые вы напишите . Вместо этого мы настроим ваш компьютер для хранения вашего ключа API в общесистемная переменная, и напишите наши сценарии для загрузки этой переменной. В этом разделе будет описано, как это сделать.
Сначала авторизуйтесь
Цитринация
— создайте учетную запись, если вы еще этого не сделали. Нажмите Account Settings , чтобы
найдите свой ключ API.Скопируйте это и выполните любой из двух вариантов, перечисленных ниже.
Простой вариант: текстовый файл
Создайте текстовый файл (.txt) в той же папке, что и файлы вашей мастерской с
имя api.txt . В этот файл вставьте свой ключ API. Это значение будет загружено
упражнения в мастерской, что позволит вам получить доступ к цитринации.
- В Windows это можно сделать с помощью Блокнота
- В Mac OSX можно использовать TextExit; не забудьте включить простой текст
- Формат> Сделать обычный текст; ИЛИ
- нажмите Command + Shift + T
- В Linux вы можете использовать все, что захотите (vim, emacs, nano, у вас есть это)
Обратите внимание, что это упрощенный способ для предоставления вашего ключа API мастерской. материалы.На практике мы рекомендуем следующий расширенный вариант при выполнении любая серьезная работа. Хотя вам не обязательно использовать расширенный вариант для этого семинара, мы настоятельно рекомендуем вам получить следующие рабочие прежде чем приступить к серьезной работе с вашим ключом API.
Расширенный вариант: переменная среды
В OS X или Linux будут работать следующие инструкции:
- В Терминале введите vim ~ / .bash_profile (или воспользуйтесь любым редактором).
- В этом файле нажмите i (режим редактирования) и добавьте строку export CITRINATION_API_KEY = ”your_api_key”.
- Замените your_api_key своим фактическим ключом API; убедитесь, что в нем нет лишних пробелов (пробелов, табуляции и т. д.)
- Сохраните и выйдите (Esc,: wq, Enter).
- Откройте новый Терминал и загрузите этот ноутбук еще раз.
В Windows это еще сложный , но все же управляемый.
Окончательная проверка
Чтобы убедиться, что вы успешно настроили компьютер для семинара, пожалуйста, запустите следующий блокнот jupyter.
Проверьте свою установку
Скачать и запустить check_install.ipynb. Он также включен в файл
plays.zipвыше.Перейдите в папку, в которую вы скачали
check_install.ipynb, выполнить командуjupyter notebook, открытьcheck_install.ipynb, и нажмитеCells> Run All. Если вы можете успешно запустить записная книжка (без ошибок), то вы готовы к мастерской!
Центр биомедицинской информатики | Ученые сети и профилирование (SNAP)
Добро пожаловать
Центр биомедицинской информатики Nemours (BRIC) (бывший центр Bioinformatics Core) предоставляет консультации, обучение и вычислительные ресурсы (как аппаратное, так и программное обеспечение) исследователям биомедицинских исследований Nemours из Делавэра и Флориды.
BRIC управляет основными серверами сети / электронной почты / баз данных для asel.udel.edu и NemoursResearch.org с 10 вычислительными ядрами, 16 ГБ ОЗУ и 5 ТБ дискового пространства для учетных записей пользователей. Дополнительные исследовательские серверы на базе Linux предоставляют 108 ядер ЦП, 462 ГБ ОЗУ и 102 ТБ дискового пространства RAID для исследовательских лабораторий. Для традиционных задач параллельных вычислений можно использовать до 32 ядер ЦП и 256 ГБ памяти, а для проектов, связанных с большими данными, доступен выделенный 192-ядерный вычислительный кластер Apache Spark / Hadoop с 8 узлами емкостью 20 ТБ.Специальная система резервного копирования обеспечивает ежечасное резервное копирование электронной почты, ежедневное резервное копирование файловых томов на диск и еженедельное резервное копирование на магнитную ленту LTO5, которые хранятся вне офиса.
Услуги, предлагаемые BRIC, включают экспериментальный дизайн параллельных, перекрестных, продольных, поперечных и микроматричных исследований; интеллектуальный анализ данных микрочипов и высокопроизводительные исследования секвенирования; и стандартный статистический анализ. BRIC использует множество программных пакетов, включая SPSS, SAS, R, Bioconductor, MeV, CMfinder, MOSAIK и другие.Используя несколько языков программирования (PERL, MySQL, PHP, HTML, Javascript), BRIC также может разрабатывать собственные скрипты и приложения для обучения. BRIC разработал и поддерживает несколько веб-приложений, в том числе TimeKeeper, базу данных по биобезопасности, NED (экспериментальная база данных Nemours, электронная лабораторная записная книжка), регистрация на симпозиумы и SNAP (сайт «Сети и профили ученых» с использованием Drupal, системы управления веб-контентом).
BRIC также управляет установкой Nemours системы REDCap TM (система сбора электронных данных от консорциума REDCap).REDCap — это безопасное веб-приложение, которое позволяет пользователям создавать и управлять онлайн-опросами и базами данных для различных целей, включая базовые, клинические, трансляционные и поведенческие исследования, репозитории и оперативную поддержку. Помимо поддержки этого приложения, BRIC предоставляет обучение и другую поддержку, чтобы сотрудники Nemours могли использовать все инструменты, которые предоставляет этот ресурс.
Свяжитесь с нами для получения дополнительной информации.
Дополнительная информация о REDCap
Вот несколько причин, по которым вам следует рассмотреть возможность использования REDCap вместо электронных таблиц для записи данных исследований:
1.) Доступность — каждый в лаборатории может получить доступ к данным и выполнить ввод данных из любого места.
2.) Безопасность — данные хранятся на наших серверах, резервное копирование производится каждую ночь. Пользователи должны пройти аутентификацию, чтобы просмотреть его. Пользователям могут быть назначены различные уровни разрешений, такие как только чтение, добавление / редактирование данных, удаление записей и т. Д. Пользователям также можно запретить просмотр определенных форм и экспорт определенных полей.
3.) Универсальность — данные или подмножества данных можно легко выбирать, сортировать и экспортировать в форматах, непосредственно готовых для анализа в SPSS, SAS, S + / R или даже в Excel.Расписания могут быть автоматически созданы на основе определенных событий.
4.) Достоверность. Использование такого инструмента, как REDCap, для создания базы данных и форм ввода данных для исследования — ценный первый шаг к уверенности в том, что вы охватили все дьявольские детали, которые присущи разработке действительного исследования. Персонал биоинформатики имеет опыт проектирования баз данных и биостатистики и может помочь убедиться, что ваше исследование хорошо продумано и реализовано.
Чтобы узнать больше о REDCap, отправьте нам электронное письмо или посетите страницу учебных ресурсов REDCap.(Обратите внимание, что вам не нужно входить в REDCap для просмотра обучающих видео).
| Приложение | Размер |
|---|---|
| Ресурс БРИК (формат NIH) | 50,5 КБ |
Степень информатики: Университет Индианы, Кокомо,
, Индианский университет Кокомо,.edu »заполните экран.
Интервью с Джонатаном Ньюсомом, Computer Science ’21: «Я всегда мечтал владеть технологической компанией.Я выбрал информатику, потому что именно так сегодня движется мир. Есть много нового … есть больше рабочих мест и возможностей в области информатики, и я чувствую, что это поможет мне добраться туда, где я должен быть, чтобы осуществить свои мечты.
Джонатан Ньюсом работает в компьютерной лаборатории.
Интервью с Рене Харшбаргер, Computer Science ’21: «Я работаю с компьютерами еще до того, как научился составлять связные предложения. Мне казалось, что это было вполне естественно, и одной из самых больших страстей в моей жизни были видеоигры, поэтому я хочу стать разработчиком видеоигр.
Рене Харшбаргер работает за портативным компьютером в библиотеке.
Кайлер Спенсер, Computer Science ’21, дает интервью: «Я имею в виду, что меня всегда интересовали технологии, компьютеры и тому подобное. Но я думаю, что момент «ВАУ» наступил, когда я понял, как далеко я могу зайти, сколько я могу с этим сделать, понимаешь? Особенно с программированием. Вы действительно можете делать все, что хотите. Если вы можете придумать это в уме, скорее всего, у вас это получится, если вы потратите время и усилия.
Кайлер Спенсер показан работающим со сверстниками в компьютерной лаборатории.
Шадрах Джейкс, специалист по информатике ’18, дает интервью: «Если вы тот, кто любит технологии, изучать новые технологии, оставаться со старыми технологиями, что угодно. Вы можете получить это здесь. Если вы хотите программировать, вы можете получить это здесь. Графический дизайн, вы можете получить это здесь. Степень — это работает практически для всех; это действительно так. Я часто использую это слово, но когда вы смотрите на универсальность программы и используете это как меру для того, какую работу вы можете получить, что вы можете делать после окончания учебы, что делать дальше, — это не дает лучше.”
Показано, как учащиеся работают вместе и независимо в компьютерных классах. Показано, что профессор работает один на один со студентом.
Мохаммад Альмалаг, доцент кафедры информатики Университета Индианы Кокомо, дает интервью: «Если вы посмотрите на то, как мы живем прямо сейчас, все, что мы делаем, связано с технологиями и информатикой, и все информатика основана на работа с технологиями и разработка новых технологий ».
Студенты работают с преподавателями в компьютерном классе.Показано преподавание профессоров в компьютерных классах.
Awny Alnusair, адъюнкт-профессор информатики Университета Индианы Кокомо, дает интервью: «Одна из интересных особенностей АйЮ Кокомо, наши классы очень маленькие, что дает нам возможность узнать наших студентов очень хорошо и как можно скорее. когда вы их узнаете, они узнают друг друга очень хорошо. Это очень важно с точки зрения общения, с точки зрения построения команды и работы над проектами в командных условиях.И наши курсы также преподают профессора, специализирующиеся в области информатики и информатики. Специалисты не учат этому, у нас есть люди, которые специализируются на разработке программного обеспечения, такие как я, у нас есть люди, которые специализируются на сетевой безопасности, больших данных, компьютерных сетях и разработке компьютерных сетей, поэтому разнообразие опыта в нашем отделе действительно будет приносить пользу студентам ».
Шедрах Джейкс дает интервью: «Таким образом, возможности карьерного роста в области информатики и информатики практически безграничны.Количество доступных рабочих мест — при этом очень хорошо оплачиваемых — увеличивается экспоненциально быстрее, чем количество людей, которые могут действительно хорошо их выполнять. Так что попасть на первый этаж, прямо здесь, получить эту степень — это первый шаг к тому, чтобы это сделать ».
На экране появляются слова «Для получения дополнительной информации о компьютерных науках и информатике посетите: iuk.edu/computerscience и iuk.edu/informatics».
Слова: «Университет Индианы Кокомо, Выполнение обещания, iuk.edu »заполните экран.
Компьютерные науки, бакалавр наук
Компьютерные науки
Чтобы получить степень бакалавра наук в области компьютерных наук , студенты получают 120 кредитов по основной последовательности, факультативным курсам и основным требованиям, как указано ниже.
Основные требования к сфере: 36 кредитов
CUS | 1115 | Основы компьютерного программирования I | 3 | 3 | 3 | 90273 |
CUS | 1126 | Введение в структуры данных | 3 | 3 | ||
CUS | 1156 | Методы проектирования программного обеспечения | 3 | 0|||
| CUS 3 | Архитектура компьютера | 3 | ||||
|---|---|---|---|---|---|---|
CUS | 1163 | Операционные системы | 3 | 3 | ||
CUS | 1166 | Программная инженерия | 3 | |||
| 932 CUS Языки | 3 | |||||
CUS | 1185 | Безопасность данных и криптография | 3 | 9281 | 39281 | 03 | 3 |
Дополнительные требования к области: 9 кредитов
(3 кредита из следующих курсов теории информатики)
CUS | 3 | ||
CUS | 1187 Конечные автоматы и формальные языки | 3 | 90US3 932
(6 кредитов от факультативов CUS)
Любые курсы CUS выше 1126
Общие основные требования: 39 кредитов
82 Творческий Процесс | 3 | |||||||
DNY | 1000C | Откройте для себя Нью-Йорк | 3 | |||||
82 | ||||||||
ENG ENG Контекст | 3 | |||||||
FYW | 1000C | Первый год написания | 3 | 932 932 | Общество | 3 | ||
LAC | 1000C * | Язык и культура | 3 | |||||
| 932 Человек Человек | 3 9 0281 | |||||||
PHI | 3000C | Метафизика | 3 | |||||
PHI | 2 | 2 | SPE | 1000C | Публичные выступления для студента колледжа | 3 | ||
THE | 1000C | Христианские взгляды на католицизм | ||||||
THE | 1040 серия | Моральное богословие | 3 | |||||
THE | Elective |
* ИЛИ 6 КРЕДИТОВ НА ЛЮБОМ ВТОРОМ ЯЗЫКЕ
Другие требования к гуманитарным наукам: 32-34 Кредиты
(3 кредита из следующих экономических курсов)
| 1001 903 экономики I | 3 | ||
ECO | 1002 | Принципы экономики II | 3 |
MTH | 1009 | Расчет I | 3 | |||||||
MTH | ||||||||||
MTH | 1013 | 903 323 | ||||||||
MTH | 1014 | Вероятность и статистика II | 3 | Дискретная математика | 3 |
(6 баллов из следующих курсов математики)
MTH | 1011 2 | |||||||||||||||||||||||||||
MTH | 1012 | Линейная алгебра | 3 | |||||||||||||||||||||||||
MTH | Программные приложения для математики | 3 | ||||||||||||||||||||||||||
MTH | 1017 | Mathematical Logic | 1018 | Дифференциальные уравнения | 3 | |||||||||||||||||||||||
MTH | 1019 | Темы в Advanced Calculus | следующие научные курсы)
БЕСПЛАТНЫЕ ВЫБОРЫ (6 КРЕДИТОВ) Журнал химинформатикиMullard A (2016) Расходы на исследования и разработки в области биотехнологий выросли более чем на 15. Nat Rev Drug Discov 15 (7): 447. https://doi.org/10.1038/nrd.2016.135 CAS Статья PubMed Google Scholar Stratmann HG (2010) Плохая медицина: когда медицинские исследования идут не так, как надо. Аналоговый научно-фантастический факт CXXX (9): 20–30 Google Scholar DiMasi JA, Grabowski HG, Hansen RW (2016) Инновации в фармацевтической промышленности: новые оценки затрат на исследования и разработки.J Health Econ 47: 20–33. https://doi.org/10.1016/j.jhealeco.2016.01.012 Артикул PubMed Google Scholar Организация по инновациям в биотехнологии (2016) Показатели успешных клинических разработок, 2006–2015 гг. Ogu CC, Maxa JL (2000) Взаимодействие с лекарствами, вызванное цитохромом p450. Труды медицинского центра Baylor Univ, 13 (4): 421–423. https://doi.org/10.1080/08998280.2000.11 CAS Статья Google Scholar Fox S, Farr-Jones S, Sopchak L, Boggs A, Nicely HW, Khoury R, Biros M (2006) Скрининг с высокой пропускной способностью: обновленная информация о методах и успехах. J Biomol Screen 11 (7): 864–869. https://doi.org/10.1177/10870571062 Артикул PubMed Google Scholar Хьюз Дж. П., Рис С., Калинджян С. Б., Филпотт К. Л. (2011) Принципы раннего открытия лекарств. Br J Pharmacol 162 (6): 1239–1249. https://doi.org/10.1111/j.1476-5381.2010.01127.x CAS Статья PubMed PubMed Central Google Scholar Ruddigkeit L, van Deursen R, Blum LC, Reymond J-L (2012) Перечисление 166 миллиардов органических малых молекул в базе данных химической вселенной gdb-17. J Chem Inform Model 52 (11): 2864–2875. https://doi.org/10.1021/ci300415d CAS Статья Google Scholar Villoutreix BO, Renault N, Lagorce D, Sperandio O, Montes M, Miteva MA (2007) Бесплатные ресурсы для помощи экспериментам по виртуальному скринингу лигандов на основе структур.Curr Protein Pept Sci 8 (4): 381–411 CAS Статья Google Scholar Nantasenamat C, Prachayasittikul V (2015) Максимизация вычислительных инструментов для успешного открытия лекарств. Мнение экспертов по наркотикам Discov 10 (4): 321–329. https://doi.org/10.1517/17460441.2015.1016497 CAS Статья PubMed Google Scholar Фен Б., Симеонов А., Джадхав А., Бабаоглу К., Инглезе Дж., Шойхет Б.К., Остин С.П. (2007) Высокопроизводительный скрининг для ингибирования на основе агрегации в большой библиотеке соединений.J Med Chem 50 (10): 2385–2390. https://doi.org/10.1021/jm061317y CAS Статья PubMed Google Scholar Soares KM, Blackmon N, Shun TY, Shinde SN, Takyi HK, Wipf P, Lazo JS, Johnston PA (2010) Профилирование хранилища малых молекул для соединений, которые генерируют H 2 O 2 за счет окислительно-восстановительного цикла в восстанавливающих средах. Пробирный препарат Dev Technol 8 (2): 152–174. https://doi.org/10.1089/adt.2009.0247 CAS Статья PubMed PubMed Central Google Scholar Янг Д., Мартин Т., Венкатапати Р., Хартен П. (2008) Правильны ли химические структуры в вашем QSAR? QSAR Combinatorial Sci 27 (11–12): 1337–1345. https://doi.org/10.1002/qsar.200810084 CAS Статья Google Scholar Zhao L, Wang W, Sedykh A, Zhu H (2017) Экспериментальные ошибки в наборах моделирования QSAR: что мы можем и чего не можем.ACS Omega 2 (6): 2805–2812. https://doi.org/10.1021/acsomega.7b00274 CAS Статья PubMed PubMed Central Google Scholar Clark RD (2019) Путь к воспроизводимости нового поколения в хеминформатике. Журнал Cheminform 11:62. https://doi.org/10.1186/s13321-019-0385-0 CAS Статья PubMed Central Google Scholar Уолтерс П. (2019) Где код? http: // Practicalcheminformatics.blogspot.com/2019/05/wheres-code.html. По состоянию на 1 ноября 2019 г. Гарабедян Т.Е. (1997) Лабораторный учет. Nat Biotechnol 15 (8): 799–800. https://doi.org/10.1038/nbt0897-799 CAS Статья PubMed Google Scholar Plavén-Sigray P, Matheson GJ, Schiffler BC, Thompson WH (2017) Читаемость научных текстов со временем снижается. eLife. https://doi.org/10.7554/eLife.27725 Артикул PubMed PubMed Central Google Scholar Dirnagl U, Przesdzing I (2016) Карманный справочник по электронным лабораторным блокнотам в академических науках о жизни. F1000 Res 5: 2 https://doi.org/10.12688/f1000research.7628.1 Артикул Google Scholar Rubacha M, Rattan AK, Hosselet SC (2011) Обзор электронных лабораторных ноутбуков, доступных сегодня на рынке. J Lab Autom 16 (1): 90–98. https://doi.org/10.1016/j.jala.2009.01.002 CAS Статья PubMed Google Scholar Mascarelli A (2014) Инструменты исследования: сразу со страницы. Nature 507 (7493): 523–525. https://doi.org/10.1038/nj7493-523a CAS Статья PubMed Google Scholar Schnell S (2015) Десять простых правил для лабораторной записной книжки компьютерного биолога. PLoS Comput Biol 11 (9): 1004385. https://doi.org/10.1371/journal.pcbi.1004385 CAS Статья Google Scholar Bradley J-C, Neylon C (2008) Данные на дисплее. Интервью Кэтрин Сандерсон. Природа 455 (7211): 273. https://doi.org/10.1038/455273a CAS Статья PubMed Google Scholar Батлер Д. (2005) Электронные записные книжки: новый лист. Nature 436 (7047): 20–21. https://doi.org/10.1038/436020a CAS Статья PubMed Google Scholar Project Jupyter (2019) Записная книжка Jupyter. http://www.jupyter.org/. По состоянию на 9 января 2019 г. Project Jupyter (2019) nbviewer. http://nbviewer.jupyter.org/. По состоянию на 9 января 2019 г. Freeman Lab (2019) Binder. http://mybinder.org/. По состоянию на 9 января 2019 г. Google (2019) Colaboratory. https://colab.research.google.com/. По состоянию на 9 января 2019 г. Baker M (2016) 1500 ученых закрывают глаза на воспроизводимость.Nature 533 (7604): 452–454. https://doi.org/10.1038/533452a CAS Статья PubMed Google Scholar Head ML, Holman L, Lanfear R, Kahn AT, Jennions MD (2015) Масштабы и последствия p-хакинга в науке. PLoS Biol 13 (3): 1002106. https://doi.org/10.1371/journal.pbio.1002106 CAS Статья Google Scholar Simonsohn U, Nelson LD, Simmons JP (2014) P-образная кривая: ключ к ящику с файлами.J Exp Psychol Gen 143 (2): 534–547. https://doi.org/10.1037/a0033242 Артикул PubMed Google Scholar Иоаннидис JPA (2008) Влияние формальной статистической значимости на надежность наблюдательных ассоциаций. Am J Epidemiol 168 (4): 374–83384. https://doi.org/10.1093/aje/kwn156 Артикул PubMed Google Scholar Risch NJ (2000) Поиск генетических детерминант в новом тысячелетии.Nature 405 (6788): 847–856. https://doi.org/10.1038/35015718 CAS Статья PubMed Google Scholar Wacholder S, Chanock S, Garcia-Closas M, El Ghormli L, Rothman N (2004) Оценка вероятности того, что положительный отчет является ложным: подход к исследованиям молекулярной эпидемиологии. J Natl Cancer Inst 96 (6): 434–442 Статья Google Scholar Wilkinson MD, Dumontier M, Aalbersberg IJ, Appleton G, Axton M, Baak A, Blomberg N, Boiten JW, da Silva Santos LB, Bourne PE, Bouwman J, Brookes AJ, Clark T, Crosas M, Dillo I, Dumon O , Edmunds S, Evelo CT, Finkers R, Gonzalez-Beltran A, Gray AJ, Groth P, Goble C, Grethe JS, Heringa J, 't Hoen PA, Hooft R, Kuhn T., Kok R, Kok J, Lusher SJ, Martone ME, Mons A, Packer AL, Persson B, Rocca-Serra P, Roos M, van Schaik R, Sansone SA, Schultes E, Sengstag T, Slater T, Strawn G, Swertz MA, Thompson M, van der Lei J, van Mulligen E, Velterop J, Waagmeester A, Wittenburg P, Wolstencroft K, Zhao J, Mons B (2016) Руководящие принципы FAIR для управления научными данными и рационального использования.Научные данные 3: 1600 18. https://doi.org/10.1038/sdata.2016.18 Артикул PubMed PubMed Central Google Scholar Guha R, Willighagen E (2017) Помощь в улучшении практики хеминформатики. Журнал Cheminform 9 (1): 40. https://doi.org/10.1186/s13321-017-0217-z Артикул PubMed PubMed Central Google Scholar Collin’s English Dictionary (2019) Reproduce.http://www.dictionary.com/browse/reproducibility. По состоянию на 9 января 2019 г. Schwab M, Karrenbach M, Claerbout J (2000) Обеспечение воспроизводимости научных вычислений. Comput Sci Eng 2: 61–67 Статья Google Scholar Casadevall A, Fang FC (2010) Воспроизводимая наука. Заражение иммунной 78 (12): 4972–4975. https://doi.org/10.1128/IAI.00908-10 CAS Статья PubMed PubMed Central Google Scholar Kerr Bernal S (2006) Огромный снежный ком мошенничества и обмана. Дж. Андрол 27 (3): 313–315. https://doi.org/10.2164/jandrol.06007 Артикул PubMed Google Scholar Объединенный комитет руководств по метрологии (2008) Оценка данных измерений - Руководство по выражению неопределенности измерения. https://www.bipm.org/utils/common/documents/jcgm/JCGM_100_2008_E.pdf. По состоянию на 1 ноября 2019 г. Oudeyer P-Y, Merrick K (2016) Вычислительное моделирование в различных дисциплинах. Информационный бюллетень IEEE Cogn Dev Syst 13 (2): 1 Google Scholar Taylor CF, Field D, Sansone SA, Aerts J, Apweiler R, Ashburner M, Ball CA, Binz PA, Bogue M, Booth T, Brazma A, Brinkman RR, Michael Clark A, Deutsch EW, Fiehn O, Fostel J, Ghazal P, Gibson F, Gray T, Grimes G, Hancock JM, Hardy NW, Hermjakob H, Julian RK, Kane M, Kettner C, Kinsinger C, Kolker E, Kuiper M, Le Novere N, Leebens- Мак Дж., Льюис С. Е., Лорд П., Мэллон А. М., Мартандан Н., Масуя Х, МакНалли Р., Мехрле А., Моррисон Н., Орчард С., Квакенбуш Дж., Ризи Дж. М., Робертсон Д. Г., Рокка-Серра П., Родригес Х, Розенфельдер Х, Santoyo-Lopez J, Scheuermann RH, Schober D, Smith B, Snape J, Stoeckert CJ, Tipton K, Sterk P, Untergasser A, Vandesompele J, Wiemann S (2008) Продвижение согласованных минимальных руководящих принципов отчетности для биологических и биомедицинских исследований: MIBBI проект.Nat Biotechnol 26 (8): 889–896. https://doi.org/10.1038/nbt.1411 CAS Статья PubMed PubMed Central Google Scholar Hermjakob H, Montecchi-Palazzi L, Bader G, Wojcik J, Salwinski L, Ceol A, Moore S, Orchard S, Sarkans U, von Mering C, Roechert B, Poux S, Jung E, Mersch H , Kersey P, Lappe M, Li Y, Zeng R, Rana D, Nikolski M, Husi H, Brun C, Shanker K, Grant SG, Sander C, Bork P, Zhu W, Pandey A, Brazma A, Jacq B, Vidal M, Sherman D, Legrain P, Cesareni G, Xenarios I, Eisenberg D, Steipe B, Hogue C, Apweiler R (2004) Формат молекулярного взаимодействия HUPO PSI - стандарт сообщества для представления данных о взаимодействии белков.Nat Biotechnol 22 (2): 177–183. https://doi.org/10.1038/nbt926 CAS Статья PubMed Google Scholar Демир Э., Кэри М.П., Палей С., Фукуда К., Лемер К., Вастрик И., Ву Дж., Д'Эустахио П., Шефер С., Лучано Дж., Шехерер Ф, Мартинес-Флорес И., Ху З., Хименес -Jacinto V, Joshi-Tope G, Kandasamy K, Lopez-Fuentes AC, Mi H, Pichler E, Rodchenkov I, Splendiani A, Tkachev S, Zucker J, Gopinath G, Rajasimha H, Ramakrishnan R, Shah I, Syed M, Анвар Н., Бабур О., Блинов М., Браунер Е., Корвин Д., Дональдсон С., Гиббонс Ф., Голдберг Р., Хорнбек П., Луна А., Мюррей-Руст П., Нойман Е., Рубенакер О., Рубенакер О., Самвальд М., ван Ирсель М. , Wimalaratne S, Allen K, Braun B, Whirl-Carrillo M, Cheung KH, Dahlquist K, Finney A, Gillespie M, Glass E, Gong L, Haw R, Honig M, Hubaut O, Kane D, Krupa S, Kutmon M , Леонард Дж., Маркс Д., Мерберг Д., Петри В., Пико А., Равенскрофт Д., Рен Л., Шах Н., Саншайн М., Танг Р., Уэйли Р., Летовский С., Буетоу К. Х., Ржецкий А., Шахтер В., Собрал Б. С., Догрусоз U, McWeeney S, Aladjem M, Birney E, Collado-Vides J, Goto S, Hucka M, Le Novere N, Maltsev N, Pandey A, Thomas P, Wingender E, Karp PD, Sander C, Bader GD (2010) Стандарт сообщества BioPAX для обмена данными о путях.Nat Biotechnol 28 (9): 935–942. https://doi.org/10.1038/nbt.1666 CAS Статья PubMed PubMed Central Google Scholar Проект Wf4Ever (2019) Репозиторий Wf4Ever на github. http://wf4ever.github.io/. По состоянию на 9 января 2019 г. Cooper J, Vik JO, Waltemath D (2015) Призыв к виртуальным экспериментам: ускорение научного процесса. Прогр Биофиз Мол Биол 117 (1): 99–106. https://doi.org/10.1016 / j.pbiomolbio.2014.10.001 Артикул Google Scholar Gentleman RC, Carey VJ, Bates DM, Bolstad B, Dettling M, Dudoit S, Ellis B, Gautier L, Ge Y, Gentry J et al (2004) Bioconductor: открытая разработка программного обеспечения для вычислительной биологии и биоинформатики . Геном Биол 5 (10): 80. https://doi.org/10.1186/gb-2004-5-10-r80 Артикул Google Scholar Бланкенберг Д., Фон Кустер Г., Кораор Н., Ананда Г., Лазарус Р., Манган М., Некрутенко А., Тейлор Дж. (2010) Галактика: веб-инструмент анализа генома для экспериментаторов. Curr Protoc Mol Biol Глава 19: 19–10121. https://doi.org/10.1002/0471142727.mb1910s89 Артикул Google Scholar Giardine B, Riemer C, Hardison RC, Burhans R, Elnitski L, Shah P, Zhang Y, Blankenberg D, Albert I, Taylor J et al (2005) Galaxy: платформа для интерактивного крупномасштабного генома анализ.Genome Res 15 (10): 1451–1455. https://doi.org/10.1101/gr.4086505 CAS Статья PubMed PubMed Central Google Scholar Goecks J, Nekrutenko A, Taylor J (2010) Galaxy Team: Galaxy: комплексный подход для поддержки доступных, воспроизводимых и прозрачных вычислительных исследований в науках о жизни. Геном Биол 11 (8): 86. https://doi.org/10.1186/gb-2010-11-8-r86 Артикул Google Scholar Bouckaert R, Heled J, Kühnert D, Vaughan T, Wu C-H, Xie D, Suchard MA, Rambaut A, Drummond AJ (2014) Beast 2: программная платформа для байесовского эволюционного анализа. PLoS Comput Biol 10 (4): 1003537. https://doi.org/10.1371/journal.pcbi.1003537 CAS Статья Google Scholar Bouckaert R, Vaughan TG, Barido-Sottani J, Duchêne S, Fourment M, Gavryushkina A, Heled J, Jones G, Kühnert D, De Maio N, Matschiner M, Mendes FK, Müller NF, Ogilvie HA , дю Плесси Л., Попинга А., Рамбо А., Расмуссен Д., Сиверони И., Сушард М. А., Ву СН, Се Д., Чжан С., Стадлер Т., Драммонд А. Дж. (2019) Зверь 2.5: Передовая программная платформа для байесовского эволюционного анализа. PLoS Comput Biol 15 (4): 1006650. https://doi.org/10.1371/journal.pcbi.1006650 CAS Статья Google Scholar Teytelman Ltocols.io - репозиторий научных методов №1 Группа экспертов высокого уровня по научным данным (2010) На волне - как Европа может извлечь выгоду из растущего потока научных данных. https: //www.fosteropenscience.ЕС / содержание / верховая-волна-как-европа-может-получить-рост-прилив-научные-данные /. По состоянию на 9 января 2019 г. Национальные институты здравоохранения (2019 г.) Заявление о политике в отношении грантов NIH. https://grants.nih.gov/policy/nihgps/index.htm. По состоянию на 9 января 2019 г. NordForsk (2019) Открытый доступ к данным исследований - состояние, проблемы и перспективы. https://www.nordforsk.org/en/publications/publications_container/open-access-to-research-data-2013-status-issues-and-outlook/. По состоянию на 9 января 2019 г. Borgman CL (2015) Большие данные, мало данных, нет данных: стипендия в сетевом мире. MIT Press, Кембридж Google Scholar Марголис Р., Дерр Л., Данн М., Хуэрта М., Ларкин Дж., Шихан Дж., Гайер М., Грин ED (2014) Инициатива национальных институтов здравоохранения «большие данные к знаниям» (bd2k): использование больших биомедицинских ресурсов данные. J Am Med Inform Assoc. 21 (6): 957–958. https://doi.org/10.1136/amiajnl-2014-002974 Артикул PubMed PubMed Central Google Scholar Pasquetto IV, Randles BM, Borgman CL (2017) О повторном использовании научных данных. Data Sci J. https://doi.org/10.5334/dsj-2017-008 Артикул Google Scholar Wallis JC, Rolando E, Borgman CL (2013) Если мы поделимся данными, будет ли кто-нибудь их использовать? обмен и повторное использование данных в «длинном хвосте» науки и технологий. PLoS ONE 8 (7): 67332. https://doi.org/10.1371/journal.pone.0067332 CAS Статья Google Scholar Чаван В., Пенев Л. (2011) Информационный документ: механизм стимулирования публикации данных в науке о биоразнообразии. BMC Bioinform 12 Приложение 15: 2. https://doi.org/10.1186/1471-2105-12-S15-S2 Артикул Google Scholar Горголевски К.Дж., Маргулис Д.С., Милхэм М.П. (2013) Обеспечение общего доступа к данным: решение на основе публикаций. Front Neurosci 7: 9. https://doi.org/10.3389/fnins.2013.00009 Артикул PubMed PubMed Central Google Scholar Searls DB (2010) Корни биоинформатики. PLoS Comput Biol 6 (6): 1000809. https://doi.org/10.1371/journal.pcbi.1000809 CAS Статья Google Scholar Канвал С., Хан Ф.З., Лони А., Синнотт Р.О. (2017) Исследование воспроизводимости и отслеживание происхождения - тематическое исследование геномного рабочего процесса. BMC Bioinform 18 (1): 337. https://doi.org/10.1186/s12859-017-1747-0 Артикул Google Scholar Ким И-М, Полайн Дж-Б, Дюма Дж. (2017) Эксперименты с воспроизводимостью в биоинформатике. BioRxiv. https://doi.org/10.1101/143503 Артикул Google Scholar Sandve GK, Nekrutenko A, Taylor J, Hovig E (2013) Десять простых правил для воспроизводимых вычислительных исследований. PLoS Comput Biol 9 (10): 1003285. https://doi.org/10.1371/journal.pcbi.1003285 Артикул Google Scholar Van Neste C, Gansemans Y, De Coninck D, Van Hoofstat D, Van Criekinge W, Deforce D, Van Nieuwerburgh F (2015) Инструмент массового параллельного анализа данных судебно-медицинской экспертизы: реализация MyFLq в качестве автономного веб- и Illumina BaseSpace ® -приложение. Forensic Sci Int Genet 15: 2–7. https://doi.org/10.1016/j.fsigen.2014.10.006 CAS Статья PubMed Google Scholar Dove ES, Joly Y, Tassé A-M (2015) Международный руководящий комитет Public Population Project in Genomics and Society (P3G) и Комитет по этике и политике Международного консорциума генома рака (ICGC), Knoppers, B.М .: Геномные облачные вычисления: юридические и этические аспекты для рассмотрения. Eur J Human Genet 23 (10): 1271–1278. https://doi.org/10.1038/ejhg.2014.196 Артикул Google Scholar Docker Inc. (2019) Docker. https://www.docker.com/. По состоянию на 9 января 2019 г. da Veiga Leprevost F, Gruning BA, Alves Aflitos S, Rost HL, Uszkoreit J, Barsnes H, Vaudel M, Moreno P, Gatto L, Weber J, Bai M, Jimenez RC, Sachsenberg T, Pfeuffer J, Vera Alvarez R, Griss J, Несвижский AI, Perez-Riverol Y (2017) BioContainers: платформа с открытым исходным кодом и управляемая сообществом для стандартизации программного обеспечения.Биоинформатика 33 (16): 2580–2582. https://doi.org/10.1093/bioinformatics/btx192 CAS Статья PubMed PubMed Central Google Scholar Ким Б., Али Т., Лиджерон К., Афган Э, Крампис К. (2017) Биодоклеты: контейнеры виртуализации для одношагового выполнения конвейеров ngs. GigaScience 6 (8): 1–7. https://doi.org/10.1093/gigascience/gix048 CAS Статья PubMed PubMed Central Google Scholar Menegidio FB, Jabes DL, de Oliveira R Costa, Nunes LR (2018) Dugong: образ Docker, основанный на Ubuntu Linux, ориентированный на воспроизводимость и воспроизводимость для биоинформатического анализа. Биоинформатика 34 (3): 514–515. https://doi.org/10.1093/bioinformatics/btx554 CAS Статья PubMed Google Scholar Кулькарни Н., Алессандри Л., Панеро Р., Аригони М., Оливеро М., Ферреро Дж., Кордеро Ф., Беккути М., Калоджеро Р.А. (2018) Проект воспроизводимой биоинформатики: сообщество для трубопроводов воспроизводимого биоинформатического анализа.BMC Bioinform 19 (Приложение 10): 349. https://doi.org/10.1186/s12859-018-2296-x Артикул Google Scholar Розенблатт-Розен О., Стаббингтон М.Ю., Регев А., Тайхманн С.А. (2017) Атлас клеток человека: от видения к реальности. Nature 550 (7677): 451–453. https://doi.org/10.1038/550451a CAS Статья PubMed Google Scholar Peng RD (2011) Воспроизводимые исследования в области вычислительной науки.Наука 334 (6060): 1226–1227. https://doi.org/10.1126/science.1213847 CAS Статья PubMed PubMed Central Google Scholar Stodden V, Leisch F, Peng RD (2014) Проведение воспроизводимых исследований. CRC Press / Taylor & Francis Group, Бока-Ратон Google Scholar Scientific Data (2019) Рекомендуемые репозитории данных. https: // www.nature.com/sdata/policies/repositories/. По состоянию на 9 января 2019 г. Dryad (2019) Dryad Digital Repository. https://datadryad.org/. По состоянию на 9 января 2019 г. Dryad (2019) DryadLab. http://datadryad.org/pages/dryadlab/. По состоянию на 9 января 2019 г. figshare (2019) figshare - кредит на все ваши исследования. http://www.figshare.com/. По состоянию на 9 января 2019 г. Singh J (2011) Figshare. J Pharmacol Pharmacother 2 (2): 138–139.https://doi.org/10.4103/0976-500X.81919 Артикул PubMed PubMed Central Google Scholar Zenodo (2019) Zenodo - Research. Общий. https://zenodo.org/. По состоянию на 9 января 2019 г. Open Science Framework (2019) OSF Home. https://osf.io/. По состоянию на 9 января 2019 г. Center for Open Science (2019) Center for Open Science Website. https://cos.io/. По состоянию на 9 января 2019 г. Фостер Э.Д., Дирдорф А. (2017) Фреймворк открытой науки (osf). J Med Lib Assoc 105 (2): 203–206. https://doi.org/10.5195/JMLA.2017.88 Артикул Google Scholar Macmillan Publishers Limited (2019) Научные данные. https://www.nature.com/sdata/. По состоянию на 9 января 2019 г. Elsevier (2019) Краткие данные. https://www.journals.elsevier.com/data-in-brief/. По состоянию на 9 января 2019 г. MDPI (2019) Данные. http://www.mdpi.com/journal/data/. Доступ 9 января 2019 г. F1000Research (2019) F1000Research | Платформа для публикации в открытом доступе | За пределами исследовательского журнала. https://f1000research.com/. По состоянию на 9 января 2019 г. arXiv (2019) arXiv.org e-Print archive. https://arxiv.org/. По состоянию на 9 января 2019 г. bioRxiv (2019) bioRxiv.org - сервер препринтов для биологии. https://www.biorxiv.org/. По состоянию на 9 января 2019 г. ChemRxiv (2019) ChemRxiv: Сервер препринтов для химии. https://chemrxiv.org/. По состоянию на 9 января 2019 г. PeerJ (2019) PeerJ Preprints. https://peerj.com/preprints/. По состоянию на 9 января 2019 г. Bitbucket (2019) Bitbucket - решение Git для профессиональных команд. https://bitbucket.org/. По состоянию на 9 января 2019 г. GitLab (2019) GitLab. https://about.gitlab.com/. По состоянию на 9 января 2019 г. Assembla (2019) Assembla: Secure Git, безопасная разработка программного обеспечения в облаке.https://www.assembla.com/. По состоянию на 9 января 2019 г. Google (2019) Cloud Source Repositories. https://cloud.google.com/source-repositories/. По состоянию на 9 января 2019 г. Софронев Н.Дж., Власов Ю.А., Хайрес С.А., Фриман Дж., Свобода К. (2015) Нейронное кодирование в коре головного мозга во время движения, управляемого усами. eLife. https://doi.org/10.7554/eLife.12559 Артикул PubMed PubMed Central Google Scholar Ли Н., Дайе К., Свобода К., Друкманн С. (2016) Надежная динамика нейронов в премоторной коре во время двигательного планирования. Nature 532 (7600): 459–464. https://doi.org/10.1038/nature17643 CAS Статья PubMed PubMed Central Google Scholar Code Ocean (2019) Code Ocean - профессиональные инструменты для исследователей. https://codeocean.com/. По состоянию на 9 января 2019 г. Cornell Tech (2019) Code Ocean: Tackling Воспроизводимость и прозрачность в научных исследованиях.https://tech.cornell.edu/news/code-ocean-tackling-reproducibility-and-transparency-in- Scientific-Research. По состоянию на 9 января 2019 г. Perkel J (2019) TechBlog: C. Titus Brown: Predicting the paper of the future. http://blogs.nature.com/naturejobs/2017/06/01/techblog-c-titus-brown-predicting-the-paper-of-the-future/. По состоянию на 9 января 2019 г. Software Carpentry (2019) Software Carpentry - Обучение базовым лабораторным навыкам для исследовательских вычислений. https: // программно-столярные изделия.org /. По состоянию на 9 января 2019 г. Data Carpentry (2019) Data Carpentry - создание сообществ, обучающих универсальной информационной грамотности. http://www.datacarpentry.org/. По состоянию на 9 января 2019 г. Birney E, Hudson TJ, Green ED, Gunter C, Eddy S, Rogers J, Harris JR, Ehrlich SD, Apweiler R, Austin CP, Berglund L, Bobrow M, Bountra C, Brookes А.Дж., Камбон-Томсен А., Картер Н.П., Чисхолм Р.Л., Контрерас Дж.Л., Кук Р.М., Кросби В.Л., Дьюар К., Дурбин Р., Дайк С.О., Эккер Дж.Р., Эль-Эмам К., Феук Л., Габриэль С.Б., Галлахер Дж., Гелбарт В.М., Гранелл А., Гварнер Ф., Хаббард Т., Джексон С. А., Дженнингс Дж. Л., Джоли И., Джонс С. М., Кэй Дж., Кеннеди К. Л., Кнопперс Б. М., Кирпидес Н. С., Лоуренс В. В., Ло Дж., Маккей Д. Дж., Мартин-Ривера Л., Маккомби В. Р., McPherson JD, Miller L, Miller W, Moerman D, Mooser V, Morton CC, Ostell JM, Ouellette BF, Parkhill J, Raina PS, Rawlings C, Scherer SE, Scherer SW, Schofield PN, Sensen CW, Stodden VC, Sussman MR , Tanaka T, Thornton J, Tsunoda T., Valle D, Vuorio EI, Walker NM, Wallace S, Weinstock G, Whitman WB, Worley KC, Wu C, Wu J, Yu J (2009) Обмен данными перед публикацией.Nature 461 (7261): 168–170. https://doi.org/10.1038/461168a CAS Статья PubMed Google Scholar González-Medina M, Naveja JJ, Sánchez-Cruz N, Medina-Franco JL (2017) Откройте хемоинформатические ресурсы для изучения структуры, свойств и химического пространства молекул. RSC Adv 7 (85): 54153–54163. https://doi.org/10.1039/C7RA11831G Артикул Google Scholar Hasegawa K, Funatsu K (2014) Анализ данных хемогеномики с использованием бимодальных методов PLS и химической интерпретации для молекулярного дизайна. Мол Информ 33 (11–12): 749–756. https://doi.org/10.1002/minf.201400061 CAS Статья PubMed Google Scholar Mendez D, Gaulton A, Bento AP, Chambers J, De Veij M, Félix E, Magariños MP, Mosquera JF, Mutowo P, Nowotka M, Gordillo-Marañón M, Hunter F, Junco L, Mugumbate G , Родригес-Лопес М., Аткинсон Ф., Боск Н., Раду С.Дж., Сегура-Кабрера А., Херси А., Лич А.Р. (2019) ChEMBL: к прямому депонированию данных биотестов.Нуклеиновые кислоты Res 47 (D1): 930–940. https://doi.org/10.1093/nar/gky1075 CAS Статья Google Scholar Kim S, Chen J, Cheng T, Gindulyte A, He J, He S, Li Q, Shoemaker BA, Thiessen PA, Yu B, Zaslavsky L, Zhang J, Bolton EE (2019) Обновление PubChem за 2019 год : улучшен доступ к химическим данным. Нуклеиновые кислоты Res 47 (D1): 1102–1109. https://doi.org/10.1093/nar/gky1033 Артикул Google Scholar Gilson MK, Liu T, Baitaluk M, Nicola G, Hwang L, Chong J (2016) BindingDB в 2015 году: общедоступная база данных по медицинской химии, вычислительной химии и системной фармакологии. Nucleic Acids Res 44 (D1): 1045–53. https://doi.org/10.1093/nar/gkv1072 CAS Статья Google Scholar Гилсон М.К. (2019) BindingDB. https://www.bindingdb.org. По состоянию на 9 января 2019 г. Урсу О., Холмс Дж., Нокель Дж., Болога К.Г., Ян Дж.Дж., Матиас С.Л., Нельсон С.Дж., Опреа Т.И. (2017) DrugCentral: онлайн-справочник лекарств.Nucleic Acids Res 45 (D1): 932–939. https://doi.org/10.1093/nar/gkw993 CAS Статья Google Scholar Law V, Knox C, Djoumbou Y, Jewison T, Guo AC, Liu Y, Maciejewski A, Arndt D, Wilson M, Neveu V, Tang A, Gabriel G, Ly C, Adamjee S, Dame ZT , Han B, Zhou Y, Wishart DS (2014) DrugBank 4.0: проливаем новый свет на метаболизм лекарств. Nucleic Acids Res 42 (выпуск базы данных): 1091–1097. https://doi.org/10.1093/nar/gkt1068 CAS Статья Google Scholar Wishart DS, Feunang YD, Guo AC, Lo EJ, Marcu A, Grant JR, Sajed T, Johnson D, Li C, Sayeeda Z, Assempour N, Iynkkaran I, Liu Y, Maciejewski A, Gale N, Wilson A, Chin Л., Каммингс Р., Ле Д., Пон А., Нокс С., Уилсон М. (2018) DrugBank 5.0: крупное обновление базы данных DrugBank за 2018 год. Nucleic Acids Res 46 (D1): 1074–1082. https://doi.org/10.1093/nar/gkx1037 CAS Статья Google Scholar Mathias SL, Hines-Kay J, Yang JJ, Zahoransky-Kohalmi G, Bologa CG, Ursu O, Oprea T.I (2013) База данных CARLSBAD: конфедеративная база данных по химической биоактивности.База данных 2013: 044. https://doi.org/10.1093/database/bat044 CAS Статья Google Scholar Placzek S, Schomburg I, Chang A, Jeske L, Ulbrich M, Tillack J, Schomburg D (2017) Бренда в 2017 году: новые перспективы и новые инструменты в бренде. Нуклеиновые кислоты Res 45 (D1): 380–388. https://doi.org/10.1093/nar/gkw952 CAS Статья Google Scholar Sun J, Jeliazkova N, Chupakin V, Golib-Dzib JF, Engkvist O, Carlsson L, Wegner J, Ceulemans H, Georgiev I, Jeliazkov V, Kochev N, Ashby TJ, Chen H (2017) ExCAPE-DB: интегрированный крупномасштабный набор данных, облегчающий анализ больших данных в хемогеномике. J Cheminform 9:17. https://doi.org/10.1186/s13321-017-0203-5 CAS Статья PubMed PubMed Central Google Scholar Güner OF (2002) История и эволюция концепции фармакофоров в компьютерном дизайне лекарств.Curr Top Med Chem 2 (12): 1321–1332. https://doi.org/10.2174/15680260233 Артикул PubMed Google Scholar Patel Y, Gillet VJ, Bravi G, Leach AR (2002) Сравнение программ идентификации фармакофоров: катализатор, дискотека и вздох. J Comput Aided Mol Des 16 (8–9): 653–681. https://doi.org/10.1023/a:1021954728347 CAS Статья PubMed Google Scholar Sliwoski G, Kothiwale S, Meiler J, Lowe EW (2014) Вычислительные методы в открытии лекарств. Pharmacol Rev 66 (1): 334–395. https://doi.org/10.1124/pr.112.007336 CAS Статья PubMed PubMed Central Google Scholar Колосов Е., Лимон А. (2006) Инструменты медицинской химии: понимание данных hts. Eur J Med Chem 41 (2): 166–175. https://doi.org/10.1016/j.ejmech.2005.10.005 CAS Статья PubMed Google Scholar Doke SK, Dhawale SC (2015) Альтернативы тестированию на животных: обзор. Саудовская Фарм. Журнал 23 (3): 223–229. https://doi.org/10.1016/j.jsps.2013.11.002 Артикул Google Scholar Cronin MT, Jaworska JS, Walker JD, Comber MH, Watts CD, Worth AP (2003) Использование QSAR в международных структурах принятия решений для прогнозирования воздействия химических веществ на здоровье. Environ Health Perspect 111 (10): 1391–1401. https://doi.org/10.1289 / ehp.5760 CAS Статья PubMed PubMed Central Google Scholar Hofer T, Gerner I, Gundert-Remy U, Liebsch M, Schulte A, Spielmann H, Vogel R, Wettig K (2004) Тестирование на животных и альтернативные подходы к оценке риска для здоровья человека в рамках предлагаемого нового европейского стандарта. регулирование химикатов. Arch Toxicol 78 (10): 549–564. https://doi.org/10.1007/s00204-004-0577-9 CAS Статья PubMed Google Scholar Ashby J (1985) Основные структурные предупреждения о потенциальной канцерогенности или неканцерогенности. Environ Mutagen 7 (6): 919–921. https://doi.org/10.1002/em.2860070613 CAS Статья PubMed Google Scholar Эшби Дж., Теннант Р.В. (1991) Определенные взаимосвязи между химической структурой, канцерогенностью и мутагенностью для 301 химического вещества, протестированного NTP США. Резолюция о мутации 257 (3): 229–306. https://doi.org/10.1016 / 0165-1110 (91) -эCAS Статья PubMed Google Scholar Девиллерс Дж., Момбелли Э., Самсера Р. (2011) Структурные предупреждения для оценки канцерогенности пестицидов и биоцидов. SAR QSAR Environ Res 22 (1–2): 89–106. https://doi.org/10.1080/1062936X.2010.548349 CAS Статья PubMed Google Scholar Aptula AO, Patlewicz G, Roberts DW (2005) Сенсибилизация кожи: области применимости механистических реакций для взаимосвязей структура-активность.Chem Res Toxicol 18 (9): 1420–1426. https://doi.org/10.1021/tx050075m CAS Статья PubMed Google Scholar Робертс Д.В., Патлевич Г., Керн П.С., Герберик Ф., Кимбер И., Дирман Р.Дж., Райан К.А., Баскеттер Д.А., Аптула А.О. (2007) Классификация области механической применимости набора данных анализа локальных лимфатических узлов для сенсибилизации кожи. Chem Res Toxicol 20 (7): 1019–1030. https://doi.org/10.1021/tx700024w CAS Статья PubMed Google Scholar Blake JF (2005) Идентификация и оценка молекулярных свойств, связанных с доклинической оптимизацией и клинической судьбой. Med Chem 1 (6): 649–655. https://doi.org/10.2174/157340605774598081 CAS Статья PubMed Google Scholar Hann M, Hudson B, Lewell X, Lifely R, Miller L, Ramsden N (1999) Стратегическое объединение соединений для высокопроизводительного скрининга. J Chem Inform Comput Sci 39 (5): 897–902. https: // doi.org / 10.1021 / ci9 o CAS Статья Google Scholar Pearce BC, Sofia MJ, Good AC, Drexler DM, Stock DA (2006) Эмпирический процесс разработки высокопроизводительных фильтров-грохотов. J Chem Inform Model 46 (3): 1060–1068. https://doi.org/10.1021/ci050504m CAS Статья Google Scholar Алвес В., Муратов Е., Капуцци С., Полити Р., Лоу И, Брага Р., Захаров А.В., Седых А., Мокшина Е., Фараг С., Андраде СН, Кузьмин В.Е., Фурчеш Д., Тропша А. ( 2016) Оповещения о структурных предупреждениях.Грин Chem 18 (16): 4348–4360. https://doi.org/10.1039/C6GC01492E CAS Статья PubMed PubMed Central Google Scholar Labute P (2000) Широко применимый набор дескрипторов. Модель графа Дж. Моля 18 (4–5): 464–477. https://doi.org/10.1016/s1093-3263(00)00068-1 CAS Статья PubMed Google Scholar Nantasenamat C, Isarankura-Na-Ayudhya C, Naenna T, Prachayasittikul V (2009) Практический обзор количественной взаимосвязи структуры и активности.ИСКЛЮЧЕНИЕ J 8: 74–88. https://doi.org/10.17877/DE290R-690 Артикул Google Scholar Nantasenamat C, Isarankura-Na-Ayudhya C, Prachayasittikul V (2010) Достижения в области вычислительных методов для прогнозирования биологической активности соединений. Мнение экспертов по наркотикам Discov 5 (7): 633–654. https://doi.org/10.1517/17460441.2010.4 CAS Статья PubMed Google Scholar Randić M (2001) Новые дескрипторы формы для молекулярных графов. J Chem Inform Comput Sci 41 (3): 607–613. https://doi.org/10.1021/ci0001031 CAS Статья Google Scholar Senese CL, Duca J, Pan D, Hopfinger AJ, Tseng YJ (2004) 4D-отпечатки пальцев, универсальные дескрипторы QSAR и QSPR. J Chem Inform Comput Sci 44 (5): 1526–1539. https://doi.org/10.1021/ci049898s CAS Статья Google Scholar Shoombuatong W, Prathipati P, Owasirikul W, Worachartcheewan A, Simeon S, Anuwongcharoen N, Wikberg JES, Nantasenamat C (2017) На пути к возрождению интерпретируемых моделей QSAR. В: Рой К. (ред.) Достижения в задачах моделирования QSAR и достижения в области вычислительной химии и физики, том 24. Спрингер, Чам, стр. 3–55. https://doi.org/10.1007/978-3-319-56850-8_1 Google Scholar Hawkins DM, Basak SC, Shi X (2001) QSAR с небольшим количеством соединений и множеством функций.J Chem Inform Comput Sci 41 (3): 663–670. https://doi.org/10.1021/ci0001177 CAS Статья Google Scholar Rücker C, Rücker G, Meringer M (2007) y-рандомизация и ее варианты в QSPR / QSAR. J Chem Inform Model 47 (6): 2345–2357. https://doi.org/10.1021/ci700157b CAS Статья Google Scholar Weaver S, Gleeson MP (2008) Важность области применимости в моделировании QSAR.Модель графа Дж. Моля 26 (8): 1315–1326. https://doi.org/10.1016/j.jmgm.2008.01.002 CAS Статья PubMed Google Scholar Gleeson MP, Modi S, Bender A, Robinson RLM, Kirchmair J, Promkatkaew M, Hannongbua S, Glen RC (2012) Проблемы моделирования данных о токсичности in silico: обзор. Curr Pharm Des 18 (9): 1266–1291. https://doi.org/10.2174/138161212799436359 CAS Статья PubMed Google Scholar Коновалов Д.А., Ллевеллин Л.Е., Вандер Хейден Ю., Куманс Д. (2008) Надежная перекрестная проверка моделей линейной регрессии QSAR. J Chem Inform Model 48 (10): 2081–2094. https://doi.org/10.1021/ci800209k CAS Статья Google Scholar Эклунд М., Нориндер У., Бойер С., Карлссон Л. (2012) Применение конформного прогнозирования в QSAR. IFIP Adv Inform Commun Technol 382: 166–175. https://doi.org/10.1007/978-3-642-33412-2_17 Артикул Google Scholar Bosc N, Atkinson F, Felix E, Gaulton A, Hersey A, Leach AR (2019) Крупномасштабное сравнение методов QSAR и конформного прогнозирования и их применения в открытии лекарств. Журнал Cheminform 11 (1): 4. https://doi.org/10.1186/s13321-018-0325-4 Артикул PubMed PubMed Central Google Scholar Глисон М.П., Монтанари Д. (2012) Стратегии создания, проверки и применения моделей in silico ADMET для генерации лидов и оптимизации.Exp Opin Drug Metab Toxicol 8 (11): 1435–1446. https://doi.org/10.1517/17425255.2012.711317 CAS Статья Google Scholar Топлисс Дж. Г., Эдвардс Р. П. (1979) Факторы вероятности в исследованиях количественных взаимосвязей структура – активность. J Med Chem 22 (10): 1238–1244. https://doi.org/10.1021/jm00196a017 CAS Статья PubMed Google Scholar Ломбардо Ф., Гиффорд Э., Шалаева М.Ю. (2003) Прогноз In silico ADME: данные, модели, факты и мифы. Mini Rev Med Chem 3 (8): 861–875. https://doi.org/10.2174/1389557033487629 CAS Статья PubMed Google Scholar Wood DJ, Buttar D, Cumming JG, Davis AM, Norinder U, Rodgers SL (2011) Автоматизированный QSAR с иерархией глобальных и локальных моделей. Мол информ 30 (11–12): 960–972. https://doi.org/10.1002/minf.201100107 CAS Статья PubMed Google Scholar Tetko IV, Bruneau P, Mewes H-W, Rohrer DC, Poda GI (2006) Можем ли мы оценить точность прогнозов adme-tox? Диск с наркотиками сегодня 11 (15–16): 700–707. https://doi.org/10.1016/j.drudis.2006.06.013 CAS Статья Google Scholar 37-е совместное совещание Комитета по химическим веществам (2004 г.) Принципы ОЭСР для валидации (количественных) моделей взаимосвязи между структурой и деятельностью в целях регулирования.https://www.oecd.org/chemicalsafety/risk-assessment/37849783.pdf. По состоянию на 9 января 2019 г. Judson PN, Barber C, Canipa SJ, Poignant G, Williams R (2015) Создание надлежащей практики компьютерного моделирования (gcmp) для прогнозирования химической токсичности. Мол информ 34 (5): 276–283. https://doi.org/10.1002/minf.201400137 CAS Статья PubMed Google Scholar Tropsha A (2010) Лучшие практики для разработки, проверки и эксплуатации модели QSAR.Мол Информ 29 (6–7): 476–488. https://doi.org/10.1002/minf.201000061 CAS Статья PubMed Google Scholar Patel M, Chilton ML, Sartini A, Gibson L, Barber C, Covey-Crump L, Przybylak KR, Cronin MTD, Madden JC (2018) Оценка и воспроизводимость количественных моделей взаимосвязи структура – активность неспециалистом . J Chem Inform Model 58 (3): 673–682. https://doi.org/10.1021/acs.jcim.7b00523 CAS Статья Google Scholar Арора П.К., Патил В.М., Гупта С.П. (2010) Исследование QSAR по некоторым сериям агентов против вируса гепатита B (HBV). Биоинформация 4 (9): 417–420. https://doi.org/10.6026/97320630004417 Артикул Google Scholar Kurdekar V, Jadhav HR (2015) Новый скрипт Python для анализа данных с открытым исходным кодом для исследования QSAR и его проверки. Med Chem Res 24 (4): 1617–1625. https://doi.org/10.1007/s00044-014-1240-5 CAS Статья Google Scholar Научно-исследовательское сотрудничество в области структурной биоинформатики (2019) The Protein Data Bank (PDB). http://www.rcsb.org/pdb/. По состоянию на 9 января 2019 г. Fiser A, Sali A (2003) Modeller: создание и уточнение моделей структуры белков на основе гомологии. Методы Enzymol 374: 461–491. https://doi.org/10.1016/S0076-6879(03)74020-8 CAS Статья PubMed Google Scholar Юинг Т.Дж., Макино С., Скиллман АГ, Кунц И.Д. (2001) Док 4.0: стратегии поиска для автоматической молекулярной стыковки гибких баз данных молекул. J Comput Aided Mol Des 15 (5): 411–428. https://doi.org/10.1023/a:1011115820450 CAS Статья PubMed Google Scholar Goodsell DS, Olson AJ (1990) Автоматическая стыковка субстратов с белками путем имитации отжига. Белки 8 (3): 195–202. https://doi.org/10.1002/prot.340080302 CAS Статья PubMed Google Scholar Warren GL, Andrews CW, Capelli AM, Clarke B, LaLonde J, Lambert MH, Lindvall M, Nevins N, Semus SF, Senger S, Tedesco G, Wall ID, Woolven JM, Peishoff CE, Head MS (2006) Критический оценка стыковочных программ и функций подсчета очков. J Med Chem 49 (20): 5912-5931. https://doi.org/10.1021/jm050362n CAS Статья PubMed Google Scholar Kubinyi H (1997) QSAR и 3D QSAR в дизайне лекарств. Часть 2: приложения и проблемы.Drug Discov Today 2: 538–546. https://doi.org/10.1016/S1359-6446(97)01084-2 CAS Статья Google Scholar Kubinyi H (1997) QSAR и 3D QSAR в разработке лекарств. Часть 1: методология. Drug Discov Today 2 (11): 457–467. https://doi.org/10.1016/S1359-6446(97)01079-9 CAS Статья Google Scholar Cramer RD, Wendt B (2007) Расширяя границы 3D-QSAR.J Comput Aided Mol Des 21 (1–3): 23–32. https://doi.org/10.1007/s10822-006-9100-0 CAS Статья PubMed Google Scholar Leach AR (2001) Молекулярное моделирование: принципы и приложения, 2-е изд. Pearson Education, Харлоу Google Scholar Menikarachchi LC, Gascón JA (2010) Подходы QM / MM в исследованиях в области медицинской химии. Curr Top Med Chem 10 (1): 46–54.https://doi.org/10.2174/1568026107 CAS Статья PubMed Google Scholar Mulholland AJ (2007) Химическая точность расчетов QM / MM для реакций, катализируемых ферментами. Chem Cent J 1:19. https://doi.org/10.1186/1752-153X-1-19 CAS Статья PubMed PubMed Central Google Scholar Senn HM, Thiel W (2007) QM / MM исследования ферментов.Curr Opin Chem Biol 11 (2): 182–187. https://doi.org/10.1016/j.cbpa.2007.01.684 CAS Статья PubMed Google Scholar Senn HM, Thiel W (2009) Методы QM / MM для биомолекулярных систем. Angewandte Chemie 48 (7): 1198–1229. https://doi.org/10.1002/anie.200802019 CAS Статья PubMed Google Scholar Walker RC, Crowley MF, Case DA (2008) Реализация быстрого и точного метода определения потенциала QM / MM в янтаре.J. Comput Chem 29 (7): 1019–1031. https://doi.org/10.1002/jcc.20857 CAS Статья PubMed Google Scholar Butcher EC, Berg EL, Kunkel EJ (2004) Системная биология в открытии лекарств. Nat Biotechnol 22 (10): 1253–1259. https://doi.org/10.1038/nbt1017 CAS Статья PubMed Google Scholar Pujol A, Mosca R, Farres J, Aloy P (2010) Раскрытие роли сетевой и системной биологии в открытии лекарств.Тенденции Pharmacol Sci 31 (3): 115–123. https://doi.org/10.1016/j.tips.2009.11.006 CAS Статья PubMed Google Scholar Кейзер MJ, Сетола V, Ирвин JJ, Laggner C, Abbas AI, Hufeisen SJ, Jensen NH, Kuijer MB, Matos RC, Tran TB, Whaley R, Glennon RA, Hert J, Thomas KL, Edwards DD , Shoichet BK, Roth BL (2009) Прогнозирование новых молекулярных мишеней для известных лекарств. Nature 462 (7270): 175–181. https://doi.org/10.1038/nature08506 CAS Статья PubMed PubMed Central Google Scholar Ye H, Wei J, Tang K, Feuers R, Hong H (2016) Репозиционирование лекарств с помощью сетевой фармакологии. Curr Top Med Chem 16 (30): 3646–3656. https://doi.org/10.2174/1568026616666160530181328 CAS Статья PubMed Google Scholar Кейзер М.Дж., Рот Б.Л., Армбрустер Б.Н., Эрнсбергер П., Ирвин Дж. Дж., Шойхет Б.К. (2007) Связь фармакологии белков с помощью химии лигандов. Nat Biotechnol 25 (2): 197–206. https://doi.org/10.1038/nbt1284 CAS Статья PubMed Google Scholar Wu W, Zhang R, Salahub DR (2009) Нельфинавир: волшебная пуля для уничтожения раковых клеток? Cancer Biol Ther 8 (3): 233–235. https://doi.org/10.4161/cbt.8.3.7789 CAS Статья PubMed Google Scholar Dakshanamurthy S, Issa NT, Assefnia S, Seshasayee A, Peters OJ, Madhavan S, Uren A, Brown ML, Byers SW (2012) Прогнозирование новых показаний для одобренных препаратов с использованием протеохимометрического метода. J Med Chem 55 (15): 6832–6848.https://doi.org/10.1021/jm300576q CAS Статья PubMed PubMed Central Google Scholar Schaduangrat N, Anuwongcharoen N, Phanus-umporn C, Sriwanichpoom N, Wikberg JES, Nantasenamat C (2019) Глава 10. Протеохимометрическое моделирование репозиционирования лекарств. В: Рой К. (ред.) В дизайне лекарств Silico. Academic Press, Лондон, стр. 281–302. https://doi.org/10.1016/B978-0-12-816125-8.00010-9 Google Scholar Waltemath D, Wolkenhauer O (2016) Как стандарты моделирования, программное обеспечение и инициативы поддерживают воспроизводимость в системной биологии и системной медицине. IEEE Trans Biomed Eng 63 (10): 1999–2006. https://doi.org/10.1109/TBME.2016.2555481 Артикул PubMed Google Scholar Медли Дж. К., Голдберг А. П., Карр Дж. Р. (2016) Рекомендации по воспроизводимому построению и моделированию моделей системной биологии. IEEE Trans Biomed Eng 63 (10): 2015–2020.https://doi.org/10.1109/TBME.2016.25 Артикул PubMed PubMed Central Google Scholar Waltemath D, Henkel R, Winter F, Wolkenhauer O (2013) Воспроизводимость результатов на основе моделей в системной биологии. В: Prokop A, Csukás B (eds) Syst Biol. Springer, Dordrecht, стр. 301–320. https://doi.org/10.1007/978-94-007-6803-1_10 Google Scholar Le Novère N, Bornstein B, Broicher A, Courtot M, Donizelli M, Dharuri H, Li L, Sauro H, Schilstra M, Shapiro B, Snoep JL, Hucka M (2006) База данных биомоделей: бесплатная централизованная база данных опубликованы количественные кинетические модели биохимических и клеточных систем. Nucleic Acids Res 34: 689–691. https://doi.org/10.1093/nar/gkj092 CAS Статья Google Scholar Kirouac DC, Cicali B, Schmidt S (2019) Воспроизводимость количественных моделей фармакологии систем: текущие проблемы и будущие возможности.CPT Pharmacometrics Syst Pharmacol 8 (4): 205–210. https://doi.org/10.1002/psp4.12390 CAS Статья PubMed PubMed Central Google Scholar Watanabe L, Barhak J, Myers C (2019) К воспроизводимым моделям болезней с использованием языка разметки системной биологии. Моделирование 95 (10): 895–930. https://doi.org/10.1177/00375497187 Артикул Google Scholar Hucka M, Finney A, Sauro HM, Bolouri H, Doyle JC, Kitano H, Arkin AP, Bornstein BJ, Bray D, Cornish-Bowden A, Cuellar AA, Dronov S, Gilles ED, Ginkel M, Gor V, Goryanin II , Hedley WJ, Hodgman TC, Hofmeyr JH, Hunter PJ, Juty NS, Kasberger JL, Kremling A, Kummer U, Le Novere N, Loew LM, Lucio D, Mendes P, Minch E, Mjolsness ED, Nakayama Y, Nelson MR, Nielsen PF, Sakurada T, Schaff JC, Shapiro BE, Shimizu TS, Spence HD, Stelling J, Takahashi K, Tomita M, Wagner J, Wang J (2003) Язык разметки системной биологии (SBML): среда для представления и обмена моделей биохимических сетей.Биоинформатика 19 (4): 524–531. https://doi.org/10.1093/bioinformatics/btg015 CAS Статья PubMed Google Scholar Сват MJ, Moodie S, Wimalaratne SM, Kristensen NR, Lavielle M, Mari A, Magni P, Smith MK, Bizzotto R, Pasotti L, Mezzalana E, Comets E, Sarr C, Terranova N, Blaudez E , Chan P, Chard J, Chatel K, Chenel M, Edwards D, Franklin C, Giorgino T, Glont M, Girard P, Grenon P, Harling K, Hooker AC, Kaye R, Keizer R, Kloft C, Kok JN, Kokash N, Laibe C, Laveille C, Lestini G, Mentre F, Munafo A, Nordgren R, Nyberg HB, Parra-Guillen ZP, Plan E, Ribba B, Smith G, Troconiz IF, Yvon F, Milligan PA, Harnisch L, Karlsson M, Hermjakob H, Le Novere N (2015) Язык разметки фармакометрии (PharmML): открытие новых перспектив для обмена моделями при разработке лекарств.CPT Pharmacometrics Syst Pharmacol 4 (6): 316–319. https://doi.org/10.1002/psp4.57 CAS Статья PubMed PubMed Central Google Scholar Barhak J (2019) MIST: Инструмент микромоделирования для поддержки моделирования заболеваний. https://github.com/scipy-conference/scipy2013_talks/tree/master/talks/jacob_barhak. По состоянию на 1 ноября 2019 г. Hedley WJ, Nelson MR, Bullivant DP, Nielsen PF (2001) Краткое введение в CellML.Philos Trans R Soc A 359 (1783): 1073–1089. https://doi.org/10.1098/rsta.2001.0817 Артикул Google Scholar Medley JK, Choi K, Konig M, Smith L, Gu S, Hellerstein J, Sealfon SC, Sauro HM (2018) Блокноты с теллуром - среда для воспроизводимого динамического моделирования в системной биологии. PLoS Comput Biol 14 (6): 1006220. https://doi.org/10.1371/journal.pcbi.1006220 CAS Статья Google Scholar Choi K, Medley JK, Konig M, Stocking K, Smith L, Gu S, Sauro HM (2018) Tellurium: расширяемая среда моделирования на основе Python для систем и синтетической биологии. Биосистемы 171: 74–79. https://doi.org/10.1016/j.biosystems.2018.07.006 CAS Статья PubMed PubMed Central Google Scholar Колпаков Ф., Акбердин И., Кашапов Т., Киселев Л., Колмыков С., Кондрахин Ю., Кутумова Е., Мандрик Н., Пинтус С., Рябова А., Шарипов Р., Евшин И., Кел А. (2019) BioUML: an интегрированная среда для системной биологии и совместного анализа биомедицинских данных.Нуклеиновые кислоты Res 47 (W1): 225–233. https://doi.org/10.1093/nar/gkz440 Артикул Google Scholar Drawert B, Trogdon M, Toor S, Petzold L, Hellander A (2016) MOLNs: облачная платформа для интерактивных, воспроизводимых и масштабируемых пространственных стохастических вычислительных экспериментов в системной биологии с использованием PyURDME. SIAM J Sci Comput 38 (3): 179–202. https://doi.org/10.1137/15M1014784 Артикул Google Scholar Schadt EE, Linderman MD, Sorenson J, Lee L, Nolan GP (2010) Вычислительные решения для крупномасштабного управления и анализа данных. Нат Рев Генет 11 (9): 647–657. https://doi.org/10.1038/nrg2857 CAS Статья PubMed PubMed Central Google Scholar Noble WS (2009) Краткое руководство по организации проектов в области вычислительной биологии. PLoS Comput Biol 5 (7): 1000424. https://doi.org/10.1371/journal.pcbi.1000424 CAS Статья Google Scholar Hassan M, Brown RD, VarmaO’brien S, Rogers D (2006) Химинформатический анализ и обучение в среде конвейерной обработки данных. Mol Divers 10 (3): 283–299. https://doi.org/10.1007/s11030-006-9041-5 CAS Статья PubMed Google Scholar Бертольд М.Р., Себрон Н., Дилл Ф., Габриэль Т.Р., Кёттер Т., Мейнл Т., Ол П., Тиль К., Висведель Б. (2009) KNIME - поиск информации Констанца. Информационный бюллетень ACM SIGKDD Explor 11 (1): 26.https://doi.org/10.1145/1656274.1656280 Артикул Google Scholar Cox R, Green DVS, Luscombe CN, Malcolm N, Pickett SD (2013) Инструментальные средства QSAR: автоматизация моделирования QSAR для управления составным дизайном. J Comput Aided Mol Des 27 (4): 321–336. https://doi.org/10.1007/s10822-013-9648-4 CAS Статья PubMed PubMed Central Google Scholar Steinmetz FP, Mellor CL, Meinl T, Cronin MTD (2015) Скрининг химических веществ на предмет опосредованных рецепторами токсикологических и фармакологических конечных точек: использование общедоступных данных для создания инструментов скрининга в рамках рабочего процесса KNIME. Мол Информ 34 (2–3): 171–178. https://doi.org/10.1002/minf.201400188 CAS Статья PubMed Google Scholar Никола Г., Бертольд М.Р., Хедрик М.П., Гилсон М.К. (2015) Соединение белков с лекарственными соединениями: рабочие процессы открытия лекарств с открытым исходным кодом с BindingDB и KNIME.База данных. https://doi.org/10.1093/database/bav087 Артикул PubMed PubMed Central Google Scholar Mazanetz MP, Marmon RJ, Reisser CBT, Morao I (2012) Приложения для обнаружения лекарств для Knime: платформа интеллектуального анализа данных с открытым исходным кодом. Curr Top Med Chem 12 (18): 1965–1979. https://doi.org/10.2174/156802612804 1 CAS Статья PubMed Google Scholar Kuhn T, Willighagen EL, Zielesny A, Steinbeck C (2010) Cdk-taverna: открытая рабочая среда для хеминформатики. BMC Bioinform 11: 159. https://doi.org/10.1186/1471-2105-11-159 CAS Статья Google Scholar Steinbeck C, Han Y, Kuhn S, Horlacher O, Luttmann E, Willighagen E (2003) The Chemistry Development Kit (CDK): библиотека Java для химио- и биоинформатики с открытым исходным кодом. J Chem Inform Comput Sci 43 (2): 493–500.https://doi.org/10.1021/ci025584y CAS Статья Google Scholar Виллигхаген Е.Л., Мэйфилд Дж. В., Альварссон Дж., Берг А., Карлссон Л., Джелязкова Н., Кун С., Плюскал Т., Рохас-Черто М., Спют О, Торранс Дж., Эвело СТ, Гуха Р., Стейнбек К. ( 2017) The Chemistry Development Kit (CDK) v2.0: типирование атомов, описание, молекулярные формулы и поиск субструктур. J Cheminform 9:33. https://doi.org/10.1186/s13321-017-0220-4 CAS Статья PubMed PubMed Central Google Scholar Лукас X, Грюнинг Б.А., Гюнтер С. (2014) ChemicalToolBoX и его приложение для изучения лекарственного средства и покупаемого пространства. Журнал Cheminform 6 (Дополнение 1): 51. https://doi.org/10.1186/1758-2946-6-S1-P51 Артикул Google Scholar Di Tommaso P, Chatzou M, Floden EW, Barja PP, Palumbo E, Notredame C (2017) Nextflow обеспечивает воспроизводимые вычислительные рабочие процессы. Nat Biotechnol 35 (4): 316–319. https://doi.org/10.1038/nbt.3820 CAS Статья PubMed Google Scholar Köster J, Rahmann S (2012) Snakemake - масштабируемый механизм рабочего процесса биоинформатики. Биоинформатика 28 (19): 2520–2522. https://doi.org/10.1093/bioinformatics/bts480 CAS Статья PubMed Google Scholar Goodstadt L (2010) Ruffus: легкая библиотека Python для вычислительных конвейеров.Биоинформатика 26 (21): 2778–2779. https://doi.org/10.1093/bioinformatics/btq524 CAS Статья PubMed Google Scholar Sadedin SP, Pope B, Oshlack A (2012) Bpipe: инструмент для запуска и управления конвейерами биоинформатики. Биоинформатика 28 (11): 1525–1526. https://doi.org/10.1093/bioinformatics/bts167 CAS Статья PubMed Google Scholar Brandt J, Reisig W, Leser ULF (2017) Вычислительная семантика клинописи языка функционального научного рабочего процесса. Программа J Funct. https://doi.org/10.1017/S0956796817000119 Артикул Google Scholar Bernhardsson E, Freider E, Rouhani A (2012) Репозиторий Луиджи на GitHub. https://github.com/spotify/luigi Wilson G, Aruliah DA, Brown CT, Chue Hong NP, Davis M, Guy RT, Haddock SH, Huff KD, Mitchell IM, Plumbley MD, Waugh B, White EP, Wilson P (2014) Лучшие практики для научных вычислений.PLoS Biol 12 (1): 1001745. https://doi.org/10.1371/journal.pbio.1001745 Артикул Google Scholar Тащук М., Уилсон Г. (2017) Десять простых правил повышения надежности исследовательского программного обеспечения. PLoS Comput Biol 13 (4): 1005412. https://doi.org/10.1371/journal.pcbi.1005412 CAS Статья Google Scholar Nowotka MM, Gaulton A, Mendez D, Bento AP, Hersey A, Leach A (2017) Использование веб-сервисов ChEMBL для создания приложений и рабочих процессов обработки данных, имеющих отношение к открытию лекарств.Exp Opin Drug Discov 12 (8): 757–767. https://doi.org/10.1080/17460441.2017.1339032 Артикул Google Scholar Alvarsson J, Lampa S, Schaal W., Andersson C, Wikberg JES, Spjuth O (2016). Крупномасштабное прогнозное моделирование на основе лигандов с использованием машин опорных векторов. J Cheminform 8:39. https://doi.org/10.1186/s13321-016-0151-5 Артикул PubMed PubMed Central Google Scholar Lampa S, Alvarsson J, Spjuth O (2016) На пути к гибкому крупномасштабному прогнозному моделированию при открытии лекарств с использованием принципов проектирования потокового программирования. J Cheminform 8:67. https://doi.org/10.1186/s13321-016-0179-6 Артикул PubMed PubMed Central Google Scholar Yoo AB, Jette MA, Grondona M (2003) SLURM: простая утилита Linux для управления ресурсами. В: Feitelson D, Rudolph L, Schwiegelshohn U (eds) Стратегии планирования заданий для параллельной обработки.Конспект лекций по информатике, том 2862. Springer, Berlin, pp. 44–60 Google Scholar Amstutz P, Crusoe MR, Tijanić N, Chapman B, Chilton J, Heuer M, Kartashov A, Leehr D, Ménager H, Nedeljkovich M, Scales M, Soiland-Reyes S, Stojanovic L (2019) Общие Язык рабочего процесса, v1.0. https://doi.org/10.6084/m9.figshare.3115156.v2. По состоянию на 9 января 2019 г. Чепмен Б., Джентри Дж., Лин М., Маги П., О'Коннор Б., Прабхакаран А., Ван дер Аувера Г. (2019) OpenWDL.http://www.openwdl.org/. По состоянию на 9 января 2019 г. Davie P (2010) Облачные вычисления: изменение правил игры в открытии лекарств? Иннов Фарм Технол 33: 34–36 Google Scholar Дадли Дж. Т., Батт А. Дж. (2010) In silico Исследования в эпоху облачных вычислений. Nat Biotechnol 28 (11): 1181–1185. https://doi.org/10.1038/nbt1110-1181 CAS Статья PubMed PubMed Central Google Scholar Гарг В., Арора С., Гупта С. (2011) Подходы облачных вычислений для ускорения цепочки создания добавленной стоимости при открытии лекарств. Comb Chem High Throughput Screen 14 (10): 861–871. https://doi.org/10.2174/138620711797537085 CAS Статья PubMed Google Scholar Могхадам Б.Т., Альварссон Дж., Холм М., Эклунд М., Карлссон Л., Спют О. (2015) Масштабируемое прогнозное моделирование при разработке лекарств с помощью облачных вычислений. J Chem Inform Model 55 (1): 19-25.https://doi.org/10.1021/ci500580y CAS Статья Google Scholar Hurley DG, Budden DM, Crampin EJ (2015) Виртуальные справочные среды: простой способ сделать исследование воспроизводимым. Краткий биоинформ 16 (5): 901–903. https://doi.org/10.1093/bib/bbu043 Артикул PubMed Google Scholar Piccolo SR, Frampton MB (2016) Инструменты и методы воспроизводимости вычислений.GigaScience 5 (1): 30. https://doi.org/10.1186/s13742-016-0135-4 Артикул PubMed PubMed Central Google Scholar Jaghoori MM, Bleijlevens B, Olabarriaga SD (2016) 1001 способ запуска AutoDock Vina для виртуального просмотра. J Comput Aided Mol Des 30 (3): 237–249. https://doi.org/10.1007/s10822-016-9900-9 CAS Статья PubMed PubMed Central Google Scholar McGuire R, Verhoeven S, Vass M, Vriend G, de Esch IJ, Lusher SJ, Leurs R, Ridder L, Kooistra AJ, Ritschel T, de Graaf C (2017) 3D-e-Chem-VM: инфраструктура исследования структурной хеминформатики в свободно доступной виртуальной машине. J Chem Inf Model 57 (2): 115–121. https://doi.org/10.1021/acs.jcim.6b00686 CAS Статья PubMed PubMed Central Google Scholar Альвим-Гастон М., Грезе Т., Махуи А., Палковиц А.Д., Пинейро-Нуньес М., Уотсон И. (2014) Открытое открытие инновационных лекарств (OIDD): потенциальный путь к новому терапевтическому химическому пространству.Curr Top Med Chem 14 (3): 294–303. https://doi.org/10.2174/1568026613666131127125858 CAS Статья PubMed Google Scholar Очоа Р., Дэвис М., Пападатос Дж., Аткинсон Ф., Оверингтон Дж. П. (2014) myChEMBL: реализация на виртуальной машине инструментов открытых данных и хеминформатики. Биоинформатика 30 (2): 298–300. https://doi.org/10.1093/bioinformatics/btt666 CAS Статья PubMed Google Scholar Эллингсон С.Р., Бодри Дж. (2011) Виртуальная молекулярная стыковка с высокой пропускной способностью: реализация AutoDock4 в Hadoop в частном облаке. В: Материалы второго международного семинара по новым вычислительным методам для наук о жизни - ECMLS’11. ACM Press, Нью-Йорк, стр. 33–38. https://doi.org/10.1145/1996023.1996028 Capuccini M, Ahmed L, Schaal W, Laure E, Spjuth O (2017) Крупномасштабный виртуальный просмотр ресурсов общедоступного облака с использованием apache Spark. J Cheminform 9:15.https://doi.org/10.1186/s13321-017-0204-4 Артикул PubMed PubMed Central Google Scholar Георгиева П., Лапинс М., Спют О, Викберг Дж. (2019) Фармацевтическая биоинформатика: бесплатный интернет-курс для иностранных и шведских студентов, предлагаемый Упсальским университетом. http://www.pharmbio.org/. По состоянию на 1 ноября 2019 г. Dahlö M, Haziza F, Kallio A, Korpelainen E, Bongcam-Rudloff E, Spjuth O (2015) BioImg.org: каталог образов виртуальных машин для наук о жизни. Bioinform Biol Insights 9: 125–128. https://doi.org/10.4137/BBI.S28636 Артикул PubMed PubMed Central Google Scholar Cito J, Gall HC (2016) Использование докер-контейнеров для улучшения воспроизводимости в исследованиях программной инженерии. В: Материалы 38-й международной конференции по разработке программного обеспечения ICSE ’16. ACM Press, Нью-Йорк, стр. 906–907 Silver A (2017) Программное обеспечение упрощено. Nature 546 (7656): 173–174. https://doi.org/10.1038/546173a CAS Статья PubMed Google Scholar Курцер Г.М., Сочат В., Бауэр М.В. (2017) Singularity: научные контейнеры для мобильности вычислений. PLoS ONE 12 (5): 0177459. https://doi.org/10.1371/journal.pone.0177459 CAS Статья Google Scholar Gomes J, Campos I, Bagnaschi E, David M, Alves L, Martins J, Pina J, Lopez-Garcia A, Orviz P (2017) Включение контейнеров linux без root в многопользовательских средах: инструмент udocker . Comput Phys Commun 232: 84–97. https://doi.org/10.1016/j.cpc.2018.05.021 CAS Статья Google Scholar Warr WA (2012) Системы научного документооборота: пилотный трубопровод и известная информация. J Comput Aided Mol Des 26 (7): 801–804.https://doi.org/10.1007/s10822-012-9577-7 CAS Статья PubMed PubMed Central Google Scholar Сухартанто Х., Пасарибу А.П., Сиддик М.Ф., Фадхила М.И., Хилман М.Х., Януар А. (2017) Предварительное исследование перехода с виртуальной машины на док-контейнер для обнаружения лекарств insilico в облаке. Int J Technol 8 (4): 611. https://doi.org/10.14716/ijtech.v8i4.9478 Артикул Google Scholar Фонг Дж. (2019) Как GlaxoSmithKline ускоряет науку с помощью Docker Enterprise Edition. https://blog.docker.com/2017/10/how-gsk-is-accelerating-science-with-dockeree/. По состоянию на 9 января 2019 г. Altae-Tran H, Ramsundar B, Pappu AS, Pande V (2017) Открытие лекарств с низким объемом данных с однократным обучением. ACS Cent Sci 3 (4): 283–293. https://doi.org/10.1021/acscentsci.6b00367 CAS Статья PubMed PubMed Central Google Scholar OpenRiskNet (2019) Открытая электронная инфраструктура для поддержки обмена данными, интеграции знаний, а также анализа и моделирования in silico для прогнозирования токсикологии и оценки рисков. http://www.openrisknet.org/. По состоянию на 9 января 2019 г. Belmann P, Dröge J, Bremges A, McHardy AC, Sczyrba A, Barton MD (2015) Биобоксы: стандартизированные контейнеры для сменного программного обеспечения биоинформатики. GigaScience 4:47. https://doi.org/10.1186/s13742-015-0087-0 Артикул PubMed PubMed Central Google Scholar Li W, Kanso A (2015) Сравнение контейнеров и виртуальных машин для достижения высокой доступности. В: Международная конференция IEEE по облачной инженерии, 2015 г. IEEE, Нью-Джерси, стр. 353–358. https://doi.org/10.1109/IC2E.2015.79 Spjuth O, Willighagen EL, Guha R, Eklund M, Wikberg JE (2010) На пути к интероперабельному и воспроизводимому анализу QSAR: обмен наборами данных. Журнал Cheminform 2 (1): 5. https://doi.org/10.1186/1758-2946-2-5 CAS Статья PubMed PubMed Central Google Scholar Ruusmann V, Sild S, Maran U (2014) Банк данных QSAR - подход к цифровой организации и архивированию информации о модели QSAR. J Cheminform 6:25. https://doi.org/10.1186/1758-2946-6-25 CAS Статья PubMed PubMed Central Google Scholar Руусманн В., Сильд С., Маран Ю. (2015) Репозиторий банка данных QSAR: открытые и связанные качественные и количественные модели взаимосвязи структуры и деятельности. Журнал Cheminform 7 (1): 32.https://doi.org/10.1186/s13321-015-0082-6 CAS Статья PubMed PubMed Central Google Scholar Объединенный исследовательский центр, Служба науки и знаний Европейской комиссии (2019 г.) (Q) База данных формата отчетности по модели SAR. https://qsardb.jrc.ec.europa.eu/qmrf/. По состоянию на 1 ноября 2019 г. Hastings J, Jeliazkova N, Owen G, Tsiliki G, Munteanu CR, Steinbeck C, Willighagen E (2015) eNanoMapper: использование онтологий для интеграции данных для оценки риска наноматериалов.J Biomed Demant 6 (1): 10 Статья Google Scholar Guazzelli A, Zeller M, Lin W-C, Williams G et al (2009) PMML: открытый стандарт для совместного использования моделей. R J 1 (1): 60–65 Статья Google Scholar Center for Computational Science Research, Inc. (2019) Data Mining Group. http://dmg.org/. По состоянию на 1 ноября 2019 г. Fillbrunn A (2019) Интеграция PMML в KNIME.https://www.knime.com/blog/pmml-integration-in-knime/. По состоянию на 1 ноября 2019 г. Участники проекта ONNX (2019) Открытый формат обмена нейронной сетью: открытая экосистема для взаимозаменяемых моделей искусственного интеллекта. https://onnx.ai/. По состоянию на 1 ноября 2019 г. Stålring JC, Carlsson LA, Almeida P, Boyer S (2011) AZOrange - высокопроизводительное машинное обучение с открытым исходным кодом для моделирования QSAR в среде графического программирования. J Cheminform 3:28. https://doi.org/10.1186/1758-2946-3-28 CAS Статья PubMed PubMed Central Google Scholar Dixon SL, Duan J, Smith E, Von Bargen CD, Sherman W., Repasky MP (2016) AutoQSAR: автоматизированный инструмент машинного обучения для оптимального количественного моделирования взаимосвязи структуры и деятельности. Fut Med Chem 8 (15): 1825–1839. https://doi.org/10.4155/fmc-2016-0093 CAS Статья Google Scholar Nantasenamat C, Worachartcheewan A, Jamsak S, Preeyanon L, Shoombuatong W., Simeon S, Mandi P, Isarankura-Na-Ayudhya C, Prachayasittikul V (2015) AutoWeka: к автоматизированному программному обеспечению интеллектуального анализа данных для QSAR QSPR исследования.Методы Mol Biol 1260: 119–147. https://doi.org/10.1007/978-1-4939-2239-0_8 CAS Статья PubMed Google Scholar Холл М., Фрэнк Э., Холмс Дж., Пфарингер Б., Ройтеманн П., Виттен И. Х. (2009) Программное обеспечение для интеллектуального анализа данных weka. Информационный бюллетень ACM SIGKDD Explor 11 (1): 10. https://doi.org/10.1145/1656274.1656278 Артикул Google Scholar Kausar S, Falcao AO (2018) Автоматизированная структура для построения модели QSAR.Журнал Cheminform 10 (1): 1. https://doi.org/10.1186/s13321-017-0256-5 Артикул PubMed PubMed Central Google Scholar Dong J, Yao Z-J, Zhu M-F, Wang N-N, Lu B, Chen AF, Lu A-P, Miao H, Zeng W-B, Cao D-S (2017) ChemSAR: онлайн-конвейерная платформа для молекулярного моделирования SAR. Журнал Cheminform 9 (1): 27. https://doi.org/10.1186/s13321-017-0215-1 Артикул PubMed PubMed Central Google Scholar Цилики Г., Мунтяну С. Р., Сеоан Дж. А., Фернандес-Лозано С., Саримвейс Х., Виллигхаген Е. Л. (2015) Rregrs: пакет r для автоматизированного выбора моделей с несколькими моделями регрессии. J Cheminform 7:46. https://doi.org/10.1186/s13321-015-0094-2 CAS Статья PubMed PubMed Central Google Scholar Murrell DS, Cortes-Ciriano I, van Westen GJP, Stott IP, Bender A, Malliavin TE, Glen RC (2015) Химически осведомленный конструктор моделей (camb): пакет r для моделирования свойств и биоактивности малых молекулы.J Cheminform 7:45. https://doi.org/10.1186/s13321-015-0086-2 CAS Статья PubMed PubMed Central Google Scholar Shamsara J (2017) Ezqsar: пакет R для разработки моделей QSAR непосредственно из структур. Откройте Med Chem J 11: 212–221. https://doi.org/10.2174/1874104501711010212 CAS Статья PubMed PubMed Central Google Scholar Nantasenamat C (2020) Лучшие практики построения воспроизводимых моделей QSAR. В: Roy K (ed) Ecotoxicological QSARs. Humana Press, Нью-Джерси Google Scholar Rule A, Birmingham A, Zuniga C, Altintas I, Huang SC, Knight R, Moshiri N, Nguyen MH, Rosenthal SB, Pérez F, Rose PW (2019) Десять простых правил для написания и распространения результатов вычислительного анализа в ноутбуках jupyter. PLoS Comput Biol 15 (7): 1007007 Статья Google Scholar Landrum G (2019) RDKit руководства. Доступно в Интернете: https://github.com/greglandrum/. По состоянию на 1 ноября 2019 г. RDKit (2019) RDKit: Программное обеспечение для химинформатики с открытым исходным кодом. https://www.rdkit.org/. По состоянию на 1 ноября 2019 г. RDKit GitHub (2019) RDKit. https://github.com/rdkit/rdkit-tutorials/. По состоянию на 1 ноября 2019 г. OpenEye Scientific Software, Inc (2019) OpenEye Python Cookbook. https://docs.eyesopen.com/toolkits/cookbook/python/.По состоянию на 1 ноября 2019 г. Informatics Matters Ltd (2019) Squonk Computational Notebook. https://squonk.it/. По состоянию на 1 ноября 2019 г. CDK (2019) Chemistry Development Kit: модульные библиотеки Java с открытым исходным кодом для Cheminformatics. https://cdk.github.io/. По состоянию на 1 ноября 2019 г. Янсен Дж. М., Корнелл В., Ценг Ю. Дж., Amaro RE (2012) Teach-Discover-Treat (TDT): совместное компьютерное открытие лекарств для забытых болезней. Модель графа Дж. Мола 38: 360–362.https://doi.org/10.1016/j.jmgm.2012.07.007 CAS Статья PubMed PubMed Central Google Scholar Riniker S, Landrum GA, Montanari F, Villalba SD, Maier J, Jansen JM, Walters WP, Shelat AA (2017) Учебные пособия по рабочему процессу виртуального скрининга и предполагаемые результаты конкурса Teach-Discover-Treat 2014 против малярия. F1000 Res 6: 1136. https://doi.org/10.12688/f1000research.11905.2 Артикул Google Scholar Riniker S, Landrum GA, Montanari F, Villalba SD, Maier J, Jansen, JM, Walters WP, Shelat AA (2019) Учебное пособие для конкурса Teach-Discover-Treat (TDT) 2014-Challenge 1: обнаружение результатов противомалярии с использованием прогнозных моделей, усиленных слиянием классификаторов. https://github.com/sriniker/TDT-tutorial-2014/. По состоянию на 1 ноября 2019 г. Sydow D, Morger A, Driller M, Volkamer A (2019) TeachOpenCADD: обучающая платформа для компьютерного дизайна лекарств с использованием пакетов и данных с открытым исходным кодом.J Cheminform 11:29. https://doi.org/10.1186/s13321-019-0351-x Артикул PubMed PubMed Central Google Scholar ( 2016) команда разработчиков, Дж .: Блокноты Jupyter - формат публикации для воспроизводимых вычислительных рабочих процессов. В: Loizides F, Scmidt B (ред.) Позиционирование и власть в академическом издательстве: игроки, агенты и повестки дня.IOS Press, Амстердам, стр. 87–90. https://eprints.soton.ac.uk/403913/ Грюнберг Р., Нильгес М., Лекнер Дж. (2007) Biskit - программная платформа для структурной биоинформатики. Биоинформатика 23 (6): 769–770. https://doi.org/10.1093/bioinformatics/btl655 Артикул PubMed Google Scholar Данилюк П., Вильчинский Б., Лесинг Б. (2015) WeBIAS: веб-сервер для публикации приложений биоинформатики.Примечания BMC Res 8: 628. https://doi.org/10.1186/s13104-015-1622-x CAS Статья PubMed PubMed Central Google Scholar Osz Á, Pongor LS, Szirmai D, Gyorffy B (2017) Снимок 3649 веб-сервисов, опубликованных в период с 1994 по 2017 год, показывает снижение доступности через 2 года. Краткий биоинформ. https://doi.org/10.1093/bib/bbx159 Артикул PubMed Central Google Scholar RStudio Inc. (2018) Блестящий. https://shiny.rstudio.com/ Plotly (2019) Dash. https://plot.ly/products/dash/. По состоянию на 9 января 2019 г. Plotly (2019) Plotly: современные аналитические приложения для предприятий. https://plot.ly/. По состоянию на 9 января 2019 г. Nantasenamat C (2019) Концептуальная карта компьютерного открытия лекарств [CC-BY]. https://doi.org/10.6084/m9.figshare.5979400 Synergy Research Group (2019) Ведущие облачные провайдеры продолжают убегать вместе с рынком.https://www.srgresearch.com/articles/leading-cloud-providers-continue-run-away-market/. По состоянию на 9 января 2019 г. Dong J, Yao ZJ, Wen M, Zhu MF, Wang NN, Miao HY, Lu AP, Zeng WB, Cao DS (2016) Biotriangle: веб-платформа для создания различных молекулярных представления для химических веществ, белков, ДНК / ДНК и их взаимодействия. J Cheminform 8:34. https://doi.org/10.1186/s13321-016-0146-2 CAS Статья PubMed PubMed Central Google Scholar Dong J, Cao D-S, Miao H-Y, Liu S, Deng B-C, Yun Y-H, Wang N-N, Lu A-P, Zeng W-B, Chen AF (2015) Chemdes: интегрированная веб-платформа для вычисления молекулярных дескрипторов и отпечатков пальцев. J Cheminform 7:60. https://doi.org/10.1186/s13321-015-0109-z CAS Статья PubMed PubMed Central Google Scholar Walker T, Grulke CM, Pozefsky D, Tropsha A (2010) Chembench: инструмент для хеминформатики.Биоинформатика 26 (23): 3000–3001. https://doi.org/10.1093/bioinformatics/btq556 CAS Статья PubMed PubMed Central Google Scholar Сушко И., Новотарский С., Кёрнер Р., Пандей А.К., Рупп М., Титц В., Брандмайер С., Абдельазиз А., Прокопенко В.В., Танчук В.Ю. и др. (2011) Среда онлайн-химического моделирования (OCHEM): веб-платформа для хранения данных, разработки моделей и публикации химической информации. J Comput Aided Mol Des 25 (6): 533–554.https://doi.org/10.1007/s10822-011-9440-2 CAS Статья PubMed PubMed Central Google Scholar Гонсалес-Медина М., Медина-Франко Дж. Л. (2017) Платформа для унифицированного молекулярного анализа: Puma. J Chem Inform Model 57 (8): 1735-1740. https://doi.org/10.1021/acs.jcim.7b00253 CAS Статья Google Scholar van Zundert GCP, Rodrigues JPGLM, Trellet M, Schmitz C, Kastritis PL, Karaca E, Melquiond ASJ, van Dijk M, de Vries SJ, Bonvin AMJJ (2016) Пикша2.2 веб-сервер: удобное интегративное моделирование биомолекулярных комплексов. J Mol Biol 428 (4): 720-725. https://doi.org/10.1016/j.jmb.2015.09.014 CAS Статья PubMed Google Scholar Camps J, Carrillo O, Emperador A, Orellana L, Hospital A, Rueda M, Cicin-Sain D, D'Abramo M, Gelpí JL, Orozco M (2009) FlexServ: интегрированный инструмент для анализа гибкости белка. Биоинформатика 25 (13): 1709–1710. https: // doi.org / 10.1093 / bioinformatics / btp304 CAS Статья PubMed Google Scholar Hospital A, Andrio P, Fenollosa C, Cicin-Sain D, Orozco M, Gelpí JL (2012) MDWeb и MDMoby: интегрированная веб-платформа для моделирования молекулярной динамики. Биоинформатика 28 (9): 1278–1279. https://doi.org/10.1093/bioinformatics/bts139 CAS Статья PubMed Google Scholar Stierand K, Maass PC, Rarey M (2006) Краткий обзор молекулярных комплексов: автоматическое создание двумерных сложных диаграмм. Биоинформатика 22 (14): 1710–1716. https://doi.org/10.1093/bioinformatics/btl150 CAS Статья PubMed Google Scholar Biasini M, Bienert S, Waterhouse A, Arnold K, Studer G, Schmidt T, Kiefer F, Gallo Cassarino T, Bertoni M, Bordoli L, Torsten S (2014) Швейцарская модель: моделирование третичного и четвертичная структура с использованием эволюционной информации.Nucleic Acids Res 42 (выпуск веб-сервера): 252–8. https://doi.org/10.1093/nar/gku340 CAS Статья Google Scholar Биоинформатика Бабраама - Учебные курсыУчебные курсыВ рамках сотрудничества с Институтом Бабраама группа биоинформатики проводит регулярные серии учебных курсов по многим аспектам биоинформатики. Эти курсы регулярно проводятся на сайте Babraham, но мы также можем выходить и представлять их на других сайтах, а также доставлять их удаленно.Вы можете увидеть список текущих дат Бабраама которые доступны, и вы можете связаться с нами обсудить варианты проведения курсов на вашем сайте. Вы также можете подписаться на нашу рассылку, чтобы получать последние новости о тренингах прямо на ваш почтовый ящик каждые пару месяцев. По возможности мы также стремимся сделать материалы наших курсов общедоступными. доступны, так что любой желающий может скачать их самостоятельно использовать. Ниже приведен список курсов, которые мы проводим в настоящее время.Где они доступны есть ссылка на учебное пособие и курсовые упражнения. Основные навыки в области биоинформатики
Статистика
Курсы, ориентированные на приложения
Комплексные более длительные курсы BootcampАнализ данных отображенных последовательностей с помощью SeqMonk (один день)SeqMonk это программа, которая может анализировать большие наборы данных картированные геномные позиции.Чаще всего используется для работы с данными, поступающими из конвейеров секвенирования с высокой пропускной способностью. Программа позволяет просматривать ваши показания на фоне аннотированного генома, а также для количественной оценки и фильтрации ваших данных, чтобы вы определить интересующие регионы. Это удобный способ исследовать и анализировать очень большие наборы данных. Этот курс знакомит с основными функциями SeqMonk и проведем анализ пары различные наборы данных, чтобы показать, какие варианты анализа обеспечивает. Содержание курса
Материал курса:Статистический анализ с использованием R (один день)Статистика - важная часть большинства современных исследований и возможности эффективно использовать статистический пакет может помочь вам понять ваши полученные результаты.Этот курс представляет собой введение в статистику, проиллюстрированную хотя использование языка R. Содержание курса:
Материал курса:Статистический анализ с использованием GraphPad Prism (один день)GraphPad Prism - мощный и удобный пакет, который позволяет вам строить графики. и проанализируйте ваши данные.Этот курс действует не только как введение в Prism, но также проходит базовые статистические знания, которые должны позволяют максимально использовать ваши данные. Содержание курса:
Материал курса:Статистика буткемпа с использованием R (3 дня)Более подробный анализ статистического анализа с использованием R. Предварительное условие: Введение в R с Tidyverse (1 день) Модули содержания курса:
Модули материалов курса:Учебный курс по статистике с использованием GraphPad Prism (2.5 дней)Более подробный анализ статистического анализа с помощью GraphPad Prism Модули содержания курса:
Материал курса:Обучение программированию на Perl (6 x 1.5 час. Сеансов)В течение долгого времени Perl был популярным языком среди тех, кто только начинал программировать. Хотя это мощный язык, многие из его особенности делают его особенно подходящим для начинающих программистов, так как он снижает сложность, характерную для многих других языков. Perl тоже один самых популярных языков мира, а это значит, что существует огромное количество ресурсов, доступных любому желающему его изучить. Этот курс направлен на ознакомление с основными функциями языка Perl.В итоге у вас должно быть все необходимое, чтобы писать в меру сложный программы и достаточно указателей на другие ресурсы, чтобы вы начали более крупные проекты. Курс пытается дать базовые знания теории вам нужно будет писать программы на любом языке, а также признательность за правильный способ делать что-то в Perl. Содержание курса:
Материал курса:Введение в Python (двухдневный учебный курс)В последние годы язык программирования Python становится все более популярным в биоинформатике. сообществами вычислительной биологии, и действительно, изучение этого языка знаменует собой первое введение в написание кода.Этот успех Python обусловлен рядом факторов. Возможно, самое главное для новичка, Python относительно прост в использовании, быть тем, что мы называем языком программирования «высокого уровня». Однако пусть эта терминология вас не смущает: "высокий уровень" просто означает, что большая часть вычислительных задач решается за вас, что позволяет вам напишите более короткий и простой код, чтобы выполнять свою работу. Этот курс знакомит с основными функциями Python3. По окончании этого курса вы научитесь писать умеренно. сложные программы, и будьте в курсе дополнительных ресурсов и более широких возможностей языка, чтобы браться за более серьезные проекты.Курс пытается дать базовую основу теории. вам нужно будет писать программы на любом языке, а также знать, как правильно делать вещи в Python. Содержание курса:
Материал курса:Продвинутый Python (двухдневный учебный курс)В последние годы язык программирования Python становится все более популярным в биоинформатике. сообществами вычислительной биологии, и действительно, изучение этого языка знаменует собой первое введение в написание кода.Этот успех Python обусловлен рядом факторов. Возможно, самое главное для новичка, Python относительно прост в использовании, быть тем, что мы называем языком программирования «высокого уровня». Однако пусть эта терминология вас не смущает: "высокий уровень" просто означает, что большая часть вычислительных задач решается за вас, что позволяет вам напишите более короткий и простой код, чтобы выполнять свою работу. Этот курс основан на основных функциях Python3, представленных в курсе Introdcution to Python.По окончании этого курса вы научитесь писать умеренно. сложные программы, и будьте в курсе дополнительных ресурсов и более широких возможностей языка, чтобы браться за более серьезные проекты. Курс пытается дать базовую основу теории. вам нужно будет писать программы на любом языке, а также знать, как правильно делать вещи в Python. Содержание курса:
Материал курса:Понимание объектно-ориентированного Python (один день)Сильной стороной Python и особенностью, которая делает этот язык привлекательным для многих, является то, что Python - это так называемый объектно-ориентированный язык программирования (ООП). Это краткий курс, знакомящий с основными концепциями ООП. Затем более подробно объясняется, как создавать объекты и манипулировать ими. Хотя этот курс не обеспечивает исчерпывающего обсуждения ООП в Python, к концу курса слушатели должны быть в состоянии создавать сложные объекты для анализа и исследования. Содержание курса:
Материал курса:Введение в R с Tidyverse (один день)R - популярный язык и среда, позволяющая мощно и быстро манипулировать data, предлагая множество статистических и графических опций.Этот курс направлен на ознакомление с R как инструментом для статистики и графики, основная цель которого состоит в том, чтобы освоитесь в среде R. Помимо представления основных концепций языка R, это курс также предоставляет основы использования Tidyverse для обработки данных и ggplot для заговор. Он будет сосредоточен на вводе и обработке данных в R, а также на создании простых графиков. Несколько функций для базовых будет кратко представлена статистика, но статистические функции не будут рассматриваться подробно. Содержание курса:
Материал курса:Материалы после курса:Знакомство с Core R (полдня)R - популярный язык и среда, позволяющая мощно и быстро манипулировать data, предлагая множество статистических и графических опций.Этот курс направлен на ознакомление с R как инструментом для статистики и графики, основная цель которого состоит в том, чтобы освоитесь в среде R. Он будет сосредоточен на вводе и обработке данных в R, а также на создании простых графиков. Несколько функций для базовых будет кратко представлена статистика, но статистические функции не будут рассматриваться подробно. Содержание курса:
Материал курса:Advanced Core R (полдня)Этот курс является продолжением вводного курса.Здесь более подробно рассказывается о практических руководства по фильтрации и объединению сложных наборов данных. Также рассматриваются другие основные концепции R например, цикл с операторами применения и использование пакетов. Наконец, это выглядит о том, как документировать ваши анализы R и создавать полные аналитические отчеты. Содержание курса:
Материал курса:Построение сложных фигур с помощью Core R (полдня)Этот курс представляет собой исчерпывающее руководство по использованию встроенного построения графиков R. функциональность для построения всего, от индивидуальных простых графиков до сложных многослойные фигуры.Как следует из материала нашего вводного R курс и участники должны иметь базовое понимание R - достаточно для загрузки и выполнения основных операций с наборами данных. Содержание курса:
Материал курса:Advanced R с Tidyverse (один день)Tidyverse - это набор надстроек R-пакетов для загрузки данных, моделирования, обработки и построения графиков.Это - это попытка сделать анализ данных и построение графиков более чистыми, простыми и последовательными, решив некоторые плохие дизайнерские решения на языке оригинала. Этот курс следует из нашего введения в R с tidyverse и фокусируется на манипуляциях и реструктуризация данных с помощью пакетов tidyverse. В курсе показано, как выполнять сложные преобразования на большие структуры данных и как эффективно работать с данными, которые одновременно велики, а иногда и не очень хорошо вел себя. Содержание курса:
Материал курса:Использование ноутбуков R (полдня)Этот курс предназначен для людей, которые уже знакомы с R и готовы для более интегрированного способа выполнения и составления отчетов об их анализе.Он покажет использовать R Notebooks для интерактивного анализа, а затем продемонстрировать, как применять это для производства полных отчетов. Содержание курса:
Материал курса:Построение фигур с помощью ggplot (Один день)Этот курс обычно преподается как часть учебного лагеря R с Tidyverse.Ggplot - самое популярное расширение для R для построения графиков, которое копирует многие из типы графиков, найденные в основных библиотеках построения графиков. Этот курс дает введение в библиотеки ggplot2 и практическое руководство по используйте их для создания различных типов графиков. Содержание курса:
Материал курса:Написание пакетов R (один день)Пакеты R - лучший способ создать надежный код многократного использования для внутреннего использования. использовать или поделиться с более широким сообществом.В этом курсе мы рассмотрим, как писать функции, которые будут надежными для использования другими. Затем мы рассмотрим процесс создания пакетов R на основе функций с помощью рекомендуемых Инструменты разработки. Содержание курса:
Материал курса:Знакомство с Shiny (один день)Shiny - это пакет R, который позволяет создавать интерактивные веб-приложения с использованием R.Это отличный способ позволить пользователям исследовать набор данных и использовать графические и статистические функции R без необходимости писать какой-либо код. Содержание курса:Этот курс представляет собой комбинацию бесед и практических упражнений. Он охватывает концепции, необходимые для создания работающего приложения Shiny, в том числе:
Чтобы писать приложения на Shiny, вам должно быть комфортно использовать R.Перед посещением этого курса студентам рекомендуется пройти вводный и продвинутый курсы R (основной или тидиверс). Материалы курса:Введение в Unix (полдня)Все больше работы по биоинформатике выполняется из командной строки. среда unix. Написаны самые крупномасштабные приложения для обработки для unix и большинства крупномасштабных вычислительных сред также основаны на этом. Этот курс дает введение в концепции unix и предоставляет практическое введение в работу в этой среде.Внутри мы свяжите этот курс с более конкретным курсом, иллюстрирующим использование нашего внутренняя кластерная среда, и эта часть курса может быть адаптирована для других сайтов с другой вычислительной инфраструктурой Содержание курса:
Материал курса:Введение в машинное обучение (один день)В этом курсе машинное обучение рассматривается как инструмент анализа биологических вопросов.В нем рассматриваются основные концепции машинного обучения и обсуждаются практические аспекты его использования в интеллектуальном анализе данных. Курс представляет собой сочетание лекций и практических занятий с использованием графического программного обеспечения Weka (навыки программирования не требуются). Содержание курса:
Материалы курса (скоро):Анализ данных секвенирования метилирования бисульфита (один день)Этот курс основан на основных навыках, представленных во введении в R, Введение в Unix и курсы Введение в SeqMonk, чтобы предоставить больше более подробно рассмотрим анализ данных секвенирования бисульфита.Курс представляет собой сочетание теоретических лекций и практических занятий, которые проходят через весь конвейер анализа, начиная с необработанных данных последовательности и охватывая Контроль качества, визуализация, количественный анализ и анализ дифференциального метилирования. Содержание курса:
Материал курса:Извлечение биологической информации из списков генов (один день)Многие экспериментальные проекты в конечном итоге создают списки совпадений, обычно основанные на вокруг генов или транскриптов.Иногда эти списки достаточно малы что их можно исследовать индивидуально, но часто бывает полезно сделать более структурированный функциональный анализ, чтобы попытаться автоматически определить любые интересные биологические темы, которые встречаются в списках. В этом курсе рассматриваются различные программные пакеты, базы данных и статистические методы, которые могут быть использованы при проведении такого анализа. Помимо того, что это практическое руководство по выполнению этих типов Анализ курса также рассмотрит типы артефактов и предвзятости что может привести к ложным выводам о функциональности и найдите подходящие способы как провести анализ, так и представить результаты для публикации. Содержание курса:
Материал курса:Анализ ChIP-Seq (один день)Этот курс дает полное введение в теорию и практику анализа Данные ChIP-Seq.Он предназначен для биологов, которые могут иметь ограниченную практическую биоинформатику. навыки, но которые хотели бы использовать ChIP-Seq как часть своей работы. К концу курса студенты должны уметь обрабатывать и анализировать свои собственные данные. Студентам этого курса будет полезно посетить введение в SeqMonk или Unix. курсы, но они не обязательны для посещения. Содержание курса:
Материал курса:Анализ РНК-Seq (один день)Этот курс предоставляет введение в QC, обработку и анализ. данных RNA-Seq.Он фокусируется на рабочем процессе, в котором RNA-Seq выполняется на большой эукариотический геном, для которого доступен эталонный геном. Курс начинается с всеобъемлющей лекции, посвященной теории Генерация и анализ данных RNA-Seq, а затем практические практические занятия, которые проходят через весь конвейер анализа RNA-Seq от необработанных файлов fastq до списка дифференциально экспрессируемых генов-кандидатов. Содержание курса:
Материал курса:10-кратный анализ РНК-секвенирования одной клетки (один день)Этот курс дает практическое введение в обработку, контроль качества и анализ простого эксперимента RNA-Seq с одной клеткой, выполненного на Платформа 10X.В нем объясняется технология, используемая для создания данных и проходит через некоторые общие инструменты анализа. Курс также проходит теория и практика методов уменьшения размерности, которые очень часто используются для представления такого рода данных. Содержание курса:
Материал курса:Контроль качества в экспериментах по секвенированию (полдня)В этом курсе рассматриваются различные способы провала исследований, основанных на секвенировании. а также варианты визуализации и контроля качества, которые позволяют идентифицировать и диагностировать эти сбои на ранней стадии.Он разработан, чтобы быть полезным для всех, кто используют секвенирование как часть своих исследований, а не только те, кто занимается секвенированием удобства. Содержание курса:
Материал курса:Введение в математическое моделирование (полдня)Этот курс был разработан в сотрудничестве с лабораторией Le Novère в Институте Бабраама.Курс в настоящее время не работает и не поддерживается, но мы оставляем здесь материалы курса для справки. Он представляет собой введение в концепции моделирования биологических систем. это предназначен для биологов, которые не имеют опыта моделирования, но хотели бы знать, как это может применимы к своей области исследования. Курс дает исчерпывающую справочную информацию по истории моделирование и различные подходы, с помощью которых биологическая система может быть аппроксимирована математические методы.Курс также представляет собой практическое введение в Среда моделирования COPASI. Содержание курса:
Материал курса:Научный дизайн фигур (целый день)Этот курс представляет собой практическое руководство по созданию фигур для использования в отчеты и публикации. Это обширный курс, который рассматривает как проектировать фигуры, чтобы четко и справедливо представлять ваши данные, практические аспекты построения графа, допустимые манипулирование растровыми изображениями и компоновка и редактирование окончательных фигур. В курсе будет использоваться ряд различных пакетов программного обеспечения с открытым исходным кодом и проиллюстрирован рядом примеров рисунков, адаптированных из распространенных инструментов анализа. Содержание курса:
Материал курса:Введение в использование OneNote в качестве лабораторной записной книжки (полдня)Этот курс представляет собой практическое руководство по использованию Microsoft OneNote в качестве лабораторной записной книжки с особым вниманием к методам, политикам, ожиданиям и обязанностям в Институте Бабрахама .Курс будет использовать OneNote онлайн как кроссплатформенное приложение. Содержание курса:
Материал курса:Учебный курс "Введение в R для биологов" (3.5 дней)Этот учебный лагерь для биологов не требует предыдущего опыта. В течение 3 1/2 дней вы приобретете практический опыт проведения собственного анализа в р. .Содержание курса:
Материал курса:Учебный курс по NGS-анализу для биологов (3.5 дней)Этот учебный лагерь для биологов не требует предыдущего опыта. За 3 1/2 дня вы познакомитесь с секвенированием анализа с нуля. Поймите, исследуйте и проанализируйте свои данные и интерпретируйте результаты. Содержание курса:
Материал курса:Введение в Linux Bootcamp (2.5 дней)Этот учебный курс для биологов не требует предыдущего опыта и даст представление о среде Linux. Этот 2 1/2 дневный курс показывает, как настроить рабочая среда Linux; как вы можете устанавливать, настраивать и управлять программным обеспечением и пакетами в нем; как запускать программное обеспечение и создавать базовую простую автоматизацию, чтобы обеспечить выполнение более структурированным и масштабируемым способом. Содержание курса:
Материал курса:Jumpstart Создание заметок с помощью шаблонов OneNote - buckleyPLANETЯ открыл для себя OneNote еще в 2004 году, и с тех пор это мой основной инструмент для создания контента.Можно сказать, что я официально # фанат этого продукта. Одна из (многих) вещей, которые Microsoft сделала правильно с запуском Microsoft Teams, заключалась в том, чтобы включить OneNote в платформу в первый день, IMHO. Как и во многих других основных продуктах Microsoft Office (Excel, Word и т. Д.), Продукт обладает огромной глубиной, и я постоянно нахожу новые функции и способы их использования. Организации все чаще используют OneNote в качестве общего ресурса для создания заметок для собраний и совместной работы в команде.Независимо от того, создаете ли вы новые записные книжки с каждой командой Microsoft или используете отдельные или общие записные книжки для собраний на основе Outlook, использование OneNote растет. У большинства моих клиентов все заметки о встречах фиксируются и передаются единообразно, что позволяет заинтересованным сторонам быстро просматривать эти общие заметки и действовать в соответствии с ними. Если вы похожи на меня, когда я создавал формат для заметок на собраниях, я часто вырезал и вставлял макет записной книжки между проектами. Но вместо того, чтобы каждый раз заново создавать макет заметок о собраниях, знаете ли вы, что можно быстро и легко вставить заметки в формате с помощью шаблонов OneNote? Преимущества использования шаблона заключаются в том, что он обеспечивает согласованность, поддерживает чайМ и брендинг компании, а также упрощает передачу другой важной повторяющейся информации (пространства для названий проектов и владельцев, ссылки на ресурсы и другие ключевые метаданные). Я поделился этим советом на веб-семинаре с советами по продуктивности в августе 2019 года, но хотел расширить эту функцию здесь, в блоге. Как всегда, вы можете просмотреть весь выпуск, загрузить слайды через SlideShare или перейти к конкретному совету в записи, щелкнув здесь. Добавление существующего шаблонаПрямо из коробки Microsoft предлагает ряд шаблонов, которые можно использовать для быстрого начала ведения заметок, от обзоров проектов и заметок о собраниях до личных списков дел и декоративных макетов. Чтобы открыть шаблон, просто перейдите в Вставка > Шаблоны страниц и откройте Шаблоны страниц , чтобы развернуть меню шаблона. Если вы регулярно используете эти шаблоны, в меню будут представлены несколько последних использованных шаблонов. В меню шаблонов вы можете быстро выполнить поиск среди существующих шаблонов и вставить их в открытую записную книжку и даже установить избранный шаблон в качестве шаблона по умолчанию. В результате вы получите новую страницу, прекрасно отформатированную и готовую для заметок.Конечно, каждый аспект этого шаблона может быть изменен, заклеймен и персонализирован в соответствии с вашими потребностями, а затем сохранен как ваш собственный шаблон. Создание шаблонаИ да, вы можете создавать свои собственные шаблоны с нуля. Если вы потратили бесчисленное количество часов на создание формата, который вы повторно используете для новых проектов и встреч, просто откройте консоль шаблона и выберите Создать новый шаблон и сохраните свой формат как шаблон. Вам будет предложено дать вашему шаблону имя и дать вам возможность сохранить этот новый дизайн в качестве шаблона по умолчанию для текущего раздела OneNote. В последующих разделах и записных книжках вам нужно будет вручную настроить этот новый шаблон как ваш любимый, поскольку для шаблонов по умолчанию нет глобальных настроек. Использовать шаблоны OneNote быстро и легко, и, надеюсь, вы найдете этот совет полезным! . |

 jpg» align=»top»>
jpg» align=»top»> По умолчанию рамка всегда есть. Если вы хотите убрать рамку, то задайте border=0.
По умолчанию рамка всегда есть. Если вы хотите убрать рамку, то задайте border=0.