Как привязать ВК к другому номеру телефона
Привязывать номер мобильного телефона к новому номеру очень просто. С этой задачей сможет справиться каждый пользователь за считанные минуты.
Изменить контактную информацию можно в том случае, если у пользователя имеется доступ к старому. Однако, если этот доступ отсутствует, то процесс может растянуться приблизительно на 14 дней. На новый номер высылают код для подтверждения, а помимо этого пользователь должен будет подтверждать свои действия, используя адрес электронной почты, который был привязан ранее. Это делается ради полной безопасности пользователей.
Содержание
- Зачем привязывать ВКонтакте к другому номеру телефона
- Как привязать ВК к другому номеру телефона через настройки
- Меняем номер с помощью блокировки аккаунта
- Как перепривязать номер
- Как можно ускорить процесс изменения номера
- Как часто можно менять номер телефона в Вконтакте
Зачем привязывать ВКонтакте к другому номеру телефона
Многие пользователи, которые регистрировались в этой социальной сети после ее появления, помнят, что в то время не нужно было указывать номер мобильного телефона при регистрации. Однако, спустя 13 лет эта функция появилась, и теперь каждый пользователь должен привязывать номер мобильного телефона к своей странице.
Однако, спустя 13 лет эта функция появилась, и теперь каждый пользователь должен привязывать номер мобильного телефона к своей странице.
Благодаря этой функции пользователь не потеряет доступ к своей личной странице. Например, он забыл пароль, но если страница была привязана к контактному номеру, то ее можно восстановить благодаря этому. Главное, чтобы телефон всегда оставался под рукой.
Кроме этого, есть возможность изменить настройки так, что пользователь будет уведомляться о всех действиях в этой социальной сети. Например, ему будут приходить уведомления о новых сообщениях, комментариях и отметках «мне нравится».
Также, необходимо привязывать страницу в Вконтакте к другому номеру, если пользователь планирует менять телефон.
Личный номер телефона используется как логин для входа в Вконтакте.
Как привязать ВК к другому номеру телефона через настройки
Если у пользователя есть доступ к старому номеру телефона, то он может достаточно быстро изменить его на новый в настройках социальной сети.
Сделать это можно следующим образом:
- Для начала потребуется открыть Вконтакте и найти там раздел «настройки». Он находится в правом верхнем углу;
- Открывшееся меню необходимо пролистать в самый низ, где будет находиться пункт «Номер телефона». Нужно нажать на кнопку «изменить»;
- После этого должно появиться окошко, в которое потребуется ввести новый контактный номер, а затем нужно кликнуть на «получить код»;
- Как только придет код необходимо вписать его в специальное окошко.
После этих действий заявка пользователя на смену привязанного номера будет принята, однако его изменять только спустя 14 дней. Но, этот процесс можно легко ускорить, если имеется доступ к старым контактам. Сделать это можно кликнув на предложение об ускорении процесса.
Меняем номер с помощью блокировки аккаунта
Если телефон, который необходимо привязать к странице, привязан к другому профилю, то можно попробовать его заблокировать. После этого от заблокированной страницы можно отвязать этот номер и использовать его по назначению.
Однако, если администрация Вконтакте заблокирует страницу навсегда, то на этот же номер телефона нельзя будет в будущем создавать новые аккаунты.
Получить блокировку в этой социальной сети можно делая различные рассылки в комментариях сообществ или отправляя их в личные сообщения другим пользователям.
Также это можно сделать, используя специальные сервисы для накрутки отметок «мне нравится» и комментариев. После того, как страница будет заблокирована администрацией, можно будет отвязать от нее номер.
Для этого потребуется иметь под рукой SIM-карту, которая не привязана к какому-либо аккаунту в Вконтакте. Затем необходимо зайти на замороженный аккаунт и ввести новые контактные данные, а после нужно нажать на «продолжить». На этом этапе должно появиться окно, в котором нужно ввести код подтверждения новый пароль.
Причины, по которым страницу могут заблокировать
Чаще всего профили блокируют по двум основным причинам. А именно:
- Самая распространённая – нарушение правил этой сети.
 К этим нарушениям относится спас, оскорбление других людей, призывы к насилию, а также накрутка отметок «мне нравится» или комментариев и неиспользование аккаунта.
К этим нарушениям относится спас, оскорбление других людей, призывы к насилию, а также накрутка отметок «мне нравится» или комментариев и неиспользование аккаунта. - Взлом аккаунта. Такие случаи, как правило, происходят редко, но администрация всегда сразу принимает меры по решению этой проблемы.
Также можно посмотреть видео о том, как восстановить страницу в Вконтакте:
Как перепривязать номер
Для того, чтобы перепривязать номер в Вконтакте сначала потребуется отвязать его от страницы. Для этого снова необходима новая SIM-карта.
Лучше всего для этого приобрести новую SIM-карту.
Отвязать номер можно следующим образом:
- Для начала необходимо зайти в раздел «настройки», и найти пункт «номер телефона»;
- Затем пользователь должен кликнуть на пункт «изменить», он расположен справа от привязанного номера;
- В открывшемся окне необходимо вписать новый номер телефона, а пришедший код подтверждения нужно написать в специальное окошко.

После этого, следуя этому же плану необходимо снова привязать старый телефон к аккаунту вместо нового.
Так же можно посмотреть видео по ссылке о том, как отвязать телефон в Вконтакте:
Как можно ускорить процесс изменения номера
Нередко бывают ситуации, когда изменить контактную информацию нужно очень быстро. Например, при подозрении на взлом профиля. В таком случае необходимо подать заявку в службу технической поддержки с просьбой об изменении номера, который привязан к странице.
Для того, чтобы администрация технической поддержки рассмотрела и приняла эту заявку, необходимо заполнять ее полностью и внимательно. Не менее важно предоставлять достоверную информацию.
Если пользователь не предоставит достаточно необходимых доказательств о том, что это его страница, то администраторы технической поддержки не ободрят отправленную заявку.
Как часто можно менять номер телефона в Вконтакте
Нельзя слишком часто менять контактную информацию телефонов в этой социальной сети, поскольку администрация может заблокировать страницу навсегда без возможности восстановления.
Максимально можно изменять контактную информацию не более трех раз.
Вячеслав
Несколько лет работал в салонах сотовой связи двух крупных операторов. Хорошо разбираюсь в тарифах и вижу все подводные камни. Люблю гаджеты, особенно на Android.
Задать вопрос
Можно ли указывать несуществующий номер телефона при изменении этой информации?
К сожалению, так сделать нельзя, поскольку потребуется вводить код подтверждения в любом случае.
Что делать если пользователь случайно удалил страницу?
В данном случае можно попробовать либо восстановить ее, либо отвязать телефон от этой страницы, следуя вышеописанной инструкции. Также можно написать в службу поддержки.
Что делать если страница была взломана, а пароль изменен?
В таком случае вернуть доступ к странице можно только написав в службу поддержки.
Как зарегистрироваться в контакте без номера телефона
С некоторых пор социальная сеть ужесточила правила регистрации новых аккаунтов, сделав обязательной привязку к мобильному телефону.
Теперь при регистрации необходимо использовать номер телефона.
Рядовым пользователям это поможет сохранить свою страницу в безопасности. А вот использование сети для специальных нужд несколько усложнилось.
Содержание:
Тех, кого интересует как зарегистрироваться в контакте через электронную почту придется разочаровать, – такой возможности больше не существует.
Обратите внимание! Администрация Вконтакте исключила возможность регистрации учетной записи посредством одного только почтового ящика в 2012 году. Продиктовано это решение было соображениями безопасности и оптимизации работы социальной сети.
Во всяком случае, способ регистрации аккаунта без использования собственного номера телефона все еще существует.
Для этого понадобится использовать сервер виртуальной телефонии для приема СМС-сообщений.
Proovl SMS
Proovl SMS предлагает платный сервис для приема любого СМС, от любого отправителя или сервиса на SIM номера в США и Англии.
Все номера новые и доступны к использованию, через веб панель. Так же Proovl, предлагает виртуальные номера в 29 странах с возможностью пересылки сообщений на телефон, URL или e-mail.
Ссылка на сайт
Pinger позволяет получить виртуальный номер, на который можно получать сообщения.
Процесс регистрации в Pinger
Итак, как зарегистрироваться в контакте если уже есть страница? Для начала понадобится посетить страницу сервиса.
Форма для входа существующего пользователя нам пока что не понадобится, поэтому нужно выбрать пункт «Create account» (создать аккаунт).
Важно! Для полноценного использования сервиса понадобиться свежая версия Flash Player.
Выбор этой опции направит пользователя на страницу с регистрационной формой.
Пугаться того, что все пункты на английском не нужно, даже несмотря на то, что переводчик браузера их не переведет. Во всяком случае русской версии сервиса не существует.
Заполнять данную форму необходимо следующим образом:
- Имя пользователя, которое впоследствии станет логином для входа в службу.
- Уникальный безопасный пароль, который также понадобится для входа.
- Подтверждение пароля.
- Адрес электронной почты для получения уведомлений и восстановления доступа в экстренных ситуациях.
- Обязательное соглашение с правилами использования службы
- Кнопка создания аккаунта.
Если поля заполнить неправильно или не подтвердить согласие с правилами использования и политикой конфиденциальности ресурса, то регистрация будет невозможной.
Далее система будет определять наиболее подходящий виртуальный номер для региона пользователя.
Для этого понадобится:
- Ввести почтовый индекс (узнать его поможет этот сайт).
- Выбрать половую принадлежность (Male – мужчина, Female — женщина).
- Указать возраст (лучше указывать возраст более 18-ти лет).
- Нажать кнопку для поиска наиболее подходящего номера телефона.
После этого система некоторое время будет подбирать номера.
В итоге появится их список. Из предложенных нужно выбрать один и подтвердить выбор кнопкой «Confirm».
Выбор происходит один раз, после этого сменить номер для конкретной учетной записи невозможно.
После этого система предложит опубликовать новость о приобретении виртуального мобильного номера на странице Facebook. Это выбор необязательный, поэтому можно просто нажать «Cancel».
После этого откроется страница с менеджером сообщений.
Над ним буде кнопка обновления (пригодится если сообщение долго не приходит).
Чтобы посмотреть собственный виртуальный номер телефона нежно нажать кнопку «Options». Он будет указан в верней части формы, в графе «Phone number».
Его можно выделить и скопировать в буфер обмена (Ctrl+C).
Важно только не копировать ничего после этого, иначе номер сотрется из буфера и придется копировать его заново.
к содержанию ↑
Регистрация в ВК без номера телефона
Теперь можно переходить к регистрации аккаунта в социальной сети. Для этого нужно открыть стартовую страницу и кликнуть «Регистрация» в середине экрана или под формой входа.
Первое, что необходимо сделать – выбрать имя и фамилию. Действительными они будут или псевдонимом – личный выбор пользователя.
Нужно только помнить, что администрация блокирует откровенно выдуманные имена.
Следующий шаг – указание страны и города проживания. Правдивость данных сведений также остается на усмотрении пользователя, который проходит регистрацию.
И, наконец, то, ради чего происходила регистрация в Pinger – подтверждение по номеру телефона:
- В поле выбора страны необходимо указать ту страну, с кода которой начинается только что созданный виртуальный номер (в данном случае это США, код +1).
- Затем ввести номер телефона в международном формате.
- Нажать кнопку, чтобы получить верификационный код.
Под формой появится строка, в которую необходимо ввести полученный код. Посмотреть его можно на странице Pinger, созданной в первой части инструкции.
Кириллические символы подвергаются транслитерации. Если этого не происходит, то вместо них отображаются знаки вопроса.
Но это совершенно неважно, потому что единственные цифры в сообщение – это верификационный код, который нас интересует.
После его введения останется только придумать пароль. Его надежность отображается строкой под полем для ввода. Чем надежнее будет это сочетание символов, тем лучше.
После выбора пароля нужно нажать «Войти на сайт» и можно смело использовать свою вновь созданную страницу.
Если возникают проблемы с конкретным сервисом, предоставляющим услуги виртуального номера, то можно найти в сети другие варианты.
Услуга довольно популярная, поэтому проблем с этим не будет.
Для большей надежности можно использовать платные сервера виртуальных номеров. Плата в большинстве случаев символическая, а пользы предостаточно.
Наиболее дешевыми тарифами обладают службы, предоставляющие возможность получать только текстовые сообщения.
Более того, среди них есть такие, которые специализируются на регистрации в различных социальных сетях и на других ресурсах.
Зарегистрироваться вконтакте без номера телефона 2016
Из данного видео вы узнаете о том как можно создать огромное количество страниц вконтакте не используя при этом номер телефона, при чем это абсолютно бесплатный способ.
9 Рейтинг
Отличный и быстрый способ!
Простое и доходчивое объяснение, если четко следовать инструкции, то все получится менее чем за 15 минут. Никаких сложностей не возникает, все предельно просто и, самое главное, не требует дополнительных финансовых затрат.
Сложность
4
Время
5
Стоимость
10
Как отвязать номер от страницы ВК в 2022 году
Алевтина Зайцева 05.10.2018 Обновлено: 4.03.2020 108 комментариев
Вконтакте – популярная соцсеть, которую ежедневно посещает около 80 миллионов человек, и ежедневно количество пользователей увеличивается. У многих есть сразу несколько страниц Вконтакте, однако с принятием новой политики сайта, оформить новый аккаунт стало сложней.
Содержание
- 1 Политика ВК
- 2 Зачем откреплять номер мобильного телефона?
- 3 Как отвязать номер от страницы Вконтакте
- 4 Как отвязать номер от заблокированной страницы в ВК
Политика ВК
С 2013 года при регистрации аккаунта Вконтакте используется номер мобильного и в дальнейшем номер остается связанным с ней. Это обеспечивает дополнительный уровень защиты личных данных и возможность восстановить забытый пароль при помощи смс-сообщения. С помощью мобильного страница становится более персонифицированной, так как сайт рассчитан на, в большей части, население стран СНГ, где в большинстве стран сим-карты регистрируются при помощи паспортных данных.
Это обеспечивает дополнительный уровень защиты личных данных и возможность восстановить забытый пароль при помощи смс-сообщения. С помощью мобильного страница становится более персонифицированной, так как сайт рассчитан на, в большей части, население стран СНГ, где в большинстве стран сим-карты регистрируются при помощи паспортных данных.
Зачем откреплять номер мобильного телефона?
Из-за привязки к определенной странице, на один номер телефона можно зарегистрировать одну страницу. Данное правило введено для борьбы со спамом и накрутками. Также телефон может быть утерян, и это может представлять прямую угрозу для данных пользователя. Во всех этих случаях может возникнуть вопрос: как сменить номер телефона или как отвязать номер от ВК. Для этого существует несколько вариантов.
Как отвязать номер от страницы Вконтакте
Существует официальный способ, применение которого полностью безопасно и не грозит блокировкой страницы. Однако, при использовании этого варианта стоит учитывать, что процесс займет до 14 дней. Для изменения номера Вконтакте, следует:
Для изменения номера Вконтакте, следует:
- Зайти на свой аккаунт Вконтакте;
- Выбрать раздел «Настройки» – «Общее» – «Номер телефона».
- Выбрать пункт «Изменить».
- В появившемся поле указать новый мобильный.
- Появится оповещение о том, что на оба телефона отправлены смс-сообщения с кодами подтверждения.
- Вводим пароли, подтверждаем оба номера и подаем заявку. По прошествию двух недель телефон будет изменен.
Если нужно отвязать номер перед тем, как удалить аккаунт, существует эффективный и быстрый способ, который, однако, может привести к блокировке номера навсегда. Для этого можно зарегистрировать фиктивный номер в специальном стороннем сервисе, и заменить им действующий телефон для получения смс-сообщений из Вконтакте.
Также, если имеются профили, зарегистрированные при помощи электронного адреса (такой способ регистрации работал до 2013 года), попробуйте заменить на действующий или удалить номер телефона в ВК. После этого произойдет автоматическая отвязка номера от новой страницы.
Закрепление номера
Если к странице нет доступа, открепить номер можно в процессе создания нового профиля. После регистрации нового профиля на телефон придет смс-сообщение, и после введения пароля номер сразу же будет закреплен за новой страницей.
«Заморозка» страницы
Если не пугает перспектива длительной заморозки аккаунта, воспользуйтесь вариантом, основанном на баге соцсети. Он состоит в том, что при замораживании аккаунта прикрепленный к нему номер телефона сразу же открепляется. Для ускорения процесса «заморозки» необходимо произвести несложные процедуры, связанные с накруткой или распространением спама: вступить в несколько десятков групп или добавить большое количество новых друзей. После заморозки страницы, при ее обновлении видим надпись:«укажите другой номер телефона». После введения действующего телефона, система отправит на него смс с кодом проверки. Вводим пароль и система сообщает, что к этой странице прикреплен другой номер. В связи с этим система предложит отвязать номер.
После того, как будет введен новый номер, придумываем и вводим новый пароль и нажимаем на синюю кнопку «разморозить аккаунт». Таким образом, номер изменится менее чем за 24 часа.
Привязка к электронной почте
Открепить номер телефона от профиля можно и без привязки нового номера, прикрепив к нему электронную почту. Для этого :
- Выбираем графу «Оповещения» в настройках, и в ней выбираем вариант «Привязать страницу к е-mail».
- В поле заносим электронный адрес и нажимаем клавишу «Сохранить».
- После того, как будет получено письмо с подтверждением, открепляем номер телефона, перенаправив все оповещения на электронную почту.
Главное запомнить, что теперь вход на страницу будет осуществляться с использованием почты.
Как отвязать номер от заблокированной страницы в ВК
Если же после вышеуказанных способов профиль оказался заблокирован или удален, напишите сообщение специалист техподдержки Вконтакте. В ответ придет инструкция с пошаговым описанием процесса восстановления аккаунта и рекомендациями от администрации насчет того, как отвязать номер от страницы в ВК.
В ответ придет инструкция с пошаговым описанием процесса восстановления аккаунта и рекомендациями от администрации насчет того, как отвязать номер от страницы в ВК.
Алевтина Зайцева (Написано статей: 109)
Задать вопрос
Автор аналитических статей, обзоров, руководств и инструкций. Благодаря своему образованию и опыту, Алевтина — настоящий специалист в пользовании социальными сетями и умными устройствами. Просмотреть все записи автора →
Оценка статьи:
Загрузка…
Поделиться с друзьями:
Adblock
detector
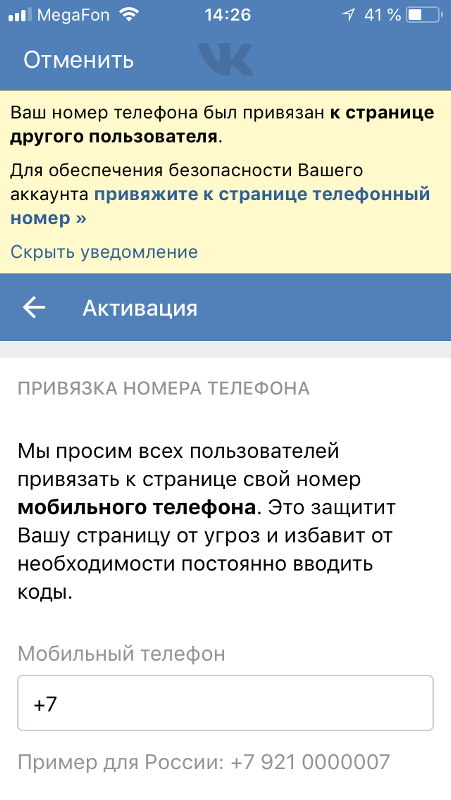
Вконтакте просит ввести номер мобильного телефона
Автор Neutronfan Просмотров 124к. Опубликовано
Уже довольно давно социальная сеть Вконтакте ввела привязку страницы пользователя к номеру мобильного телефона. Но многие по-прежнему удивляются и даже паникуют, когда, открыв данный сайт, видят такое сообщение: “Пожалуйста, привяжите Вашу страницу к номеру сотового телефона, чтобы защитить ее от злоумышленников”.
Но многие по-прежнему удивляются и даже паникуют, когда, открыв данный сайт, видят такое сообщение: “Пожалуйста, привяжите Вашу страницу к номеру сотового телефона, чтобы защитить ее от злоумышленников”.
Или такое:
“Мы зафиксировали попытку взлома Вашей страницы. Не беспокойтесь, она в безопасности. Чтобы обезопасить Вашу страницу от злоумышленников и в будущем, мы просим Вас подтвердить привязку к телефону и придумать новый сложный пароль
Правда в интернете многие на полном серьезе обсуждают теорию заговора Павла Дурова против пользователей, согласно которой сеть Вконтакте таким образом собирает их телефоны и передает в случае необходимости соответствующим органам.
Заговор это или нет, но для того чтобы снова зайти на свою страничку, ее придется привязать к номеру мобильника. К тому же это действительно удобно: если забыли свой пароль, то просто вводите номер телефона и на него сразу приходит sms с кодом. Данный код нужно ввести на сайте, и вы получаете доступ к своей странице.
Итак, с этим вроде бы все понятно. Но есть одна серьезная проблема: как только Вконтакте ввели эту привязку к номеру, почти сразу же активизировались злоумышленники, которые написали вирусы, представляющие собой поддельные (фейковые) страницы сайта Вконтакте. На них один в один (или почти один в один) повторяются сообщения, представленные на скриншотах в начале этой статьи. Данные вирусы маскируются под реальную страницу Вконтакте и просят вас ввести мобильный телефон, либо сразу отправить смс на какой-нибудь номер. Если вдруг по каким-то причинам ваш компьютер оказался заражен таким вирусом, то открыв однажды свой любимый сайт, вы можете увидеть подобные сообщения, о которых мы уже говорили выше.
[stextbox]Внимание! Настоящий сайт ВКонтакте не попросит вас отправить СМС на какой-либо номер. Ни в коем случае не отправляйте смс – это пустая трата денег![/stextbox]
[adsense1]
Самое сложное в такой ситуации – это понять: реальная перед вами страница или фейковая (поддельная). Сейчас я расскажу, как это можно сделать:
1. Смотрим адресную строку браузера
В нашем случае в адресной строке написано следующее:Но это еще не гарантия того, что у нас нет вируса.
А вот если вместо первых символов vk.com, стоит какой-то другой адрес (например, vkmvkvk.com), то это значит, что мы точно находимся на поддельном сайте. Закройте его и лечите свой компьютер от вируса.
2. Зайдите на сайт Вконтакте с другого компьютера или мобильного устройства
Если на другом компьютере вы увидите точно такое же сообщение, то значит это действительно сообщение от администрации сайта Вконтакте. Поэтому можете ввести свой номер телефона.
Поэтому можете ввести свой номер телефона.
Если же на другом компьютере или мобильном устройстве, вы без проблем сможете зайти на свою страницу и никаких сообщений не появится, значит тот компьютер, на котором вы видите подобное сообщение заражен вирусом.
[adsense2]
3. Если у вас нет возможности зайти на сайт с другого устройства, тогда проверьте системный файл hosts
Идем по следующему пути C:\Windows\System32\drivers\etc. Здесь в папке etc должны находится только следующие файлы: hosts, networks, protocol, services (все без расширения) и файл imhosts с расширением .sam (в Windows 7 также может находится файл hosts в виде календарика с расширением .ics):В этом же окне нажмите сверху слева кнопку “Упорядочить” – “Параметры папок и поиска” – вкладка “Вид”. Здесь поставьте маркер в пункте “Показывать скрытые файлы и папки”, а также уберите галки в строчках “Скрывать защищённые системные файлы” и “Скрывать расширения для зарегистрированных типов файлов”. Нажмите “Применить” – “ОК”:
Здесь поставьте маркер в пункте “Показывать скрытые файлы и папки”, а также уберите галки в строчках “Скрывать защищённые системные файлы” и “Скрывать расширения для зарегистрированных типов файлов”. Нажмите “Применить” – “ОК”:
[adsense3]
Если после этого в папке появится файл (или файлы) hosts с каким-либо расширением (например hosts.bak или hosts.txt) – знайте, это вирусные файлы и их нужно удалить. Дело в том, что настоящий файл hosts не имеет никакого расширения.
Это еще не все. Щелкните правой клавишей мыши по файлу hosts (тот, который без расширения) – выберите “Открыть”. Далее в списке программ выберите Блокнот и нажмите “ОК”:
[adsense4]
В Windows 7 незараженный файл hosts должен выглядеть вот так:
[adsense5]
Если у вас после данного текста есть еще какие-то записи, значит ваш файл hosts уже изменен вирусом. Также часто бывает, что вирусные записи расположены не сразу под основным текстом, а намного ниже. Поэтому если здесь справа есть полоса прокрутки – прокрутите ее до самого конца:
Поэтому если здесь справа есть полоса прокрутки – прокрутите ее до самого конца:
Итак, если вы убедились, что адрес в строке браузера введен верный и с системным файлом hosts тоже все в порядке, тогда можете вводить на странице Вконтакте свой номер мобильного телефона. В ответ вам придет код, который нужно ввести на сайте. После этого вы получите доступ к своей странице.
[adsense6]
Но если вдруг вместо кода придет сообщение с просьбой отправить смс на какой-либо номер, ни в коем случае этого не делайте! Вконтакте никогда не просят отправить им смс!
В этом случае на компьютере 100% имеется вирус, и от него нужно избавляться. Как это сделать я подробно описал в статье “Не могу зайти на сайт ВКонтакте, не заходит на Одноклассники, не открывается Яндекс”.
Как привязать, изменить или отвязать телефон
Благодаря привязке к мобильному телефону вы всегда сможете восстановить доступ к аккаунту. Если мошенники узнают ваш пароль, вы сможете просто изменить его, запросив код подтверждения на номер своего мобильного телефона, и сохранить учётную запись в безопасности.
Привязка игрового аккаунта к мобильному телефону — бесплатная операция.
При привязке учётной записи к мобильному телефону ваш номер будет использоваться только для отправки вам SMS с кодами. Никаких рассылок на указанный номер отправляться не будет.
Как привязать телефон
- Авторизуйтесь и перейдите в Личный кабинет. На странице Личного кабинета, в настройках безопасности аккаунта, нажмите кнопку «Привязать».
- На электронную почту, которую вы используете для входа в игру, будет отправлено письмо со ссылкой активации.
- Проверьте свой почтовый ящик и перейдите по ссылке, чтобы активировать привязку.
Что делать, если нет доступа к почте?
Что делать, если письмо не приходит?
- Введите адрес электронной почты и пароль, которые вы используете для входа в игру, чтобы подтвердить привязку.
- Укажите свою страну проживания и номер мобильного телефона, а затем нажмите Продолжить.
- Проверьте правильность введённых данных и нажмите на кнопку Получить СМС-код.

- На указанный вами номер телефона придёт SMS с кодом подтверждения. Введите полученный код в специальное поле и нажмите Активировать.
Что делать, если код не пришёл?Вы можете запросить код ещё раз, но только два раза в сутки и не ранее, чем через 15 минут после предыдущего запроса.
- Если привязка прошла успешно, вы увидите соответствующее сообщение. После этого нажмите Закрыть.
- Информация о привязке телефона также будет обновлена в Личном кабинете.
Как изменить телефон
Если вы привязали учётную запись к номеру телефона, то всегда можете его изменить. Однако если вы не привязывали учётную запись к своему номеру телефона и доступ к ней получил злоумышленник, то он может совершить привязку раньше вас. Что делать, если аккаунт взломали и нет доступа к учётной записи?
Мы настоятельно рекомендуем привязывать аккаунт к номеру телефона, чтобы в случае потери контроля над учётной записью вы смогли самостоятельно восстановить доступ к ней.
Изменить номер телефона можно не раньше, чем через месяц после привязки, и не чаще, чем трижды за полгода. Если у вас нет доступа к своему номеру мобильного телефона, отправьте запрос оператору мобильной связи для восстановления номера.
- Авторизуйтесь и перейдите в Личный кабинет. На странице Личного кабинета, в настройках безопасности аккаунта, нажмите на слово «Настройки» справа от текущего телефонного номера. Затем нажмите кнопку «Изменить».
- Выберите свою страну, введите новый номер телефона и нажмите Продолжить.
- Проверьте правильность введённых данных и нажмите на кнопку Получить СМС-код.
- На новый номер придёт SMS с кодом активации. Введите полученный код в соответствующее поле и нажмите Активировать.
Что делать, если код не пришёл? - На номер, к которому привязан аккаунт в данный момент, придёт код подтверждения. Введите полученный код в соответствующее поле и нажмите Подтвердить.
Что делать, если код не приходит? - Отобразится сообщение о том, что номер успешно изменён. Завершите изменение номера, нажав ОK.
- Информация также будет обновлена в Личном кабинете.
Как отвязать телефон (доступ к номеру есть)
Если вы привязали аккаунт к номеру телефона, то всегда можете удалить привязку.
Однако, если вы не привязывали учётную запись к своему номеру телефона и доступ к ней получил злоумышленник, то он может совершить привязку раньше вас.
Что делать, если аккаунт взломали и нет доступа к учётной записи?
Мы настоятельно рекомендуем привязывать аккаунт к номеру телефона, чтобы в случае потери контроля над учётной записью вы смогли самостоятельно восстановить доступ к ней.
- Авторизуйтесь на официальном портале любой из наших игр или на сайте Wargaming.net и перейдите в Личный кабинет.
- На странице Личного кабинета, в настройках безопасности аккаунта, нажмите слово «Настройки» справа от текущего телефонного номера, а затем на ссылку «Удалить привязку».
- Чтобы подтвердить своё желание удалить привязку, нажмите Продолжить.
- На номер, к которому привязан аккаунт, придёт SMS с кодом активации. Введите полученный код в соответствующее поле и нажмите Удалить.
Что делать, если код не пришёл? - Отобразится сообщение о том, что удаление привязки прошло успешно. Завершите удаление привязки, нажав ОK.
- Если вы удалили привязку, то совершить её заново можно не раньше, чем через месяц после предыдущей привязки и не чаще трёх раз в полгода. Если у вас нет доступа к своему номеру телефона, отправьте запрос оператору мобильной связи для восстановления номера.
Как отвязать телефон (доступа к номеру нет)
Если вы привязали аккаунт к номеру телефона, то всегда можете удалить привязку. Но если аккаунт не привязан к телефону и доступ к нему получил злоумышленник, то он может совершить привязку раньше вас. Что делать, если аккаунт взломали и нет доступа к учётной записи?
- Войдите в Личный кабинет с помощью электронной почты и пароля.
 В настройках безопасности аккаунта, нажмите слово «Настройки» справа от текущего телефонного номера, а затем на ссылку «Удалить привязку».
В настройках безопасности аккаунта, нажмите слово «Настройки» справа от текущего телефонного номера, а затем на ссылку «Удалить привязку». - Чтобы подтвердить удаление, нажмите на ссылку «Нет доступа к телефону».
- Введите номер телефона, к которому привязан аккаунт, и нажмите Продолжить.
- Чтобы подтвердить удаление привязки, перейдите по ссылке из письма, которое придёт на вашу почту. Ссылка действительна 24 часа.
- В Личном кабинете отобразится сообщение, что ваш запрос принят. В нём вы также найдёте дату, когда привязка будет удалена, и ссылку для отмены удаления.
- Вам также придёт уведомление на сайте и в клиенте игры. Такие уведомления будут приходить вам раз в неделю, пока привязка не будет удалена.
Отмена удаления привязки
- Войдите в Личный кабинет с помощью электронной почты и пароля. Нажмите на ссылку «Отменить операцию» в инфоокне сверху.
- Укажите причину отмены и нажмите Отменить отвязку.
 Готово!
Готово!
Как узнать номер в вк если он скрыт
Главная » Вк » Как узнать номер в вк если он скрыт
Определить скрытый номер на страничке ВКонтакте и Facebook
Личные данные зарегистрированного пользователя социальных сетей «ВКонтакте» и «Facebook» можно просмотреть. Для этого достаточно знать только
E-Mail человека, об этом написал в своем блоге руководитель из отдела аналитики по защищенности компании Positive Technologies, Дмитрий, так зовут этого человека, нашел этот «баг» в системе защиты данных пользователя в социальных сетях.
В 2012 году сайт социальной сети «ВКонтакте» начал просить у своих пользователей, для регистрации на сайте вводить свой номер телефона для того, чтобы защитить как можно лучше свою страницу.
При запросе который посылается к функции восстановления утерянного пароля по СМС, страница аутентификации просить вписать номер телефона который привязан к электронной почте — без последних четырех цифр.
В Facebook не нужно даже знать электронный адрес пользователя. Для получения номера телефона нужно просто ввести Имя или логин человека и вам будет показан номер телефона на который зарегистрирована страница.
Для получения номера телефона нужно просто ввести Имя или логин человека и вам будет показан номер телефона на который зарегистрирована страница.
Таким образом получается, что для ВКонтакте нужно знать почтовый ящик пользователя, чтобы узнать его личный номер а для Facebook нужно просто ввести Имя или логин человека.
Определить скрытый номер на страничке ВКонтакте и Facebook, 3.7 out of 5 based on 3 ratingsКак узнать, к какому номеру привязана страница в ВК?
Несколько лет назад всех пользователей ВКонтакте «заставили» привязать свои странички к телефонным номерам. Многим пользователям это не понравилось, поскольку они не смогли войти на сайт, но, как показало время, это была правильная позиция администрации. Теперь аккаунты крадут заметно реже, а пользователям в случае возникновения каких-либо трудностей проще восстановить доступ к своей страничке.
Поскольку теперь каждая страничка привязана к номеру телефона, некоторые пользователи задаются вопросом — можно ли этот самый номер узнать? Отвечаем — нет.
Если вы хотите узнать номер, к которому привязана чужая страница, то вы можете спросить у пользователя этот номер. В редких случаях номер можно найти на странице человека, хотя это не говорит о том, что аккаунт привязан именно к указанному номеру. И даже в том случае, если по каким-либо причинам у вас окажется доступ к чужому аккаунту, вы все равно не сможете узнать заветные цифры — система их скрывает даже от владельца страницы, показывая лишь последние две цифры!
Да-да, если вы вдруг забыли номер телефона, к которому привязана ваша страница, максимум, на что можно рассчитывать, это на две последние цифры, которые, возможно, вам ничего не скажут.
Что бы найти эти цифры, зайдите в раздел «Мои настройки», вкладка «Общее».
Затем опустите страничку до подраздела «Номер вашего телефона».
Вы увидите две последние цифры (в нашем случае мы их закрасили), а остальные цифры будут скрыты под звездочками. Это сделано для безопасности вашей же страницы.
Как узнать, к какому номеру привязана страница в ВК
Раньше регистрация в данной сети была упрощенной, но на текущий момент для регистрации обязательно указание номера мобильного телефона. Это действие является необходимым, чтобы лучше идентифицировать пользователя и обеспечить большую безопасность в социальной сети.
Это действие является необходимым, чтобы лучше идентифицировать пользователя и обеспечить большую безопасность в социальной сети.
Привязка страницы к личному номеру телефона не только повышает общий уровень безопасности в социальной сети, но и частный. Только у пользователя есть доступ к своему номеру телефона, поэтому все важные операции будут им отслеживаться и подтверждаться через данный номер телефона.
Но что делать в ситуации, когда человек забыл номер телефона, к которому привязывал страницу? И возможно ли другим людям узнать, к какому номеру привязана чужая страница? Ответы на эти вопросы вы можете обнаружить ниже.
Как узнать, к какому номеру привязана страница в ВКонтакте?
Для того чтобы узнать, к какому номеру привязана страница вконтакте, вы можете воспользоваться следующими способами:
- Спросить непосредственно у пользователя, которому принадлежит номер.
- Посмотреть на личной странице пользователя. Однако, в данном случае вновь не помешает уточнить у самого пользователя: это ли телефонный номер, к которому привязана страница, либо он был указан в качестве дополнительного?
К сожалению, для повышения уровня безопасности, политикой администрации были ликвидированы все другие способы узнать мобильный номер, к которому относится страница человека в социальной сети.
У некоторых может возникнуть вопрос: эти действия актуальны для восстановления страницы с компьютера, но как узнать к какому номеру привязана страница в вк с телефона? Каких-то дополнительных способов при использовании телефона не появляется, способы будут аналогичны тем, что приведены выше.
Будьте внимательны с номером телефона, который вы используете при регистрации, запишите его. Потому что в последующем вы не сможете узнать его даже через настройки страницы, потому что информация о номере телефона будет представлена в зашифрованном виде. В расшифрованном виде вам будет доступна лишь такая информация: «+7» и две последние цифры вашего номера (рис. 1).
Теперь вы понимаете, что выяснить телефонный номер, к которому выполнялась привязка, достаточно сложно. Данные сложности могут быть неприятными, но они того стоят, потому что значительно повышают степень безопасности для пользователей. Для того чтобы не испытывать негатива от этих сложностей, старайтесь запоминать либо записывать телефонный номер, указанный при регистрации.
Как узнать ID страницы Вконтакте
Доброго дня всем тем, кто читает блог cho-cho! Сегодня мы поговорим о том, как узнать ID любой страницы ВКонтакте.
Для тех, кто не в курсе:
ID страницы Вконтакте — это уникальный идентификационный номер любого аккаунта, зарегистрированного в соцсети, а также пабликов, групп, постов и даже фотографий с видеозаписями в ней.
«Айди» всегда присваивается при создании чего-либо ВКонтакте. Изменить его нельзя — разве что сменить адрес страницы на «человекопонятный», например, vk.com/smeshnye.kotiki. Однако и в этом случае ID страницы не меняется, он лишь делается скрытым.
Вот как выглядят различные идентификаторы Вконтакте:
- профиль пользователя — vk.com/id78563931
- паблик — vk.com/public13675315
- группа — vk.com/club2505625
- встреча — vk.com/event4552624
Где цифры в конце — это и есть ID.
Как узнать ID любого пользователя Вконтакте
Самый простой способ узнать id у друга — это открыть нужный аккаунт в браузере и посмотреть на адресную строку, точнее, на цифры, которые идут после https://vk.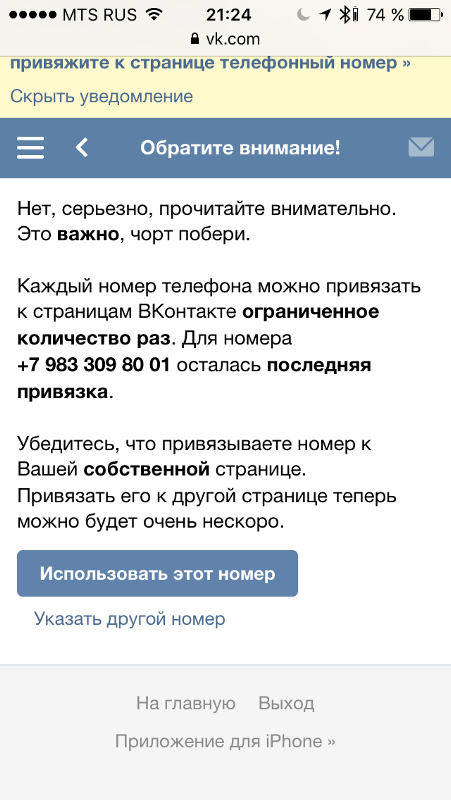 com/id…
com/id…
Однако бывает, что ID оказывается скрытым, потому что человек успел поменять в настройках адрес своей страницы на удобный ему короткий — к примеру, «meowmeow1» вместо обычных цифр.
Что делать в таком случае?
Вариант первый: приложение LinkApp
Найти его можно по ссылке vk.com/linkapp. Введя туда ссылку на нужного вам пользователя, например, vk.com/meowmeow1, нажмите «Узнать» и получите его ID.
Вариант второй: аватарка
Щелкните по аватарке профиля нужного человека и вновь обратите внимание на адресную строку. Цифры, которые находятся между словом «photo» и символом «_», и есть искомый номер.
Вариант третий: стена
Если вы можете смотреть записи на стене юзера и они там имеются, щелкните по дате любого поста и между словом «wall» и «_» будет нужный вам идентификатор пользователя.
Вариант четвертый: сообщения
Если вы нажмете «отправить сообщение» нужному вам человек, то при открытии диалога с пользователем вид будет такой: https://vk. com/im?sel=295772019 или, например, такой:
com/im?sel=295772019 или, например, такой:
Последние цифры и есть «айди».
Как узнать свой ID в Вк
Сработают все вышеперечисленные способы, и к ним добавляется еще один.
- зайдите в меню «Настройки» вашей страницы;
- там перейдите на вкладку «Общее»;
- прокрутите страницу вниз до пункта «Адрес страницы»;
- если у вас уже есть короткий адрес, просто щелкните по нему — вам предложат его изменить, а заодно покажут строку «Номер страницы — такой-то».
Как узнать ID группы, паблика или встречи Вконтакте
Здесь вы также можете сразу узнать «айди» из адресной строки, или снова наткнуться на «красивый» адрес — вместо vk.com/club106559582, где последние цифры — искомый номер, увидите vk.com/ero_blog_ru (ссылка откроется в обоих случаях).
Однако способы поиска номера, описанные выше, также работают и для любых ВК-сообществ. Вы можете воспользоваться приложением, аватаркой сообщества, постами на его стене.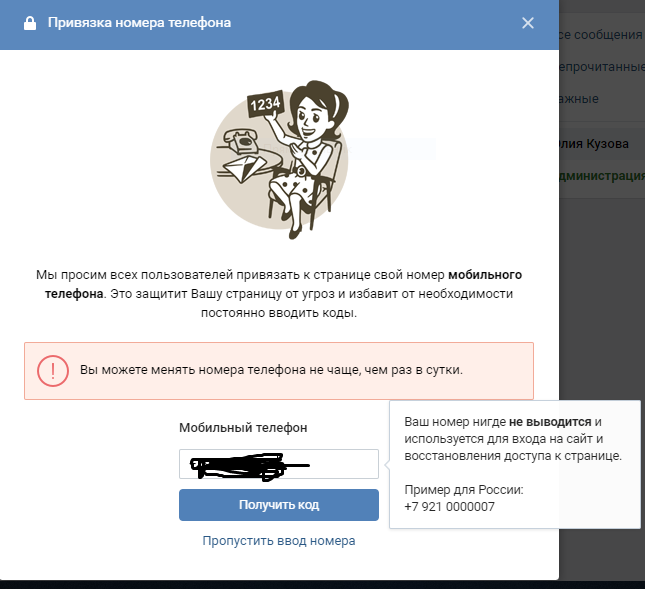
Как узнать ID фотографий, видеозаписей, постов на стене ВК
Для того, чтобы узнать их номер, нужно открыть видео/фото/пост, просто щелкнув на них, и посмотреть на адрес в браузере.
Например, у фотографии https://vk.com/photo279984181_352425030 после слова «photo» идет ID пользователя, который выложил этот самый контент в социальную сеть, а вот цифры, которые идут после символа «_» (352425030) — и есть уникальный номер.
В случае, когда после слова «photo» стоит дефис, как тут https://new.vk.com/do_iposle?z=photo-57950162_400116475%2Fwall-57950162_39678, это означает, что картинка принадлежит и выложена неким ВК-сообществом с айди 57950162, и уникальный номер фотографии — 400116475.
Та же схема относится ко всем видеороликам и постам ВК.
Как узнать номер телефона, почту, логин, IP по ID Вконтакте
Вот чего-чего, а такие данные вы никак не вычислите и не узнаете по «айди» пользователя социальной сети. Зато можете найти человека по определенному номеру телефона, об этом мы писали здесь.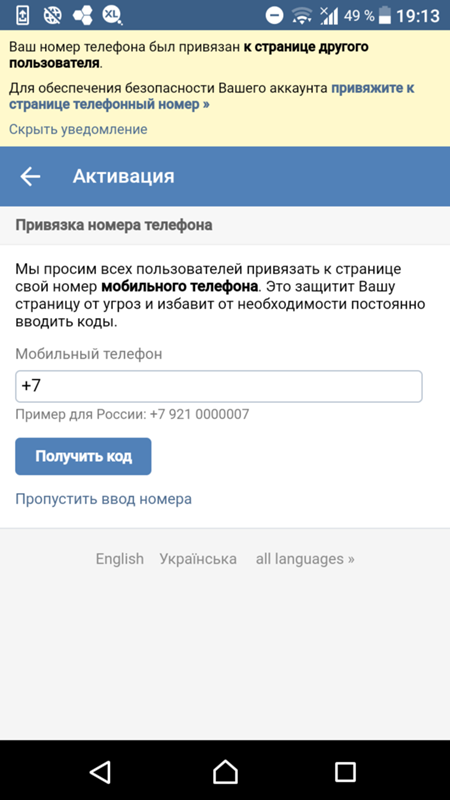
Вот и все, теперь вы свободно можете выяснить уникальный номер любого человека, сообщества или ВК-контента!
Обязательно к ознакомлению:— руководство по Vulkan
До сих пор мы использовали push-константы для загрузки данных из CPU в GPU. Хотя push-константы полезны, у них есть много ограничений. Например, вы не можете загрузить массив, вы не можете указать на буфер и не можете использовать с ними текстуры. Для этого вам нужно использовать наборы дескрипторов, которые являются основным способом подключения данных ЦП к графическому процессору.
Для этого вам нужно использовать наборы дескрипторов, которые являются основным способом подключения данных ЦП к графическому процессору.
Наборы дескрипторов могут быть очень сложными в использовании по сравнению со всеми другими подобными альтернативами, используемыми в других графических API. По этой причине мы начнем с них очень просто и будем использовать их только для буферов, и делать с ними больше вещей по мере продолжения главы. Текстурная сторона наборов дескрипторов будет рассмотрена в главе 5.
Ментальная модель
Думайте об одном дескрипторе как о дескрипторе или указателе на ресурс. Этот ресурс является буфером или изображением, а также содержит другую информацию, например размер буфера или тип сэмплера, если он предназначен для изображения. VkDescriptorSet — это набор тех указателей, которые связаны друг с другом. Vulkan не позволяет связывать отдельные ресурсы в шейдерах. Они должны быть сгруппированы в наборы. Если вы по-прежнему настаиваете на возможности связывания их по отдельности, вам потребуется набор дескрипторов для каждого ресурса. Это очень неэффективно и не будет работать на многих аппаратных средствах. Если вы посмотрите на этот https://vulkan.gpuinfo.org/displaydevicelimit.php?name=maxBoundDescriptorSets&platform=windows, вы увидите, что некоторые устройства позволяют привязывать только до 4 наборов дескрипторов к данному конвейеру на ПК. Из-за этого мы действительно можем использовать до 4 наборов дескрипторов в наших конвейерах, если мы хотим, чтобы движок работал на интегрированных графических процессорах Intel. Распространенный и эффективный способ справиться с этим ограничением в 4 дескриптора — сгруппировать их по частоте связывания.
Это очень неэффективно и не будет работать на многих аппаратных средствах. Если вы посмотрите на этот https://vulkan.gpuinfo.org/displaydevicelimit.php?name=maxBoundDescriptorSets&platform=windows, вы увидите, что некоторые устройства позволяют привязывать только до 4 наборов дескрипторов к данному конвейеру на ПК. Из-за этого мы действительно можем использовать до 4 наборов дескрипторов в наших конвейерах, если мы хотим, чтобы движок работал на интегрированных графических процессорах Intel. Распространенный и эффективный способ справиться с этим ограничением в 4 дескриптора — сгруппировать их по частоте связывания.
Набор дескрипторов с номером 0 будет использоваться для глобальных ресурсов механизма и привязываться один раз к кадру. Набор дескрипторов номер 1 будет использоваться для ресурсов на проход и привязываться один раз за проход. Набор дескрипторов номер 2 будет использоваться для материальных ресурсов, а номер 3 будет использоваться для ресурсов каждого объекта. Таким образом, внутренние циклы рендеринга будут связывать только наборы дескрипторов 2 и 3, и производительность будет высокой.
Распределение дескрипторов
Наборы дескрипторов должны выделяться непосредственно ядром из Вкдескрипторпул . Распределение набора дескрипторов обычно размещается в разделе видеопамяти графического процессора. Как только набор дескрипторов выделен, вам нужно написать его, чтобы он указывал на ваши буферы/текстуры. После привязки набора дескрипторов и использования его в функции vkCmdDraw() вы больше не сможете изменять его, если не укажете флаг VK_DESCRIPTOR_POOL_CREATE_UPDATE_AFTER_BIND_BIT . Когда выделяется пул дескрипторов, вы должны сообщить драйверу, сколько наборов дескрипторов и какое количество ресурсов вы будете использовать. Обычно по умолчанию используются большие числа, например 1000 дескрипторов, и когда в пуле дескрипторов заканчивается место, выделение нового дескриптора завершается с ошибкой. Затем вы можете просто пойти и создать новый пул для хранения большего количества дескрипторов.
Выделение наборов дескрипторов может быть очень дешевым, если вы явно запретите освобождение отдельных наборов, установив флаг VK_DESCRIPTOR_POOL_CREATE_FREE_DESCRIPTOR_SET_BIT . Используя этот флаг, драйвер может заставить пул дескрипторов использовать очень дешевый линейный распределитель. Если вы выделяете наборы дескрипторов для каждого кадра, вы должны использовать это, а затем сбрасывать весь пул вместо отдельных наборов дескрипторов. Для ваших глобальных наборов дескрипторов можно выделить их один раз и повторно использовать от кадра к кадру. Это то, что мы будем делать в учебнике, так как он также заканчивается более простым кодом.
Используя этот флаг, драйвер может заставить пул дескрипторов использовать очень дешевый линейный распределитель. Если вы выделяете наборы дескрипторов для каждого кадра, вы должны использовать это, а затем сбрасывать весь пул вместо отдельных наборов дескрипторов. Для ваших глобальных наборов дескрипторов можно выделить их один раз и повторно использовать от кадра к кадру. Это то, что мы будем делать в учебнике, так как он также заканчивается более простым кодом.
Обычный метод, используемый в производственных движках, состоит в том, чтобы иметь набор пулов дескрипторов для каждого кадра. После неудачного выделения дескриптора вы создаете новый пул и добавляете его в список. Когда фрейм отправлен и вы ждете на его заборе, вы сбрасываете все эти пулы дескрипторов.
Написание дескрипторов.
Недавно выделенный набор дескрипторов — это всего лишь часть памяти графического процессора, вам нужно сделать так, чтобы он указывал на ваши буферы. Для этого вы используете vkUpdateDescriptorSets() , который принимает массив из VkWriteDescriptorSet для каждого ресурса, на который указывает набор дескрипторов. Если вы использовали флаг «Обновить после привязки», можно использовать наборы дескрипторов, связывать их в буферах команд и обновлять прямо перед отправкой буфера команд. В основном это нишевый вариант использования, и он не используется повсеместно. Вы можете обновить набор дескрипторов только перед его первой привязкой, если вы не используете этот флаг, и в этом случае вы можете обновить его только перед отправкой командного буфера в очередь. Когда используется набор дескрипторов, он неизменяем, и попытка его обновления вызовет ошибки. Слои проверки улавливают это. Чтобы иметь возможность снова обновить наборы дескрипторов, вам нужно дождаться завершения выполнения команды.
Если вы использовали флаг «Обновить после привязки», можно использовать наборы дескрипторов, связывать их в буферах команд и обновлять прямо перед отправкой буфера команд. В основном это нишевый вариант использования, и он не используется повсеместно. Вы можете обновить набор дескрипторов только перед его первой привязкой, если вы не используете этот флаг, и в этом случае вы можете обновить его только перед отправкой командного буфера в очередь. Когда используется набор дескрипторов, он неизменяем, и попытка его обновления вызовет ошибки. Слои проверки улавливают это. Чтобы иметь возможность снова обновить наборы дескрипторов, вам нужно дождаться завершения выполнения команды.
Дескрипторы привязки
Наборы дескрипторов привязываются к определенным «слотам» в конвейере Vulkan. При создании конвейера необходимо указать макеты для каждого из наборов дескрипторов, которые можно привязать к конвейеру. Обычно это делается автоматически, генерируя отражение в шейдере. Мы будем делать это вручную, чтобы показать, как это делается. После привязки конвейера в командном буфере конвейер имеет слоты для различных наборов дескрипторов, и затем вы можете привязать набор к каждому из слотов. Если набор дескрипторов не соответствует слоту, будут ошибки. Если вы привязываете набор дескрипторов к слоту 0, а затем переключаете конвейеры, связывая другой, набор дескрипторов останется привязанным, ЕСЛИ слот в новом конвейере тот же. Если слот не совсем тот же, то слот будет «непривязанным», и вам нужно будет привязать его снова. Например, предположим, что у нас есть 2 конвейера, один из которых имеет набор дескрипторов 0, который привязывается к буферу, и набор дескрипторов 1, который привязывается к 4 изображениям. Затем другой конвейер имеет набор дескрипторов 0, который привязывается к буферу (такой же, как тот же слот в другом конвейере), но в наборе дескрипторов 1 он имеет набор дескрипторов, который привязывается к 3 изображениям, а не к 4. Если вы привязываете второй конвейер , набор дескрипторов 0 останется связанным, но дескриптор 1 будет несвязанным, поскольку он больше не соответствует.
После привязки конвейера в командном буфере конвейер имеет слоты для различных наборов дескрипторов, и затем вы можете привязать набор к каждому из слотов. Если набор дескрипторов не соответствует слоту, будут ошибки. Если вы привязываете набор дескрипторов к слоту 0, а затем переключаете конвейеры, связывая другой, набор дескрипторов останется привязанным, ЕСЛИ слот в новом конвейере тот же. Если слот не совсем тот же, то слот будет «непривязанным», и вам нужно будет привязать его снова. Например, предположим, что у нас есть 2 конвейера, один из которых имеет набор дескрипторов 0, который привязывается к буферу, и набор дескрипторов 1, который привязывается к 4 изображениям. Затем другой конвейер имеет набор дескрипторов 0, который привязывается к буферу (такой же, как тот же слот в другом конвейере), но в наборе дескрипторов 1 он имеет набор дескрипторов, который привязывается к 3 изображениям, а не к 4. Если вы привязываете второй конвейер , набор дескрипторов 0 останется связанным, но дескриптор 1 будет несвязанным, поскольку он больше не соответствует. Вот почему мы назначаем частоту слотам дескриптора, чтобы свести к минимуму привязку.
Вот почему мы назначаем частоту слотам дескриптора, чтобы свести к минимуму привязку.
Макет набора дескрипторов.
Используется как в конвейерах, так и при размещении дескрипторов. VkDescriptorSetLayout — это форма дескриптора. Например, возможным макетом будет такой, где он связывает 2 буфера и 1 изображение. При создании конвейеров или размещении самих наборов дескрипторов вы должны использовать макет. В этом руководстве мы будем повторно использовать объект макета для всего, но это не обязательно. Макеты наборов дескрипторов могут быть совместимы, если они одинаковы, даже если они созданы в двух разных местах.
Униформ-буферы
Дескриптор устанавливает точку в буферах, но мы этого не объясняли. Прямо сейчас мы создаем буферы графического процессора, содержащие данные вершин, но вы также можете создавать буферы, содержащие произвольные данные для использования в шейдерах. Для этого типа данных обычно используются унифицированные буферы. Они небольшого размера (до нескольких килобайт), но очень быстро читаются, поэтому идеально подходят для параметров шейдера. Создав унифицированный буфер и записав в него из ЦП, вы можете отправлять данные в ГП гораздо более эффективным способом, чем с помощью push-констант. Мы будем использовать его для информации о камере. Можно иметь несколько наборов дескрипторов, указывающих на один юниформ-буфер, а также можно иметь большой юниформ-буфер, и тогда каждый набор дескрипторов указывает на раздел буфера. Шейдер не заметит разницы.
Они небольшого размера (до нескольких килобайт), но очень быстро читаются, поэтому идеально подходят для параметров шейдера. Создав унифицированный буфер и записав в него из ЦП, вы можете отправлять данные в ГП гораздо более эффективным способом, чем с помощью push-констант. Мы будем использовать его для информации о камере. Можно иметь несколько наборов дескрипторов, указывающих на один юниформ-буфер, а также можно иметь большой юниформ-буфер, и тогда каждый набор дескрипторов указывает на раздел буфера. Шейдер не заметит разницы.
Далее: Настройка наборов дескрипторов
Структура комплекса транспозаза TnsB-ДНК типа V-K CRISPR-ассоциированный транспозон
Введение
Открытие адаптивной прокариотической иммунной системы под названием C lusted 90 Regular межпространственные S hort P алиндромные R повторы (CRISPR), в которых повторы связаны с Cas (CRISPR-ассоциированными) белками, произвели революцию в науках о жизни. Системы CRISPR-Cas представляют собой весьма разнообразные комплексы рибонуклеопротеинов (РНП) с разным эволюционным происхождением. Они делятся на два класса, класс 1 и класс 2, первый из которых включает многосубъединичный эффекторный комплекс, а второй — единственный белковый эффектор 9.0072 1 . Два класса далее делятся на шесть типов (I-VI) в зависимости от идентичности нуклеазного модуля и множество подтипов в зависимости от того, какие другие белки Cas присутствуют в других функциональных модулях. В частности, члены класса 2 привлекли большое внимание, поскольку они были разработаны в универсальные РНК-управляемые нуклеазы для РНК-управляемого редактирования генома, что радикально изменило науки о жизни, позволив манипулировать геномом в живых организмах 2 .
Системы CRISPR-Cas представляют собой весьма разнообразные комплексы рибонуклеопротеинов (РНП) с разным эволюционным происхождением. Они делятся на два класса, класс 1 и класс 2, первый из которых включает многосубъединичный эффекторный комплекс, а второй — единственный белковый эффектор 9.0072 1 . Два класса далее делятся на шесть типов (I-VI) в зависимости от идентичности нуклеазного модуля и множество подтипов в зависимости от того, какие другие белки Cas присутствуют в других функциональных модулях. В частности, члены класса 2 привлекли большое внимание, поскольку они были разработаны в универсальные РНК-управляемые нуклеазы для РНК-управляемого редактирования генома, что радикально изменило науки о жизни, позволив манипулировать геномом в живых организмах 2 .
Недавно было обнаружено, что несколько механизмов CRISPR-Cas связаны с Tn7-подобными системами транспозонов типов I, IV и V. Эти системы CAST 3,4 являются продуктом эволюционного процесса, в ходе которого Tn 7 -подобные транспозоны рекрутировали систему CRISPR-Cas для мобилизации транспозонов.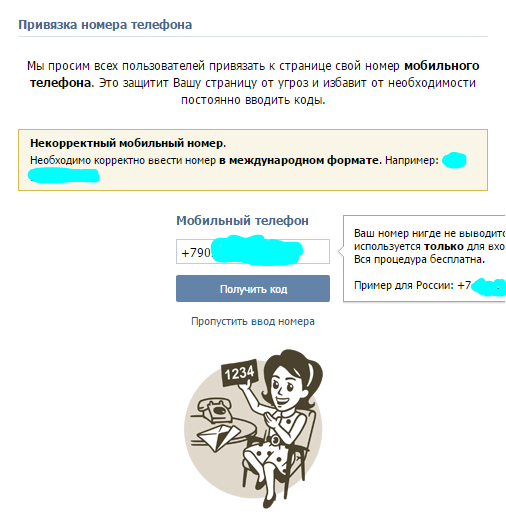 Эти комплексы не разрушают свою ДНК-мишень и действуют исключительно на прокариотах. Они вставляют большие ДНК-грузы (10–30 т.п.н.) в определенные участки генома без необходимости гомологически направленной репарации 4,5,6,7 , сочетая точность выбора сайта CRISPR-Cas с интеграционными свойствами транспозонов 8 . Поэтому считается, что CAST представляют собой очень многообещающую систему для разработки инструментов редактирования генов следующего поколения.
Эти комплексы не разрушают свою ДНК-мишень и действуют исключительно на прокариотах. Они вставляют большие ДНК-грузы (10–30 т.п.н.) в определенные участки генома без необходимости гомологически направленной репарации 4,5,6,7 , сочетая точность выбора сайта CRISPR-Cas с интеграционными свойствами транспозонов 8 . Поэтому считается, что CAST представляют собой очень многообещающую систему для разработки инструментов редактирования генов следующего поколения.
CAST I-F, I-B и V-K подтипы, из Vibrio cholerae (vc), Anabaena variabilis (av) и Scytonema hofmannii (sh) соответственно, были обнаружены первыми 3,4 , но недавние биоинформационные поиски в метагеномных базах данных значительно расширили известный репертуар CAST до более чем 1000 неповторяющихся подсистем, представляющих Типы I, IV и V 6 . На сегодняшний день все известные CAST происходят от Tn 7 -подобных транспозонов и включают соответствующие crРНК и Cas генов, необходимых для отбора мишеней 6,7 , и основной механизм транспозиции в Tn 7 -подобном локусе транспозона. Сюда входят гены TnsB, TnsC, TniQ (гомолог E. coli TnsD) и, в некоторых случаях, гены TnsA. По аналогии с системами транспозонов Tn7, белки CAST Tn 7 , как полагают, собираются в нуклеопротеиновый комплекс перед интеграцией, который включает TnsA (в типах I и IV), TnsB, TnsC и TniQ, чтобы регулировать транспозицию в инсерцию. сайт. TnsA — эндонуклеаза, расщепляющая 5′-концы транспозона 9.0072 9 и взаимодействует с TnsB, TnsC и ДНК 9,10,11 . TnsB является рекомбиназой и катализирует расщепление 3′-концов транспозона. В канонической системе Tn7 взаимодействие TnsA и TnsB необходимо для активации катализа 12 . TnsC является частью семейства AAA+ ATPase и направляет TnsA/TnsB к сайту вставки 11,13 . Считается, что взаимодействие TniQ с ДНК-мишенью, связанной комплексом CRISPR-Cas, создает искажение в структуре ДНК, позволяя TnsC распознавать как TniQ, так и ДНК 9.0072 14 и приводит к вставке транспозона в место прикрепления.
Сюда входят гены TnsB, TnsC, TniQ (гомолог E. coli TnsD) и, в некоторых случаях, гены TnsA. По аналогии с системами транспозонов Tn7, белки CAST Tn 7 , как полагают, собираются в нуклеопротеиновый комплекс перед интеграцией, который включает TnsA (в типах I и IV), TnsB, TnsC и TniQ, чтобы регулировать транспозицию в инсерцию. сайт. TnsA — эндонуклеаза, расщепляющая 5′-концы транспозона 9.0072 9 и взаимодействует с TnsB, TnsC и ДНК 9,10,11 . TnsB является рекомбиназой и катализирует расщепление 3′-концов транспозона. В канонической системе Tn7 взаимодействие TnsA и TnsB необходимо для активации катализа 12 . TnsC является частью семейства AAA+ ATPase и направляет TnsA/TnsB к сайту вставки 11,13 . Считается, что взаимодействие TniQ с ДНК-мишенью, связанной комплексом CRISPR-Cas, создает искажение в структуре ДНК, позволяя TnsC распознавать как TniQ, так и ДНК 9.0072 14 и приводит к вставке транспозона в место прикрепления. Однако системы CAST типа V-K отличаются от Tn7 из-за отсутствия TnsA в их локусах. Поскольку CAST типа V-K не содержит какого-либо другого белка для замены эндонуклеазной активности TnsA, его транспозиция продуцирует коинтеграты, которые необходимо расщепить 15,16 . Разрешение коинтегратов, по-видимому, осуществляется посредством RecA-зависимой рекомбинации, как и в случае других транспозонов, лишенных фермента с 5′-эндонуклеазной активностью. Тем не менее, транспозоны из семейства Tn5053, которые также лишены TnsA, но содержат гомологи для TnsB, TnsC и TniQ, могут разрешать коинтеграты с использованием кодируемой транспозоном резольвазы TniR. Может ли резольваза, кодируемая хозяином, обеспечивать эту активность в типе V-K CAST, еще предстоит определить.
Однако системы CAST типа V-K отличаются от Tn7 из-за отсутствия TnsA в их локусах. Поскольку CAST типа V-K не содержит какого-либо другого белка для замены эндонуклеазной активности TnsA, его транспозиция продуцирует коинтеграты, которые необходимо расщепить 15,16 . Разрешение коинтегратов, по-видимому, осуществляется посредством RecA-зависимой рекомбинации, как и в случае других транспозонов, лишенных фермента с 5′-эндонуклеазной активностью. Тем не менее, транспозоны из семейства Tn5053, которые также лишены TnsA, но содержат гомологи для TnsB, TnsC и TniQ, могут разрешать коинтеграты с использованием кодируемой транспозоном резольвазы TniR. Может ли резольваза, кодируемая хозяином, обеспечивать эту активность в типе V-K CAST, еще предстоит определить.
Транспозазы TnsB принадлежат к суперсемейству ретровирусных интеграз с каталитическим доменом рибонуклеазы H и мотивом активного сайта DDE. TnsB связывается с левым и правым концами транспозона и катализирует их расщепление с образованием свободных 3′-гидроксильных групп, которые позже используются в нуклеофильной атаке ниже целевой последовательности 17 . Наконец, он выполняет реакцию переноса цепи, которая приводит к вставке груза ДНК в целевой сайт.
Наконец, он выполняет реакцию переноса цепи, которая приводит к вставке груза ДНК в целевой сайт.
Транспозаза shTnsB имеет гомологию с другими членами семейства интеграз DDE, такими как E. coli TnsB, MuA и Tn5053 (дополнительный рисунок 1). Структура TnsB E. coli (ec) в комплексе с концом транспозона предоставила новые доказательства, объясняющие различия в распознавании левого и правого концов элемента 18 . Однако идентичность последовательности ecTnsB с shTnsB низкая, что затрудняет понимание деталей механизма интеграции CAST и других систем транспозонов, содержащих интегразы семейства DDE.
Чтобы понять, как shTnsB способствует РНК-управляемой транспозиции в системе CAST V-K, мы определяем структуру комплекса shTnsB-ДНК, захваченного после реакции переноса цепи, с помощью крио-ЭМ с одной частицей при разрешении 2,46 Å (рис. 1). Структура показывает запутанную архитектуру белка shTnsB вокруг ДНК, образующую псевдосимметричную сборку, в которой четыре субъединицы shTnsB могут быть сгруппированы в двух разных конформациях. ДНК в каталитических карманах DDE резко изгибается после реакции переноса цепи. Сайт-направленный мутагенез и анализы транспозиции in vivo выявили важную роль в транспозиции ключевых ДНК-связывающих остатков. Комплекс реакции переноса нити shTnsB открывает новые пути к пониманию управляемой РНК транспозиции в системах CAST.
ДНК в каталитических карманах DDE резко изгибается после реакции переноса цепи. Сайт-направленный мутагенез и анализы транспозиции in vivo выявили важную роль в транспозиции ключевых ДНК-связывающих остатков. Комплекс реакции переноса нити shTnsB открывает новые пути к пониманию управляемой РНК транспозиции в системах CAST.
a Общая схема реакции интеграции транспозаз семейства TnsB. Активный центр DDE катализирует атаку H 2 O на концах транспозона. Прямая атака 3’ОН груза ДНК на сайт ДНК-мишени приводит к образованию комплекса переноса цепи (STC). Промежуточный продукт Шапиро разрешается репликацией, производящей дупликацию сайта-мишени и коинтеграцию 25 , который может быть разрешен с помощью RecA или резолвазы. b Архитектура белка shTnsB. c Карта плотности крио-ЭМ с глобальным разрешением 2,46 Å TnsB (STC) в посткаталитическом состоянии (см. Также дополнительные рисунки 6–7, дополнительную таблицу 2). Карта окрашена в соответствии с карикатурой, описывающей строение на панели d . д Мультфильм ШТнсБ-СТЦ. Комплекс состоит из 4 протомеров shTnsB и 6 олигонуклеотидов ДНК, представляющих посткаталитическое состояние STC (панель 9).0142 и ). С этой точки зрения целевая ДНК расположена вверху структуры, а более длинные концы транспозона расположены внизу. На схеме отмечены непереносимая цепь (NTS) и переносимая цепь (TS) транспозона. Пунктирные линии изображают взаимодействие между MD и NTD1 из-за кривизны ветвей ДНК. e Ленточная диаграмма, показывающая обзор сборки ШТнсБ-СТЦ.
Также дополнительные рисунки 6–7, дополнительную таблицу 2). Карта окрашена в соответствии с карикатурой, описывающей строение на панели d . д Мультфильм ШТнсБ-СТЦ. Комплекс состоит из 4 протомеров shTnsB и 6 олигонуклеотидов ДНК, представляющих посткаталитическое состояние STC (панель 9).0142 и ). С этой точки зрения целевая ДНК расположена вверху структуры, а более длинные концы транспозона расположены внизу. На схеме отмечены непереносимая цепь (NTS) и переносимая цепь (TS) транспозона. Пунктирные линии изображают взаимодействие между MD и NTD1 из-за кривизны ветвей ДНК. e Ленточная диаграмма, показывающая обзор сборки ШТнсБ-СТЦ.
Полноразмерное изображение
Результаты
Выделение shTnsB и создание комплекса переноса цепи (STC)
Рекомбинантный белок shTnsB был экспрессирован в E. coli и очищен с использованием комбинации аффинной и эксклюзионной хроматографии (SEC). Белок вел себя как мономер в SEC-MALS с молекулярной массой 68 кДа (дополнительные рис. 2a, b, методы). Мы проанализировали его ДНК-связывающие свойства с помощью EMSA с использованием олигонуклеотидов с разным количеством коротких и длинных концевых повторов (SR и LTR соответственно), присутствующих на левом и правом концах (LE и RE) последовательностей транспозонов (дополнительная рис. 2c, г, дополнительная таблица 1). Анализ сдвигов полос со всеми различными субстратами выявил лестницу дискретных полос, относительная подвижность которых обратно зависела от концентрации белка, предполагая связывание одного белкового мономера на повтор. Сборки выше шести или семи белков не наблюдались, поскольку их размер препятствовал попаданию в акриламидный гель (дополнительный рисунок 2c). Затем мы проверили, может ли shTnsB независимо связывать RE или LE. Оба комплекса, shTnsB-RE и shTnsB-LE, были обнаружены, что указывает на то, что присутствие обоих концов не требуется для связывания shTnsB с повторами (дополнительный рисунок 2d). Точно так же количество полос, наблюдаемых при смешивании shTnsB с RE, соответствует 5 сайтам связывания TnsB.
2a, b, методы). Мы проанализировали его ДНК-связывающие свойства с помощью EMSA с использованием олигонуклеотидов с разным количеством коротких и длинных концевых повторов (SR и LTR соответственно), присутствующих на левом и правом концах (LE и RE) последовательностей транспозонов (дополнительная рис. 2c, г, дополнительная таблица 1). Анализ сдвигов полос со всеми различными субстратами выявил лестницу дискретных полос, относительная подвижность которых обратно зависела от концентрации белка, предполагая связывание одного белкового мономера на повтор. Сборки выше шести или семи белков не наблюдались, поскольку их размер препятствовал попаданию в акриламидный гель (дополнительный рисунок 2c). Затем мы проверили, может ли shTnsB независимо связывать RE или LE. Оба комплекса, shTnsB-RE и shTnsB-LE, были обнаружены, что указывает на то, что присутствие обоих концов не требуется для связывания shTnsB с повторами (дополнительный рисунок 2d). Точно так же количество полос, наблюдаемых при смешивании shTnsB с RE, соответствует 5 сайтам связывания TnsB. Однако количество полос, обнаруженных при смешивании shTnsB с LE, составило 4, т.е. на одну больше, чем ожидаемые три сайта связывания. Это можно объяснить взаимодействием двух комплексов shTnsB:LE, подобным тому, которое наблюдается в комплексе STC (рис. 1), или в других структурах транспозазы, не связанных с ДНК-мишенью, например, в Tn5 19 . Однако агрегация комплекса shTnsB:LE также может вызывать сдвиг полосы и не может быть исключена как возможность. Наконец, мы проверили связывание shTnsB с двухцепочечной ДНК, содержащей только LTR (6)-SR (1) (дополнительный рисунок 2e), и, как и ожидалось, обнаружили две полосы, соответствующие двум сайтам связывания shTnsB. Кроме того, мы инкубировали этот комплекс в буфере, содержащем различные концентрации NaCl, при 37 и 45 °C, чтобы определить, могут ли эти переменные изменить аффинность shTnsB. Однако на ассоциацию эти изменения не повлияли. В целом, результаты EMSA показали, что shTnsB связывает каждый повтор, присутствующий как в RE, так и в LE, независимым от груза способом.
Однако количество полос, обнаруженных при смешивании shTnsB с LE, составило 4, т.е. на одну больше, чем ожидаемые три сайта связывания. Это можно объяснить взаимодействием двух комплексов shTnsB:LE, подобным тому, которое наблюдается в комплексе STC (рис. 1), или в других структурах транспозазы, не связанных с ДНК-мишенью, например, в Tn5 19 . Однако агрегация комплекса shTnsB:LE также может вызывать сдвиг полосы и не может быть исключена как возможность. Наконец, мы проверили связывание shTnsB с двухцепочечной ДНК, содержащей только LTR (6)-SR (1) (дополнительный рисунок 2e), и, как и ожидалось, обнаружили две полосы, соответствующие двум сайтам связывания shTnsB. Кроме того, мы инкубировали этот комплекс в буфере, содержащем различные концентрации NaCl, при 37 и 45 °C, чтобы определить, могут ли эти переменные изменить аффинность shTnsB. Однако на ассоциацию эти изменения не повлияли. В целом, результаты EMSA показали, что shTnsB связывает каждый повтор, присутствующий как в RE, так и в LE, независимым от груза способом.
Первоначально мы подготовили сетки крио-ЭМ и собрали данные, используя образец, содержащий shTnsB, а также RE и LE (т.е. две последовательности двухцепочечной ДНК, одна с последовательностью RE и одна с последовательностью LE без груза, дополнительный рисунок 2e). Эта выборка была неоднородной и страдала преимущественной ориентацией. Обработка этих данных привела к реконструкциям с низким разрешением недостаточного качества для построения модели атома (дополнительный рисунок 3). Однако можно было наблюдать два выступа, связанных с удлиненной плотностью, что свидетельствует о присутствии двух протомеров shTnsB, связанных с поверхностью двухцепочечной ДНК набора SR-LTR. Удлиненная плотность, приписываемая ДНК, изгибается таким же образом, как в недавно опубликованной структуре RE-связанного ecTnsB 9.0072 18 , что также подтверждает, что карта с низким разрешением соответствует комплексу перед транспозицией.
Чтобы стабилизировать комплекс shTnsB-ДНК, мы разработали олигонуклеотиды для восстановления STC, то есть состояния, представляющего посттранспозицию, а не претранспозицию, как описано выше (Методы, дополнительная таблица 1 и дополнительная рис. 4). Целевые последовательности для восстановленного комплекса были выбраны на основе природных последовательностей, фланкирующих сайт прикрепления в геноме S. hofmannii UTEX 2349. Эта стратегия аналогична стратегии получения транспозосомной структуры Р-элемента 20 , за исключением того, что в ней используется не симметричная ДНК STC, а природные асимметричные последовательности STC S. hofmannii UTEX 2349 , чтобы представить родной комплекс. ДНК STC состоит из двух перенесенных цепей (TS) и неперенесенных цепей (NTS), представляющих LTR(8)-SR(5) и SR(1)-LTR(6) двухцепочечную ДНК, связанную с сайтом прикрепления CAST (дополнительная рис. 4). Расстояние 5 п.н. между сайтами вставки LTR(8)-SR(5) и SR(1)-LTR(6) было выбрано, поскольку транспозиция CAST вызывает дупликацию 5 п.н. в сайте вставки 9.0072 21 (рис. 1а).
4). Целевые последовательности для восстановленного комплекса были выбраны на основе природных последовательностей, фланкирующих сайт прикрепления в геноме S. hofmannii UTEX 2349. Эта стратегия аналогична стратегии получения транспозосомной структуры Р-элемента 20 , за исключением того, что в ней используется не симметричная ДНК STC, а природные асимметричные последовательности STC S. hofmannii UTEX 2349 , чтобы представить родной комплекс. ДНК STC состоит из двух перенесенных цепей (TS) и неперенесенных цепей (NTS), представляющих LTR(8)-SR(5) и SR(1)-LTR(6) двухцепочечную ДНК, связанную с сайтом прикрепления CAST (дополнительная рис. 4). Расстояние 5 п.н. между сайтами вставки LTR(8)-SR(5) и SR(1)-LTR(6) было выбрано, поскольку транспозиция CAST вызывает дупликацию 5 п.н. в сайте вставки 9.0072 21 (рис. 1а).
Восстановленная ДНК STC была смешана с очищенным белком, и сборка комплекса shTnsB-STC была проверена с помощью SEC-MALS (дополнительная рис. 2b). Наблюдали два пика: пик с более низкой молекулярной массой содержал несвязанный белок и ДНК, использованные при восстановлении, в то время как пик с высокой молекулярной массой, который элюировался при 358,7 кДа, очень хорошо совпадал с ассоциацией ДНК STC (104,6 кДа) и 4 протомеров shTnsB (68 кДа), теоретическая молекулярная масса которого составляет 376,6 кДа. Эта сборка была подвергнута анализу отдельных частиц с помощью крио-ЭМ, в результате чего была получена карта с разрешением 2,46 Å, на которой мы построили атомную модель комплекса shTnsB-STC в его посткаталитическом состоянии (рис. 1а).
2b). Наблюдали два пика: пик с более низкой молекулярной массой содержал несвязанный белок и ДНК, использованные при восстановлении, в то время как пик с высокой молекулярной массой, который элюировался при 358,7 кДа, очень хорошо совпадал с ассоциацией ДНК STC (104,6 кДа) и 4 протомеров shTnsB (68 кДа), теоретическая молекулярная масса которого составляет 376,6 кДа. Эта сборка была подвергнута анализу отдельных частиц с помощью крио-ЭМ, в результате чего была получена карта с разрешением 2,46 Å, на которой мы построили атомную модель комплекса shTnsB-STC в его посткаталитическом состоянии (рис. 1а).
Общая структура комплекса shTnsB-STC
Полипептид shTnsB состоит из N-концевого домена, который можно разделить на два субдомена (NTD1 и NTD2), каталитического домена DDE (DDE), среднего домена (MD) , домен олигомеризации (OD) и С-концевой домен (CTD) (рис. 1b). Архитектура shTnsB напоминает архитектуру Mu-транспозазы (MuA) 22 , хотя идентичность последовательности ограничена каталитическим доменом DDE (дополнительный рисунок 1a). Мы собрали 4728 фильмов и установили координаты для 90,6 миллиона частиц, которые были уменьшены до 415 тысяч после 2D-классификации. Используя этот набор частиц, мы выполнили неравномерное уточнение, трехмерный анализ изменчивости и последующее гетерогенное уточнение в cryoSPARC 23 . Этот подход дал две карты при 2,5 и 2,8 Å. Дальнейшие этапы уточнения и трехмерного анализа изменчивости с использованием 260 тыс. частиц карты 2,5 Å позволили создать крио-ЭМ-карту с глобальным разрешением 2,46 Å, что позволило смоделировать комплекс shTnsB-STC (рис. 1c, дополнительные рис. 5–7, Дополнительная таблица 2 и методы). Однако большая гибкость ДНК поставила под угрозу визуализацию концов ДНК, содержащих SR (5) и SR (1), что не позволило определить возможные контакты с shTnsB (дополнительный рисунок 7, дополнительный фильм 1 и дополнительная таблица 1).
Мы собрали 4728 фильмов и установили координаты для 90,6 миллиона частиц, которые были уменьшены до 415 тысяч после 2D-классификации. Используя этот набор частиц, мы выполнили неравномерное уточнение, трехмерный анализ изменчивости и последующее гетерогенное уточнение в cryoSPARC 23 . Этот подход дал две карты при 2,5 и 2,8 Å. Дальнейшие этапы уточнения и трехмерного анализа изменчивости с использованием 260 тыс. частиц карты 2,5 Å позволили создать крио-ЭМ-карту с глобальным разрешением 2,46 Å, что позволило смоделировать комплекс shTnsB-STC (рис. 1c, дополнительные рис. 5–7, Дополнительная таблица 2 и методы). Однако большая гибкость ДНК поставила под угрозу визуализацию концов ДНК, содержащих SR (5) и SR (1), что не позволило определить возможные контакты с shTnsB (дополнительный рисунок 7, дополнительный фильм 1 и дополнительная таблица 1).
Структура комплекса shTnsB-STC напоминает удлиненный X с изогнутыми плечами разной длины. Более длинные плечи соответствуют концам транспозона, а короткие — изогнутой ДНК-мишени (рис. 1г, д, 2а). Четыре протомера shTnsB (shTnsB1-4) представляют собой переплетенную сборку на ДНК STC. Белок находится в двух разных конформациях, чтобы ассоциироваться с ДНК STC и строить комплекс (рис. 2b). shTnsB1 и 2 изображают вытянутую конформацию вдоль разветвленной структуры нуклеиновой кислоты, в которой мы не смогли обнаружить домены OD и CTD (рис. 1б, г). В обоих протомерах каждый из доменов NTD1 и NTD2 связан с LTR(8) и LTR(6), а каталитический домен DDE визуализируется в транс-положении на стыке ДНК-мишени и концов транспозона. Такое расположение каталитического домена в транс-положении является обычным при транспозиции ДНК, поскольку оно делает расщепление фосфодиэфира зависимым от сборки комплекса (рис. 1e, 2b). Со стороны ДНК-мишени сборка стабилизируется за счет взаимодействия остова с доменами MD и DDE протомеров shTnsB1 и 2 (R416, Q427, K29).0, N428) (рис. 2б). Таким образом, shTnsB1 осуществляет распознавание ДНК на конце LTR(8), а его домен DDE катализирует атаку 3´OH в ответвлении LTR(6) на ДНК-мишень, способствуя реакции переноса цепи.
1г, д, 2а). Четыре протомера shTnsB (shTnsB1-4) представляют собой переплетенную сборку на ДНК STC. Белок находится в двух разных конформациях, чтобы ассоциироваться с ДНК STC и строить комплекс (рис. 2b). shTnsB1 и 2 изображают вытянутую конформацию вдоль разветвленной структуры нуклеиновой кислоты, в которой мы не смогли обнаружить домены OD и CTD (рис. 1б, г). В обоих протомерах каждый из доменов NTD1 и NTD2 связан с LTR(8) и LTR(6), а каталитический домен DDE визуализируется в транс-положении на стыке ДНК-мишени и концов транспозона. Такое расположение каталитического домена в транс-положении является обычным при транспозиции ДНК, поскольку оно делает расщепление фосфодиэфира зависимым от сборки комплекса (рис. 1e, 2b). Со стороны ДНК-мишени сборка стабилизируется за счет взаимодействия остова с доменами MD и DDE протомеров shTnsB1 и 2 (R416, Q427, K29).0, N428) (рис. 2б). Таким образом, shTnsB1 осуществляет распознавание ДНК на конце LTR(8), а его домен DDE катализирует атаку 3´OH в ответвлении LTR(6) на ДНК-мишень, способствуя реакции переноса цепи. Симметричное расположение ДНК STC наблюдается для shTnsB2, который катализирует перенос цепи LTR(8) на другую цепь ДНК-мишени (рис. 1а, 2б).
Симметричное расположение ДНК STC наблюдается для shTnsB2, который катализирует перенос цепи LTR(8) на другую цепь ДНК-мишени (рис. 1а, 2б).
a На верхней панели показана схема S. hofmannii подвижный элемент с левым и правым концами (LE и RE) с каждой стороны ДНК-груза. Сильный цветовой тон показывает область каждого элемента, присутствующего в структуре. Нижняя панель не включает белковые фрагменты в структуру и показывает ДНК STC. ДНК окрашена по верхней схеме для LE и RE, а участок ДНК-мишени показан серым цветом. Желтые точки указывают сайт, где произошла прямая атака 3′-ОН на ДНК-мишень, чтобы способствовать реакции переноса цепи. b Подробное изображение ассоциации протомера shTnsB с ДНК STC. Две различные ассоциации наблюдаются для молекул shTnsB. Различные участки ДНК, взаимодействующие с белками, показаны соответствующим ярким цветовым оттенком. c Подробное изображение спиральных доменов NTD1 и 2, показывающее специфические взаимодействия с основаниями ДНК в области LTR(6). Маркировка нуклеотидов соответствует цветовому коду на рис. 1d, e (см. также рис. 1d и дополнительный рис. 8).
Маркировка нуклеотидов соответствует цветовому коду на рис. 1d, e (см. также рис. 1d и дополнительный рис. 8).
Изображение полного размера
Протомеры shTnsB3 и 4 расположены в другой конформации с небольшим количеством контактов с остовом ДНК (рис. 2б). Домены NTD1 и NTD2 этих протомеров не обнаружены в нашей структуре, что свидетельствует об их высокой подвижности. Кроме того, домен DDE вытягивается из ДНК, в то время как MD размещает области NTS с концов LTR (6) и LTR (8) после катализа. Структура выявила важную роль этих двух протомеров в ассоциации shTnsB1 и shTnsB2 с ДНК, поскольку в этой конформации они демонстрируют упорядоченный домен OD. Спираль между S502 и S522 не наблюдается в shTnsB1-2, но в конформации, принятой shTnsB3-4, спираль облегчает сборку комплекса STC за счет переплетения NTD2 shTnsB1 и shTnsB2 с доменом DDE противоположного протомера. в каждой ветви ДНК, тем самым связывая распознавание двух концов транспозона с каталитическими сайтами shTnsB1-2 (рис. 1d, 2b).
1d, 2b).
Распознавание shTnsB-ДНК
Распознавание LTR(6) и LTR(8) осуществляется спиральными доменами NTD1 и 2 субъединиц shTnsB1 и 2 (рис. 2c). Структура домена NTD1 обнаруживает спираль-поворот-спираль Myb/гомеодомен-подобную складку. Этот домен является членом суперсемейства гомеобоксных факторов транскрипции 24 . Прямые контакты оснований с помощью спирали, связывающей большую бороздку, в основном объясняют специфичное для последовательности распознавание. R77, который хорошо консервативен в гомологах цианобактерий и в Tn5053, но заменен лизином в MuA и аланином в ecTnsB (дополнительная рис. 1), образует полярные контакты с dG37, тогда как R81 связывается с основаниями последовательных нуклеотидов dG36 и dT37. на комплементарной цепи LTR (6) в LE (дополнительные рис. 7, 8 и дополнительная таблица 1). Другие остатки, взаимодействующие с ДНК (R58, R66, K84 и T78), обнаруживают полярные взаимодействия с остовом обеих цепей ДНК.
NTD1 соединен с NTD2 длинной петлей, которая проходит вдоль бороздки ДНК. Домен NTD2 функционально аналогичен второму ДНК-связывающему домену в других транспозазах. Однако он демонстрирует ограниченную консервативность по отношению к ближайшим гомологам shTnsB (дополнительный рисунок 1), а его структура напоминает парный домен, обнаруженный в генах парного ящика (Pax). Хорошо консервативный R99 в этой петле создает полярные взаимодействия с основаниями dT31 и dA32 и dA45, в то время как R106 также распознает основания dT30 и dT46 в разных цепях. Остальные ассоциации петли с ДНК включают остовные контакты. Домен NTD2 также связывается с ДНК в большой бороздке. Однако он отображает меньше конкретных контактов с базами. Только R158 и K154 связаны с dA48-dG49.и dG24 соответственно, тогда как остальные остатки связаны с остовом нуклеиновой кислоты. Эти взаимодействия аналогичны в доменах NTD1 и 2 на ветвях LTR (6) и LTR (8) комплекса белок-ДНК (дополнительная рис. 7).
Домен NTD2 функционально аналогичен второму ДНК-связывающему домену в других транспозазах. Однако он демонстрирует ограниченную консервативность по отношению к ближайшим гомологам shTnsB (дополнительный рисунок 1), а его структура напоминает парный домен, обнаруженный в генах парного ящика (Pax). Хорошо консервативный R99 в этой петле создает полярные взаимодействия с основаниями dT31 и dA32 и dA45, в то время как R106 также распознает основания dT30 и dT46 в разных цепях. Остальные ассоциации петли с ДНК включают остовные контакты. Домен NTD2 также связывается с ДНК в большой бороздке. Однако он отображает меньше конкретных контактов с базами. Только R158 и K154 связаны с dA48-dG49.и dG24 соответственно, тогда как остальные остатки связаны с остовом нуклеиновой кислоты. Эти взаимодействия аналогичны в доменах NTD1 и 2 на ветвях LTR (6) и LTR (8) комплекса белок-ДНК (дополнительная рис. 7).
Сборка комплекса белок-ДНК
Домены NTD2 субъединиц shTnsB1-2 соединены с доменами DDE длинной петлей. Переплетающаяся конформация этих протомеров с ДНК позволяет shTnsB1, который распознает ДНК в LTR(8), катализировать реакцию переноса цепи на конце LTR(6), и наоборот для shTnsB2 (рис. 1c, 2b). Этому перекрестному расположению каталитических доменов DDE способствует конформация молекул shTnsB3-4, которые стабилизируют сборку за счет интеркалирования длинной двойной спирали, которая собирает домены NTD2 и DDE различных субъединиц. Соседний MD вмещает 5 ′ NTS (рис. 1d, рис. 3a–c). С-концевая спираль OD плотно прилегает между доменом DDE shTnsB2 и NTD2 shTnsB1 сетью контактов, сочетающих полярные и неполярные взаимодействия (рис. 3b), причем первая на конце (E360-K520 ), средний (R367-D512-Q508) и начальный (h372-S505) участки спирали. Другое сильное полярное взаимодействие наблюдается между боковыми цепями D514 в OD и R137 в NTD2. Короткая петля соединяет этот участок с N-концевой спиралью OD. Взаимодействие второй спирали OD с доменом DDE стабилизируется за счет неполярных взаимодействий, а группа остатков в MD shTnsB3 (R416, Q425, N428) вместе с R174 и R179в shTnsB2 образуют электроположительно заряженную область, стабилизирующую основания 5′-выступа (рис.
Переплетающаяся конформация этих протомеров с ДНК позволяет shTnsB1, который распознает ДНК в LTR(8), катализировать реакцию переноса цепи на конце LTR(6), и наоборот для shTnsB2 (рис. 1c, 2b). Этому перекрестному расположению каталитических доменов DDE способствует конформация молекул shTnsB3-4, которые стабилизируют сборку за счет интеркалирования длинной двойной спирали, которая собирает домены NTD2 и DDE различных субъединиц. Соседний MD вмещает 5 ′ NTS (рис. 1d, рис. 3a–c). С-концевая спираль OD плотно прилегает между доменом DDE shTnsB2 и NTD2 shTnsB1 сетью контактов, сочетающих полярные и неполярные взаимодействия (рис. 3b), причем первая на конце (E360-K520 ), средний (R367-D512-Q508) и начальный (h372-S505) участки спирали. Другое сильное полярное взаимодействие наблюдается между боковыми цепями D514 в OD и R137 в NTD2. Короткая петля соединяет этот участок с N-концевой спиралью OD. Взаимодействие второй спирали OD с доменом DDE стабилизируется за счет неполярных взаимодействий, а группа остатков в MD shTnsB3 (R416, Q425, N428) вместе с R174 и R179в shTnsB2 образуют электроположительно заряженную область, стабилизирующую основания 5′-выступа (рис. 3c). Наконец, домены MD и DDE протомера shTnsB3 связаны с NTD1 shTnsB2, главным образом, группой неполярных взаимодействий. Интересно, что N-концевая β-цепь shTnsB2 встроена в качестве дополнительной цепи в антипараллельный β-лист MD (рис. 3d). Дополнительные полярные взаимодействия боковых цепей (E422-K169, N462-K102 и E54-h383) дополняют эту ассоциацию. В целом, описанные здесь ассоциации очень похожи в случае shTnsB4 с shTnsB1 и 2. Отметим, что взаимодействия между shTnsB3 и 4 в комплексе shTnsB-STC не наблюдается.
3c). Наконец, домены MD и DDE протомера shTnsB3 связаны с NTD1 shTnsB2, главным образом, группой неполярных взаимодействий. Интересно, что N-концевая β-цепь shTnsB2 встроена в качестве дополнительной цепи в антипараллельный β-лист MD (рис. 3d). Дополнительные полярные взаимодействия боковых цепей (E422-K169, N462-K102 и E54-h383) дополняют эту ассоциацию. В целом, описанные здесь ассоциации очень похожи в случае shTnsB4 с shTnsB1 и 2. Отметим, что взаимодействия между shTnsB3 и 4 в комплексе shTnsB-STC не наблюдается.
a Обзор архитектуры протомеров shTnsB и взаимодействий между двумя типами конформаций, наблюдаемых для белка на одной из ветвей ДНК STC (прозрачно). Цветные овалы обозначают увеличенные области на других панелях. b , c Подробное изображение взаимодействия OD shTnsB3 с доменами NTD2 и DDE shTnsB1 и 2 соответственно. д Увеличение области LTR(6), изображающей ассоциацию N-концевого β-листа NTD1 с MD shTnsB3.
Изображение в полный размер
В совокупности эта сеть взаимодействий вдоль Х-образного комплекса предполагает, что различная конформация протомеров shTnsB1-2 и shTnsB3-4 благоприятствует транс-архитектуре, скорее всего, как стратегии связывания белок-ДНК. комплексная сборка с катализом ДДЕ.
Каталитический домен DDE
Транспозазы из нескольких суперсемейств обладают каталитическим доменом, содержащим триаду кислых аминокислот (DDE или DDD) 25,26 . Этот белковый модуль катализирует реакцию транспозиции, при которой элемент вырезается из донорского сайта и вставляется в геном или в мобильный генетический элемент. Мутации в этих каталитических остатках показали их критическую роль в транспозиции в доменах DDE транспозаз Tn5 и Tn10 27,28 . Интеграция элементов в новое место генома обычно приводит к короткой дупликации сайта-мишени из последовательностей хозяина (2–10 п.н.). В shTnsB мотив DDE состоит из двух остатков аспарагиновой кислоты (D205, D287) и остатка глутаминовой кислоты (E321), расположенных в консервативном ядре, образующем характерную РНКазу Н-подобную складку, объединяющую α-спирали и β-цепи (рис. 4а, дополнительный рис. 8е). Трехмерная структура триады ДДЭ образует каталитический карман, который связывается с ионами двухвалентного металла, которые помогают в различных нуклеофильных реакциях во время расщепления ДНК. Домены DDE четырех молекул shTnsB в сборке очень хорошо накладываются друг на друга (среднеквадратичное отклонение 0,59).Å среднеквадратичное значение более 160 Cα). Однако карман DDE демонстрирует два разных расположения в зависимости от конформации протомера shTnsB в комплексе STC (рис. 1). Карманы DDE shTnsB3 и 4 не контактируют с ДНК, и каталитические остатки этих субъединиц не расположены должным образом для реакции расщепления (рис. 4b). В случае shTnsB1 и 2 они обнаруживают каталитический карман DDE после завершения реакции переноса цепи. Можно визуализировать дополнительную плотность в кармане (рис. 4а), которую можно отнести к H 2 O или Mg 2+ . E321 находится на расстоянии 6 Å, так как структура захватывается в посткаталитическом состоянии.
4а, дополнительный рис. 8е). Трехмерная структура триады ДДЭ образует каталитический карман, который связывается с ионами двухвалентного металла, которые помогают в различных нуклеофильных реакциях во время расщепления ДНК. Домены DDE четырех молекул shTnsB в сборке очень хорошо накладываются друг на друга (среднеквадратичное отклонение 0,59).Å среднеквадратичное значение более 160 Cα). Однако карман DDE демонстрирует два разных расположения в зависимости от конформации протомера shTnsB в комплексе STC (рис. 1). Карманы DDE shTnsB3 и 4 не контактируют с ДНК, и каталитические остатки этих субъединиц не расположены должным образом для реакции расщепления (рис. 4b). В случае shTnsB1 и 2 они обнаруживают каталитический карман DDE после завершения реакции переноса цепи. Можно визуализировать дополнительную плотность в кармане (рис. 4а), которую можно отнести к H 2 O или Mg 2+ . E321 находится на расстоянии 6 Å, так как структура захватывается в посткаталитическом состоянии. Расстояния дополнительной плотности до D205, D287 и фосфатов основной цепи позволяют предположить, что плотность может быть отнесена к молекуле H 2 O, образующейся после 3′-OH-атаки одной из цепей мобильного элемента на ДНК-мишень. (рис. 1а, 4а, дополнительный рис. 8).
Расстояния дополнительной плотности до D205, D287 и фосфатов основной цепи позволяют предположить, что плотность может быть отнесена к молекуле H 2 O, образующейся после 3′-OH-атаки одной из цепей мобильного элемента на ДНК-мишень. (рис. 1а, 4а, дополнительный рис. 8).
a В обведенной области находится один из каталитических центров в комплексе shTnsB-STC. Увеличение области показывает конформацию каталитического центра после катализа. Предполагаемая молекула воды и расстояния до соседних каталитических остатков и ДНК показаны в Å. b Наложение доменов DDE двух конформаций, обнаруженных в комплексе белок-ДНК для shTnsB. c Суперпозиция доменов DDE shTnsB2 и 3 с ecTnsB (PDB: 7PIK) и основным доменом MuA (PDB: 1BCO). СКО для суперпозиции находятся в диапазоне от 0,7 до 1,2 Å для 160 и 250 Cα. d Нижняя панель показывает, что, несмотря на высокое структурное сходство доменов DDE, единственной субъединицей во всех этих структурах, которая демонстрирует компетентный каталитический карман, является shTnsB2, которая является единственной, связанной с ДНК.
Изображение в натуральную величину
Несмотря на различия в последовательностях, современная структура комплекса конечного узнавания транспозона Tn7 ecTnsB транспозазы 18 обеспечивает функциональный гомолог для сравнения. Домен DDE является наиболее консервативным модулем shTnsB, и поиск гомологов shTnsB DDE с использованием DALI 29 выявил домены ecTnsB и транспозазы фага Mu как наиболее близкие структурные гомологи вместе с различными вирусными интегразами (SRV, HIV- 1, ВТЛВ-1). Сравнение двух конформаций shTnsB с этими белками показало, что ядро домена очень хорошо накладывалось на другие интегразы (рис. 4c), но только каталитический карман shTnsB1-2 показал триаду кислых аминокислот на соответствующих расстояниях для катализа реакция переноса цепи (рис. 4d). В остальных случаях каталитические остатки не были правильно расположены. Примечательно, что протомеры ecTnsB в комплексе распознавания концов имеют конформацию, сходную с протомерами shTnsB3 и 4 в STC. Однако в последнем ДНК-связывающие домены этих двух субъединиц не визуализировались (рис. 1–2), что позволяет предположить, что высокая гибкость, наблюдаемая в дистальных отделах ДНК STC (дополнительный фильм 1), нарушает их связывание с SR(1) и SR(5) области LE и RE.
Однако в последнем ДНК-связывающие домены этих двух субъединиц не визуализировались (рис. 1–2), что позволяет предположить, что высокая гибкость, наблюдаемая в дистальных отделах ДНК STC (дополнительный фильм 1), нарушает их связывание с SR(1) и SR(5) области LE и RE.
В совокупности структурное сравнение показывает, что домен DDE поддерживает очень хорошо сохранившуюся трехмерную структуру; однако сравнение различных структурных гомологов предполагает, что архитектура каталитического кармана транспозазы устроена правильно, как только она связывается с ДНК-мишенью.
Связывание ДНК транспозазами shTnsB, ecTnsB и MuA
Белок shTnsB проявляет гомологию с транспозазами ecTnsB и MuA. Однако, помимо домена DDE, между ними сохраняется несколько остатков (дополнительный рисунок 1). Эти различия особенно очевидны в ДНК-связывающих доменах, где многочисленные выравнивания последовательностей из нескольких канонических белков Tn7 TnsB и различных классов CAST-элементов показали существенное расхождение 18 . NTD1 shTnsB демонстрирует значительное трехмерное сходство с доменом HTH DBD1 в ecTnsB (RMSD 2,4 Å для 66 Cα) и доменом Iβ MuA (RMSD 2,8 Å для 62 Cα). Тем не менее, присутствие N-концевой β-цепи (N32-T36) исключает shTnsB, и эта область играет важную роль в построении сборки комплекса STC (рис. 3d). Эта структурная особенность не наблюдается в комплексе MuA-STC 22 , единственной доступной до сих пор структуре прокариотического комплекса транспозазы STC (рис. 5).
NTD1 shTnsB демонстрирует значительное трехмерное сходство с доменом HTH DBD1 в ecTnsB (RMSD 2,4 Å для 66 Cα) и доменом Iβ MuA (RMSD 2,8 Å для 62 Cα). Тем не менее, присутствие N-концевой β-цепи (N32-T36) исключает shTnsB, и эта область играет важную роль в построении сборки комплекса STC (рис. 3d). Эта структурная особенность не наблюдается в комплексе MuA-STC 22 , единственной доступной до сих пор структуре прокариотического комплекса транспозазы STC (рис. 5).
ДНК-связывающие домены трех различных транспозаз выровнены по ДНК (PDB: 8AA5, 7PIK и 4FCY соответственно). В случае MuA показаны одни и те же домены, связанные с разными повторами. Для shTnsB были построены только домены протомеров 1 и 2. В случае ecTnsB изображен ДНК-связывающий домен одной из субъединиц концевого комплекса транспозона.
Полноразмерное изображение
Интересно, что домен NTD2 shTnsB не проявляет трехмерного сходства с его аналогами в транспозазах ecTnsB и MuA (рис. 5). Вместо этого, NTD2 хорошо накладывается на Pax6, фактор транскрипции, содержащий двудольный спаренный ДНК-связывающий домен (RMSD 2,8 Å для 60 Cα), который играет критическую роль в развитии глаза, носа, поджелудочной железы и центральной нервной системы 30 . Удлиненный линкер, соединяющий NTD1 и 2, частично визуализируется в ecTnsB и не наблюдается в ДНК-связывающих доменах MuA (рис. 5). Этот линкер образует контакты малой бороздки, а карбоксиконцевая единица спираль-поворот-спираль образует контакты основания в большой бороздке, как это наблюдается в комплексе shTnsB-STC (рис. 5).
5). Вместо этого, NTD2 хорошо накладывается на Pax6, фактор транскрипции, содержащий двудольный спаренный ДНК-связывающий домен (RMSD 2,8 Å для 60 Cα), который играет критическую роль в развитии глаза, носа, поджелудочной железы и центральной нервной системы 30 . Удлиненный линкер, соединяющий NTD1 и 2, частично визуализируется в ecTnsB и не наблюдается в ДНК-связывающих доменах MuA (рис. 5). Этот линкер образует контакты малой бороздки, а карбоксиконцевая единица спираль-поворот-спираль образует контакты основания в большой бороздке, как это наблюдается в комплексе shTnsB-STC (рис. 5).
Сравнение позволяет предположить, что белки TnsB имеют общий общий способ связывания ДНК, включая два домена, которые распознают разные SR и LTR в сборке. Однако домены, участвующие в связывании, претерпевают различные структурные вариации для осуществления специфических взаимодействий белок-ДНК для каждой транспозазы. Это разнообразие может быть необходимо для размещения последовательностей большого количества Tn7-подобных элементов, предотвращая распознавание транспозазами последовательностей ДНК, принадлежащих другим транспозонам 31 .
Комплексы STC
Структура концевого комплекса транспозона ecTnsB предоставила информацию о распознавании концов транспозона 18 , но до сих пор нет структурной информации об их комплексе STC. Конформация субъединиц ecTnsB в концевом комплексе транспозона напоминает расположение, принятое доменом DDE протомеров shTnsB3 и 4 в комплексе shTnsB-STC. Однако положения доменов MD и OD ориентированы по-разному (рис. 4c). Тем не менее, эта конформация сильно отличается от архитектуры протомеров 1 и 2 shTnsB, которые участвуют в катализе реакции переноса цепи (рис. 2b). Конформация этих субъединиц больше похожа на аналоги в комплексе MuA-STC (рис. 6а).
Рис. 6: Анализ комплексов переноса цепи shTnsB и MuA. a Комплексы shTnsB-STC и MuA-STC показаны в той же цветовой схеме, что и на рис. 1, за исключением ДНК, окрашенной в белый цвет. b Обведенные кружками области на обоих комплексах сравниваются, чтобы показать ключевые различия в сборке комплексов STC.
Изображение в полный размер
Хотя последовательность shTnsB отдаленно связана с транспозазой фага MuA (дополнительная рис. 1), общая ассоциация комплекса MuA-STC имеет сходство с белком CAST (рис. 6a). Однако сборка также имеет важные отличия. Одно из основных отличий заключается в том, что в комплексе MuA-STC ДНК-связывающие домены субъединиц, участвующих в сборке (MuA 3 и 4), связаны с соответствующими повторами в LE и RE (рис. 6а), тогда как в шТнсБ-СТЦ их нельзя было смоделировать, что свидетельствует об их высокой гибкости. Это мнение подтверждается высокой изменчивостью, наблюдаемой во внешней части Х-образной структуры ДНК (дополнительный фильм 1). Тот факт, что эти области структуры MuA-STC 22 участвуют в контактах кристаллов, могут снижать гибкость, облегчая тем самым связывание доменов Iβ и Iγ с повторами на LE и RE концах.
Любопытно, что ассоциация уникальной N-концевой β-цепи NTD1 в shTnsB не наблюдается в MuA (рис. 6а, б, верхние панели), поскольку этот элемент вторичной структуры отсутствует ни в домене Iβ MuA, ни в DBD1 ecTnsB. Это наблюдение предполагает, что в комплексе ecTnsB-STC эта ассоциация не может происходить. Другое важное отличие возникает из-за сборки комплекса, в котором домен OD в shTnsB интеркалирует с множественными взаимодействиями между NTD2 и каталитическим доменом DDE, создавая ассоциацию между белками shTnsB. В случае MuA ассоциация между четырьмя протомерами в этой области комплекса STC отличается, демонстрируя значительно меньше взаимодействий между разными доменами (рис. 6b). Кроме того, в протомерах 1 и 2 комплекса shTnsB-STC домены OD не наблюдаются, тогда как в случае MuA спирали в домене IIIα перекрещиваются внутри V-образного участка ДНК-мишени (рис. 6а). ). Нельзя было не учитывать возможность того, что взаимодействие между этими спиралями в структуре MuA происходит за счет кристаллических контактов.
Это наблюдение предполагает, что в комплексе ecTnsB-STC эта ассоциация не может происходить. Другое важное отличие возникает из-за сборки комплекса, в котором домен OD в shTnsB интеркалирует с множественными взаимодействиями между NTD2 и каталитическим доменом DDE, создавая ассоциацию между белками shTnsB. В случае MuA ассоциация между четырьмя протомерами в этой области комплекса STC отличается, демонстрируя значительно меньше взаимодействий между разными доменами (рис. 6b). Кроме того, в протомерах 1 и 2 комплекса shTnsB-STC домены OD не наблюдаются, тогда как в случае MuA спирали в домене IIIα перекрещиваются внутри V-образного участка ДНК-мишени (рис. 6а). ). Нельзя было не учитывать возможность того, что взаимодействие между этими спиралями в структуре MuA происходит за счет кристаллических контактов.
Эффект связывания ДНК при транспозиции
Чтобы подтвердить взаимодействие shTnsB:ДНК в STC, мы протестировали транспозиционную активность ряда мутантов с заменой, в которых консервативные аминокислоты, демонстрирующие полярные взаимодействия с ДНК, были заменены аланином (рис. 7, дополнительный рис. 1). Мы мутировали R77, R81 (оба присутствуют в NTD1), R99 (присутствуют в линкере между NTD1 и NTD2), R158 (присутствуют в NTD2), R188, R223, K290 и R380 (присутствуют в домене DDE) (рис. 7b). . Мутанты R77A, R81A и R158A продемонстрировали резкое влияние на активность транспозиции «на-мишень», предполагая, что shTnsB больше не может распознавать повторы в RE и LE (рис. 7b). Примечательно, что нуклеотиды, участвующие в этом взаимодействии (дополнительная рис. 7), сохраняются между различными повторами, что еще раз подтверждает важность этого взаимодействия в распознавании ДНК донора 21 . Что касается R99, хотя он и присутствует в неупорядоченной области shTnsB, он тесно взаимодействует с фосфатным остовом и основаниями LTR. Эти нуклеотиды также консервативны в наборе повторов RE/LE 21 . Однако эффект его замены был переменным: повторы демонстрировали либо более высокую, либо более низкую активность, чем дикий тип. В случае R223A и R380A это также повлияло на активность.
7, дополнительный рис. 1). Мы мутировали R77, R81 (оба присутствуют в NTD1), R99 (присутствуют в линкере между NTD1 и NTD2), R158 (присутствуют в NTD2), R188, R223, K290 и R380 (присутствуют в домене DDE) (рис. 7b). . Мутанты R77A, R81A и R158A продемонстрировали резкое влияние на активность транспозиции «на-мишень», предполагая, что shTnsB больше не может распознавать повторы в RE и LE (рис. 7b). Примечательно, что нуклеотиды, участвующие в этом взаимодействии (дополнительная рис. 7), сохраняются между различными повторами, что еще раз подтверждает важность этого взаимодействия в распознавании ДНК донора 21 . Что касается R99, хотя он и присутствует в неупорядоченной области shTnsB, он тесно взаимодействует с фосфатным остовом и основаниями LTR. Эти нуклеотиды также консервативны в наборе повторов RE/LE 21 . Однако эффект его замены был переменным: повторы демонстрировали либо более высокую, либо более низкую активность, чем дикий тип. В случае R223A и R380A это также повлияло на активность. Эти остатки, по-видимому, участвуют в стабилизации состояния после транспозиции. R223 взаимодействует с центральными нуклеотидами последовательности 5 bp между никами, образованными транспозицией, тогда как R380 взаимодействует с нерасщепленной одноцепочечной ДНК транспозона, расположенной вне комплекса. Наконец, несмотря на свою консервативность и близость к сайту прикрепления, мутанты R188A и K290A не отменял целевую транспозицию. Наши результаты подтверждают функциональную важность этих остатков, участвующих в распознавании LTR и стабилизации STC, и указывают на возможные сайты для улучшения свойств транспозазы для целей инженерии генома.
Эти остатки, по-видимому, участвуют в стабилизации состояния после транспозиции. R223 взаимодействует с центральными нуклеотидами последовательности 5 bp между никами, образованными транспозицией, тогда как R380 взаимодействует с нерасщепленной одноцепочечной ДНК транспозона, расположенной вне комплекса. Наконец, несмотря на свою консервативность и близость к сайту прикрепления, мутанты R188A и K290A не отменял целевую транспозицию. Наши результаты подтверждают функциональную важность этих остатков, участвующих в распознавании LTR и стабилизации STC, и указывают на возможные сайты для улучшения свойств транспозазы для целей инженерии генома.
a Транспозицию проводили путем трансформации бактерий плазмидой, содержащей сайт-мишень, показанный зеленым цветом, распознаваемым Cas12k:sgRNA (pTarget), плазмидой, содержащей донорную ДНК с последовательностями CAST RE и LE, показанными красным ( pDonor) и плазмиду, содержащую кодирующие последовательности sgRNA, Cas12k, TnsC, TniQ и TnsB (pHelper). Интеграцию донорной последовательности в pTarget количественно определяли с помощью количественной ПЦР с использованием набора праймеров, амплифицирующих область pTarget, в которой не происходит транспозиции (PF 9).0144-мишень и PR -мишень ) и набор праймеров, которые амплифицируют область между последовательностями LE-мишени и донора (продукт PF и продукт PR ). b Положение выбранных аминокислот в STC, подлежащих мутации и тестированию на транспозиционную активность. c Кратность изменения активности, измеренная с помощью количественной ПЦР для каждого мутанта TnsB. Для каждого мутанта проводили две или три биологические повторности. Отдельные точки данных, представляющие все технические повторы, обозначены точками. Столбцы представляют собой среднее значение кратности изменения активности, а усы, расположенные вокруг него, представляют собой стандартное отклонение. Активность дикого типа представлена пунктирной линией. Данные предоставляются в виде файла исходных данных.
Интеграцию донорной последовательности в pTarget количественно определяли с помощью количественной ПЦР с использованием набора праймеров, амплифицирующих область pTarget, в которой не происходит транспозиции (PF 9).0144-мишень и PR -мишень ) и набор праймеров, которые амплифицируют область между последовательностями LE-мишени и донора (продукт PF и продукт PR ). b Положение выбранных аминокислот в STC, подлежащих мутации и тестированию на транспозиционную активность. c Кратность изменения активности, измеренная с помощью количественной ПЦР для каждого мутанта TnsB. Для каждого мутанта проводили две или три биологические повторности. Отдельные точки данных, представляющие все технические повторы, обозначены точками. Столбцы представляют собой среднее значение кратности изменения активности, а усы, расположенные вокруг него, представляют собой стандартное отклонение. Активность дикого типа представлена пунктирной линией. Данные предоставляются в виде файла исходных данных.
Изображение в натуральную величину
Обсуждение
В прошлом году мы стали свидетелями значительного увеличения структурных знаний о системах CAST. Доступна структурная информация TnsC, TniQ и Cas12k типа V-K 32,33,34 , а также каскадного комплекса типа I-F, связанного с TniQ 35,36 . Однако, несмотря на эти достижения, нам необходимо точное понимание последовательности взаимодействий и промежуточных звеньев, ведущих к точной вставке ДНК-груза, чтобы решить возможную разработку систем CAST. Это отсутствие полного понимания того, как транспозиция осуществляется с помощью различных CAST, препятствует их повторному использованию для точной вставки ДНК в инструменты редактирования генов. Тип В-К мог представлять собой перспективную систему для развития этих средств за счет уменьшенного количества и размеров ее компонентов. Однако для этой цели необходимо решить проблему образования коинтегратов (рис. 1а).
Концевой комплекс транспозона shTnsB с низким разрешением (дополнительный рисунок 3) предполагает, что распознавание конца транспозона аналогично ecTnsB 18 ; однако высокая гетерогенность и гибкость сборки не позволили провести детальную молекулярную характеристику распознавания ДНК. Это согласуется с экспериментами в системе ecTn7, которые показали, что пре-транспозиционный комплекс менее стабилен, чем пост-транспозиционный комплекс, и что защита транспозонов меняется между пре- и посткаталитической стадиями 17,37 . Кроме того, эксперименты EMSA предполагают, что shTnsB может взаимодействовать с RE и LE в прекаталитическом состоянии в отсутствие ДНК-мишени (дополнительный рисунок 2). Таким образом, взаимодействие shTnsB с концами транспозона, по-видимому, не зависит от присутствия других компонентов системы CAST типа V-K. Более высокая стабильность посткаталитических комплексов привела нас к сборке shTnsB-STC, которую мы смогли определить с высоким разрешением. Этот комплекс представляет собой стадию после завершения реакции переэтерификации с ДНК-мишенью, обнаруживающую ее трехмерную сборку. Общая структура напоминает архитектуру транспозосомы 9 фага MuA.0072 22 . Тем не менее, можно наблюдать ключевые различия между сборками, особенно в домене NTD2 (рис.
Это согласуется с экспериментами в системе ecTn7, которые показали, что пре-транспозиционный комплекс менее стабилен, чем пост-транспозиционный комплекс, и что защита транспозонов меняется между пре- и посткаталитической стадиями 17,37 . Кроме того, эксперименты EMSA предполагают, что shTnsB может взаимодействовать с RE и LE в прекаталитическом состоянии в отсутствие ДНК-мишени (дополнительный рисунок 2). Таким образом, взаимодействие shTnsB с концами транспозона, по-видимому, не зависит от присутствия других компонентов системы CAST типа V-K. Более высокая стабильность посткаталитических комплексов привела нас к сборке shTnsB-STC, которую мы смогли определить с высоким разрешением. Этот комплекс представляет собой стадию после завершения реакции переэтерификации с ДНК-мишенью, обнаруживающую ее трехмерную сборку. Общая структура напоминает архитектуру транспозосомы 9 фага MuA.0072 22 . Тем не менее, можно наблюдать ключевые различия между сборками, особенно в домене NTD2 (рис. 6). Одно сходство между shTnsB и MuA, которое отличает их от системы Tn7, заключается в том, что типы V-K и Mu используют репликативную транспозицию, которая требует расщепления только одной цепи ДНК, в то время как Tn7 выполняет реакцию вырезания и вставки, включающую расщепление неперенесенной цепи TnsA. Следовательно, структурное сходство общего комплекса STC между shTnsB и MuA будет поддерживаться этими механистическими сходствами. Тем не менее, представляется затруднительным установить аналогии возможной комплексной сборки STC ecTnsB 9.0072 18 с использованием структуры shTnsB-STC, так как мы ожидаем, что ecTnsB-STC также должен включать ecTnsA 10 . То же самое может быть экстраполировано для CAST типов I-F и I-B, которые включают в свой состав TnsA.
6). Одно сходство между shTnsB и MuA, которое отличает их от системы Tn7, заключается в том, что типы V-K и Mu используют репликативную транспозицию, которая требует расщепления только одной цепи ДНК, в то время как Tn7 выполняет реакцию вырезания и вставки, включающую расщепление неперенесенной цепи TnsA. Следовательно, структурное сходство общего комплекса STC между shTnsB и MuA будет поддерживаться этими механистическими сходствами. Тем не менее, представляется затруднительным установить аналогии возможной комплексной сборки STC ecTnsB 9.0072 18 с использованием структуры shTnsB-STC, так как мы ожидаем, что ecTnsB-STC также должен включать ecTnsA 10 . То же самое может быть экстраполировано для CAST типов I-F и I-B, которые включают в свой состав TnsA.
Посткаталитическая структура shTnsB-STC выявила очень мало оснований-специфических контактов белок-ДНК (дополнительная рис. 7), как это также наблюдалось в прекаталитическом комплексе ecTnsB 18 . Домен NTD1 в shTnsB частично имеет такую же архитектуру, что и его аналоги в ecTnsB и MuA, в то время как NTD2 уникален в том смысле, что он имеет структурную гомологию с фактором транскрипции Pax6 (рис. 5). N-концевая β-цепь, связывающая распознавание в одном из плеч транспозона с каталитическим центром на противоположной стороне и наоборот, является сингулярной особенностью shTnsB, однако еще предстоит определить, оказывает ли эта особенность некоторую аллостерическую регуляцию в каталитическом сайте DDE. . Две субъединицы shTnsB, участвующие в реакции, демонстрируют кислотную триаду в посткаталитическом состоянии. Атака 3′-ОН цепей транспозона на 5′ ДНК-мишени оставляет несвязанные основания (dT62 и dA63) в каждой цепи и молекулу воды, связанную с остатками аспарагиновой кислоты и остовом ДНК (дополнительная рис. 7) . На основании структуры концевого комплекса shTnsB-STC и концевого комплекса транспозона ecTnsB можно сделать вывод, что активный карман белков TnsB не приспособлен для катализа до связывания ДНК-мишени (рис.
Домен NTD1 в shTnsB частично имеет такую же архитектуру, что и его аналоги в ecTnsB и MuA, в то время как NTD2 уникален в том смысле, что он имеет структурную гомологию с фактором транскрипции Pax6 (рис. 5). N-концевая β-цепь, связывающая распознавание в одном из плеч транспозона с каталитическим центром на противоположной стороне и наоборот, является сингулярной особенностью shTnsB, однако еще предстоит определить, оказывает ли эта особенность некоторую аллостерическую регуляцию в каталитическом сайте DDE. . Две субъединицы shTnsB, участвующие в реакции, демонстрируют кислотную триаду в посткаталитическом состоянии. Атака 3′-ОН цепей транспозона на 5′ ДНК-мишени оставляет несвязанные основания (dT62 и dA63) в каждой цепи и молекулу воды, связанную с остатками аспарагиновой кислоты и остовом ДНК (дополнительная рис. 7) . На основании структуры концевого комплекса shTnsB-STC и концевого комплекса транспозона ecTnsB можно сделать вывод, что активный карман белков TnsB не приспособлен для катализа до связывания ДНК-мишени (рис. 4d). Скорее всего, этот механизм сочетается с транс-сборкой STC, чтобы предотвратить неспецифическое расщепление.
4d). Скорее всего, этот механизм сочетается с транс-сборкой STC, чтобы предотвратить неспецифическое расщепление.
ShTnsB вставляет транспозон со смещением направленности 16 . Хотя концы LTR8-SR5 и LTR6-SR1 почти симметричны, длина и последовательности с правого и левого концов различаются (дополнительный рисунок 2f), что позволяет предположить, что эти различия в сочетании с необходимостью PAM для определения вставки сайте, может повлиять на направленность грузовой ДНК. Это мнение подтверждается наблюдением, что расстояние между PAM и местом прикрепления транспозона показывает очень узкое распределение 16 . Однако механизм, управляющий интеграцией ДНК-груза, до конца не ясен. Были предложены две альтернативные модели 32,34 . В обоих случаях сборка филамента shTnsC будет работать как молекулярная линейка, направляющая shTnsB к месту прикрепления. Действительно, С-концевая область shTnsB взаимодействует с shTnsC 32,34 , и взаимодействие shTnsB-shTnsC приводит к разборке филамента. Однако одна модель предполагает, что филамент shTnsC растет из shCas12k, а ассоциация головки филамента с shTniQ указывает на место вставки shTnsB и груз ДНК 32 . Вторая модель предполагает, что филамент shTnsC растет в противоположном направлении, и комплекс shCas12k-shTniQ останавливает рост, рекрутируя shTnsB с грузом ДНК, тем самым продолжая интеграцию 34 . Обе модели ставят дополнительные вопросы относительно взаимодействия shTnsB-shTnsC. Как показано в структуре shTnsB-STC, не все протомеры имеют одинаковую конформацию, и неясно, все ли протомеры shTnsB, каталитически активные или неактивные протомеры, взаимодействуют с shTnsC (рис. 1d, e), поскольку в обоих случаях C -конец в структуре не визуализируется. Однако, по-видимому, он топологически доступен для дополнительных взаимодействий. В свете современных биохимических и структурных данных 32,34 , мы хотели бы предложить альтернативную гипотетическую рабочую модель (рис. 8), которая считает, что shTnsC и shTnsB будут связаны до того, как shTnsC взаимодействует с ДНК, чтобы избежать неспецифических взаимодействий с геномом, которые могут быть токсичными для клетка.
Однако одна модель предполагает, что филамент shTnsC растет из shCas12k, а ассоциация головки филамента с shTniQ указывает на место вставки shTnsB и груз ДНК 32 . Вторая модель предполагает, что филамент shTnsC растет в противоположном направлении, и комплекс shCas12k-shTniQ останавливает рост, рекрутируя shTnsB с грузом ДНК, тем самым продолжая интеграцию 34 . Обе модели ставят дополнительные вопросы относительно взаимодействия shTnsB-shTnsC. Как показано в структуре shTnsB-STC, не все протомеры имеют одинаковую конформацию, и неясно, все ли протомеры shTnsB, каталитически активные или неактивные протомеры, взаимодействуют с shTnsC (рис. 1d, e), поскольку в обоих случаях C -конец в структуре не визуализируется. Однако, по-видимому, он топологически доступен для дополнительных взаимодействий. В свете современных биохимических и структурных данных 32,34 , мы хотели бы предложить альтернативную гипотетическую рабочую модель (рис. 8), которая считает, что shTnsC и shTnsB будут связаны до того, как shTnsC взаимодействует с ДНК, чтобы избежать неспецифических взаимодействий с геномом, которые могут быть токсичными для клетка. Таким образом, образование филаментов shTnsC будет динамичным, так как присутствие shTnsB вызовет диссоциацию сборки в отсутствие shCas12k и shTniQ. В этом сценарии ассоциация shCas12k с ДНК-мишенью и shTniQ стабилизирует сборку комплексов shTnsC-shTsnB в месте вставки, а затем запускает интеграцию груза ДНК. Необходим дальнейший анализ ассоциации shTnsB-shTnsC, чтобы полностью понять интеграцию ДНК-груза в CAST типа V-K и подтвердить любую из этих рабочих моделей.
Таким образом, образование филаментов shTnsC будет динамичным, так как присутствие shTnsB вызовет диссоциацию сборки в отсутствие shCas12k и shTniQ. В этом сценарии ассоциация shCas12k с ДНК-мишенью и shTniQ стабилизирует сборку комплексов shTnsC-shTsnB в месте вставки, а затем запускает интеграцию груза ДНК. Необходим дальнейший анализ ассоциации shTnsB-shTnsC, чтобы полностью понять интеграцию ДНК-груза в CAST типа V-K и подтвердить любую из этих рабочих моделей.
ShTnsC связывается с ДНК, образуя филаменты, которые деполимеризуются с помощью shTnsB (слева). Учитывая взаимодействия shTnsC с shTnsB и shCas12k-shTniQ, возникают две возможности (штриховые квадраты). Во-первых, shTnsC, связанный с shTnsB, может рекрутироваться с помощью нацеливающего комплекса, включающего shCas12k и shTniQ (верхний пунктирный квадрат). Во-вторых, shTnsC может сначала связываться с нацеливающим комплексом, а затем рекрутировать shTnsB (нижний пунктирный квадрат). Обе возможности продуцируют комплекс перед транспозицией, активирующий активность интегразы shTnsB и приводящий к вставке транспозона.
Обе возможности продуцируют комплекс перед транспозицией, активирующий активность интегразы shTnsB и приводящий к вставке транспозона.
Полноразмерное изображение
Отсутствие TnsA в типе V-K приводит к дополнительным вопросам, касающимся промежуточного разрешения интеграции, поскольку нерасщепленные 5′-концы транспозона приводят к продуктам коинтеграции транспозиции 15,16 . Транспозоны, полагающиеся на DDE-транспозазы, имеют разные механизмы для решения этой проблемы 26,38,39 . Например, Tn5053 содержит резольвазу TniR, которая использует сайты res , также присутствующие в транспозоне, для разрешения коинтеграта 9.0072 40,41 , в то время как другие транспозазы генерируют шпильки для вырезания оставшейся ДНК 42 . Однако отсутствие домена вставки, который стабилизирует шпильку в каталитическом домене DDE shTnsB, не поддерживает эту возможность -16- для предотвращения коинтеграций. Кроме того, репликация ДНК и гомологичная рекомбинация могут разрешить коинтеграт в случае репликативной транспозиции 26 .
С другой стороны, наличие специфичной для хозяина резольвазы подтверждается тем фактом, что при транспозиции в E. coli , 20% продуктов внедрения являются коинтегратами 16 . Действительно, набор генов системы V-K (TnsB, TnsC и TniQ) сходен с набором генов Tn5053, который содержит TniA, TniB (гомологи TnsB и TnsC) и TniQ 16 , и можно предположить, что резольваза, не кодируемая в Локус транспозона VK, но все еще присутствующий в естественном хозяине, может иметь ту же активность, что и резольваза TniR. Необходимо провести дальнейшие исследования на S. hofmannii или других родственных цианобактериях, прежде чем мы сможем полностью понять РНК-управляемую транспозицию в типе V-K. Решение этих вопросов является ключом к успешному созданию новых комплексов CAST, способных интегрировать большие ДНК-грузы в геномы эукариот.
Наша структура обеспечивает механистическое понимание того, как CAST shTnsB распознает последовательности ДНК RE и LE транспозона в STC. Структура также показывает конформационную гимнастику белка shTnsB для сборки с ДНК и дает подробное представление об активном центре после катализа. Полное выяснение механизмов сборки shTnsB с остальными компонентами CAST потребуется, чтобы использовать системы CAST для РНК-управляемой геномной инженерии.
Структура также показывает конформационную гимнастику белка shTnsB для сборки с ДНК и дает подробное представление об активном центре после катализа. Полное выяснение механизмов сборки shTnsB с остальными компонентами CAST потребуется, чтобы использовать системы CAST для РНК-управляемой геномной инженерии.
Методы
Получение и очистка белков
Кодирующая последовательность (IDT) Scytonema hofmannii (UTEX 2349) tnsB (IDT) была клонирована с C -концевой гексагистидиновой (His)-меткой в вектор pET-21 (дополнительная таблица). Белки экспрессировали в клетках E. coli BL21 pRARE. культур E. coli выращивали при 37°C в среде Terrific Broth (TB) с 100 мг/л ампициллина до OD 600 0,6. Сверхэкспрессию белков индуцировали 250 мМ ИПТГ в течение 16–18 ч при 18°С. Клетки собирали центрифугированием, мгновенно замораживали в жидком азоте и хранили при -80°С. Затем осадки ресуспендировали в лизирующем буфере (50 мМ HEPES pH 7,5, 500 мМ NaCl, 5 мМ MgCl 9). 0144 2 , 1 мМ TCEP, 1 таблетка полного коктейля ингибиторов без ЭДТА (Roche) на 50 мл, 50 ЕД/мл бензоназы, 2 мг на 50 мл лизоцима). Ресуспендированные осадки перемешивали в течение 1 ч при 4°С. Лизис завершали обработкой ультразвуком, а лизат центрифугировали при 10 000 × g в течение 45 минут для разделения растворимой и нерастворимой фракций. Растворимую фракцию загружали в 5 мл колонку HisTrap FF Crude (Cytiva), уравновешенную в буфере IMAC-A (50 мМ HEPES, pH 7,5, 500 мМ NaCl, 1 мМ TCEP, 10 мМ имидазол, 1 таблетка полного коктейля ингибиторов, не содержащего ЭДТА ( Roche) на 500 мл), а связанные белки элюировали ступенчатым повышением концентрации имидазола буфером IMAC-B (50 мМ HEPES pH 7,5, 500 мМ NaCl, 1 мМ TCEP, 500 мМ имидазол, 1 таблетка коктейля «Полный ингибитор»). без ЭДТА (Roche) на 500 мл). Обогащенные белковые фракции объединяли, а концентрацию NaCl снижали до 200 мМ с использованием буфера для разбавления (50 мМ HEPES, pH 7,5, 50 мМ NaCl, 1 мМ TCEP).
0144 2 , 1 мМ TCEP, 1 таблетка полного коктейля ингибиторов без ЭДТА (Roche) на 50 мл, 50 ЕД/мл бензоназы, 2 мг на 50 мл лизоцима). Ресуспендированные осадки перемешивали в течение 1 ч при 4°С. Лизис завершали обработкой ультразвуком, а лизат центрифугировали при 10 000 × g в течение 45 минут для разделения растворимой и нерастворимой фракций. Растворимую фракцию загружали в 5 мл колонку HisTrap FF Crude (Cytiva), уравновешенную в буфере IMAC-A (50 мМ HEPES, pH 7,5, 500 мМ NaCl, 1 мМ TCEP, 10 мМ имидазол, 1 таблетка полного коктейля ингибиторов, не содержащего ЭДТА ( Roche) на 500 мл), а связанные белки элюировали ступенчатым повышением концентрации имидазола буфером IMAC-B (50 мМ HEPES pH 7,5, 500 мМ NaCl, 1 мМ TCEP, 500 мМ имидазол, 1 таблетка коктейля «Полный ингибитор»). без ЭДТА (Roche) на 500 мл). Обогащенные белковые фракции объединяли, а концентрацию NaCl снижали до 200 мМ с использованием буфера для разбавления (50 мМ HEPES, pH 7,5, 50 мМ NaCl, 1 мМ TCEP). Образец наносили на колонку HiTrap Heparin HP (GE Healthcare), уравновешенную буфером Heparin-A (50 мМ HEPES, pH 7,5, 200 мМ NaCl, 1 мМ TCEP). Белок элюировали линейным градиентом 0-100% буфера Heparin-B (50 мМ HEPES pH 7,5, 1 мМ NaCl, 1 мМ TCEP). Фракции, содержащие белок, объединяли и смешивали с TEV-протеазой для расщепления гистидиновой метки. Затем белок концентрировали и дополнительно очищали методом эксклюзионной хроматографии (SEC) с использованием колонки HiLoad 16/600 Superdex 200 (Cytiva), уравновешенной буфером SEC (50 мМ HEPES, pH 7,5, 500 мМ NaCl, 1 мМ TCEP). Фракции, содержащие чистый белок, объединяли, концентрировали до 9мг/мл, мгновенно замороженные в жидком азоте и хранящиеся при температуре -80 °C.
Образец наносили на колонку HiTrap Heparin HP (GE Healthcare), уравновешенную буфером Heparin-A (50 мМ HEPES, pH 7,5, 200 мМ NaCl, 1 мМ TCEP). Белок элюировали линейным градиентом 0-100% буфера Heparin-B (50 мМ HEPES pH 7,5, 1 мМ NaCl, 1 мМ TCEP). Фракции, содержащие белок, объединяли и смешивали с TEV-протеазой для расщепления гистидиновой метки. Затем белок концентрировали и дополнительно очищали методом эксклюзионной хроматографии (SEC) с использованием колонки HiLoad 16/600 Superdex 200 (Cytiva), уравновешенной буфером SEC (50 мМ HEPES, pH 7,5, 500 мМ NaCl, 1 мМ TCEP). Фракции, содержащие чистый белок, объединяли, концентрировали до 9мг/мл, мгновенно замороженные в жидком азоте и хранящиеся при температуре -80 °C.
Производство ДНК
Последовательности ДНК, соответствующие отдельным повторам, двойным повторам и одноцепочечной одноцепочечной ДНК, были приобретены у IDT. Молекулы ДНК, содержащие три или более повторов, были получены с помощью ПЦР с донорной ДНК, клонированной в pUC57 (Genewiz) в качестве матрицы (дополнительная таблица 1). Последовательности ДНК были выбраны из природного транспозона для CAST типа V-K в S. hofmannii , описанном в 4,7,21 . Полную ДНК донора амплифицировали с использованием праймеров 5′-tgctacgtctctacgtgtacagtgactaat-3′ и 5′ ccacaaaaggcgtagtgtacagtgacaaat-3′, RE амплифицировали с использованием праймеров 5′-tgctacgtctctacgtgtacagtgactaat-3′ и 5′-atacaaaagcgacagtcaatttgtcattatgaaa -attatattgatgacatttaatttgtcatcaattaattaag-3′ и 5′-ccacaaaaggcgtagtgtacagtgacaaat-3′. Амплифицированные последовательности очищали с использованием набора QIAquick PCR Purification Kit (Qiagen) и элюировали водой, не содержащей нуклеаз.
Последовательности ДНК были выбраны из природного транспозона для CAST типа V-K в S. hofmannii , описанном в 4,7,21 . Полную ДНК донора амплифицировали с использованием праймеров 5′-tgctacgtctctacgtgtacagtgactaat-3′ и 5′ ccacaaaaggcgtagtgtacagtgacaaat-3′, RE амплифицировали с использованием праймеров 5′-tgctacgtctctacgtgtacagtgactaat-3′ и 5′-atacaaaagcgacagtcaatttgtcattatgaaa -attatattgatgacatttaatttgtcatcaattaattaag-3′ и 5′-ccacaaaaggcgtagtgtacagtgacaaat-3′. Амплифицированные последовательности очищали с использованием набора QIAquick PCR Purification Kit (Qiagen) и элюировали водой, не содержащей нуклеаз.
Анализ сдвига электромобильности (EMSA)
ДНК (донорскую, RE, LE или LTR(6)-SR(1)) смешивали с shTnsB в буфере, содержащем 50 мМ HEPES pH 7,5, 300 мМ NaCl, 5 мМ MgCl 2 и 1 мМ TCEP. Концентрации ДНК и shTnsB указаны на дополнительных рисунках 3c – e. Образцы инкубировали при 37 °C в течение 30 мин и загружали в гели для замедления ДНК 6% (Thermo Fisher Scientific). ДНК выявляли окрашиванием геля GelRed (VWR). Затем тот же гель окрашивали InstantBlue (Life Technologies) для обнаружения присутствия shTnsB и сравнения сдвигов полос.
ДНК выявляли окрашиванием геля GelRed (VWR). Затем тот же гель окрашивали InstantBlue (Life Technologies) для обнаружения присутствия shTnsB и сравнения сдвигов полос.
Крио-ЭМ пробоподготовка
Прекаталитический комплекс готовили с использованием 1 мкМ РЭ, 1 мкМ LE и 8 мкМ shTnsB в 300 мкл буфера, содержащего 50 мМ HEPES pH 7,5, 300 мМ NaCl, 5 мМ MgCl. 2 и 1 мМ TCEP. Образец инкубировали в течение 30 мин при 37°С и концентрировали до 50 мкл в фильтрах 10 кДа. Восстановление STC показано на дополнительном рисунке 4 и проводилось следующим образом. Перенесенные и неперенесенные нити (TS и NTS) из RE (олигонуклеотиды RE_Target и RE_polyA в дополнительной таблице 1) и LE (олигонуклеотиды LE_Target и LE_polyA в дополнительной таблице 1) смешивали по отдельности, инкубировали при 95 °C в течение 5 мин и оставили на льду на 10 мин. Впоследствии комплементарная одноцепочечная ДНК-мишень была добавлена к двухцепочечной ДНК TS-NTS из RE и LE (олигонуклеотиды Target_1 и Target_2 в дополнительной таблице 1 соответственно). ДцДНК TS-NTS из RE и LE инкубировали отдельно при 70 ° C в течение 5 минут и оставляли на льду на 10 минут. Затем их перемешивали и инкубировали при комнатной температуре в течение 10 мин. Наконец, 50 мМ HEPES pH 7,5, 300 мМ NaCl, 5 мМ MgCl 2 и 1 мМ TCEP добавляли к 10 мкМ ДНК STC ДНК перед добавлением 40 мкМ shTnsB. Образец инкубировали в течение 30 мин при 37°С.
ДцДНК TS-NTS из RE и LE инкубировали отдельно при 70 ° C в течение 5 минут и оставляли на льду на 10 минут. Затем их перемешивали и инкубировали при комнатной температуре в течение 10 мин. Наконец, 50 мМ HEPES pH 7,5, 300 мМ NaCl, 5 мМ MgCl 2 и 1 мМ TCEP добавляли к 10 мкМ ДНК STC ДНК перед добавлением 40 мкМ shTnsB. Образец инкубировали в течение 30 мин при 37°С.
Сетки для обоих образцов (прекаталитический комплекс и STC) были приготовлены путем нанесения 3 мкл свежеприготовленного комплекса (разведение 1:2) на сетки с отверстиями UltraAuFoil 300 меш R1,2/1,3 (Quantifoil), подвергнутые тлеющему разряду в течение 60 с при 10 мА (Leica EM ACE200) и глубокой заморозке в жидком этане (предварительно охлажденном жидким азотом) с использованием Vitrobot Mark IV (FEI, Thermo Fisher Scientific — время блоттинга 3 с, влажность 100%, 4 °C) .
Крио-ЭМ обработка данных
Сбор данных осуществлялся с помощью EPU 2.12.1 (ThermoFisher). Обработку данных проводили с помощью программы cryoSPARC версии 3. 3.2 23 . Выполняли коррекцию движения заплаты и оценку CTF заплаты. Частицы 9646 K были первоначально отобраны из 4574 микрофотографий с использованием устройства для сбора капель с минимальным и максимальным диаметром 50 и 200 Å соответственно, а затем извлечены и отсортированы с помощью двух раундов 2D-классификации. Выбранные 2D-классы содержали 415 K частиц, извлеченных с использованием блока размером 416 × 416 пикселей. Трехмерная реконструкция ab initio использовалась в качестве начального объема для неравномерного уточнения, а полученные частицы были проанализированы с помощью трехмерного анализа изменчивости 43 (дополнительный рисунок 5), который выявил непрерывную конформационную изменчивость дистальных концов ДНК и близлежащих доменов shTnsB (N-концевая часть и CTD цепей A и B). Два наиболее пространственно отличающихся полученных объема использовались в качестве эталонов для гетерогенного уточнения и были дополнительно уточнены с помощью неоднородного (NU) уточнения до глобального разрешения 2,47 Å (частицы 269 K) и 2,81 Å (частицы 155 K) на основе золотой стандарт корреляции оболочки Фурье (GSFSC) 0,143 критерий отсечки (дополнительный рисунок 6).
3.2 23 . Выполняли коррекцию движения заплаты и оценку CTF заплаты. Частицы 9646 K были первоначально отобраны из 4574 микрофотографий с использованием устройства для сбора капель с минимальным и максимальным диаметром 50 и 200 Å соответственно, а затем извлечены и отсортированы с помощью двух раундов 2D-классификации. Выбранные 2D-классы содержали 415 K частиц, извлеченных с использованием блока размером 416 × 416 пикселей. Трехмерная реконструкция ab initio использовалась в качестве начального объема для неравномерного уточнения, а полученные частицы были проанализированы с помощью трехмерного анализа изменчивости 43 (дополнительный рисунок 5), который выявил непрерывную конформационную изменчивость дистальных концов ДНК и близлежащих доменов shTnsB (N-концевая часть и CTD цепей A и B). Два наиболее пространственно отличающихся полученных объема использовались в качестве эталонов для гетерогенного уточнения и были дополнительно уточнены с помощью неоднородного (NU) уточнения до глобального разрешения 2,47 Å (частицы 269 K) и 2,81 Å (частицы 155 K) на основе золотой стандарт корреляции оболочки Фурье (GSFSC) 0,143 критерий отсечки (дополнительный рисунок 6). Параметры расфокусировки для каждой частицы и параметры CTF для каждой группы были уточнены во время уточнения NU. При визуальном осмотре плотность карты 2,47 Å оказалась значительно более непрерывной. Эта карта была дополнительно улучшена на дистальных концах с помощью еще одной итерации трехмерного анализа изменчивости частиц 43 и использование наиболее пространственно различных объемов в качестве эталонов для еще одного раунда неоднородного уточнения. Большинство частиц попали в один класс (208 K), который был окончательно уточнен с помощью уточнения NU до карты 2,46 Å. Анализ 3DFSC показал сферичность 0,928 и диапазон углового разрешения 2,4–3,1 Å (дополнительный рисунок 6). Анализ локального разрешения был выполнен с помощью MonoRes 44 с использованием обертки cryoSPARC, который показал диапазон ~ 2–10 Å, при этом большая часть составляла около 2,5 Å, а дистальная часть — 4–8 Å. Построение модели выполнялось на картах, прошедших постобработку с помощью DeepEMhancer 0,13 45 .
Параметры расфокусировки для каждой частицы и параметры CTF для каждой группы были уточнены во время уточнения NU. При визуальном осмотре плотность карты 2,47 Å оказалась значительно более непрерывной. Эта карта была дополнительно улучшена на дистальных концах с помощью еще одной итерации трехмерного анализа изменчивости частиц 43 и использование наиболее пространственно различных объемов в качестве эталонов для еще одного раунда неоднородного уточнения. Большинство частиц попали в один класс (208 K), который был окончательно уточнен с помощью уточнения NU до карты 2,46 Å. Анализ 3DFSC показал сферичность 0,928 и диапазон углового разрешения 2,4–3,1 Å (дополнительный рисунок 6). Анализ локального разрешения был выполнен с помощью MonoRes 44 с использованием обертки cryoSPARC, который показал диапазон ~ 2–10 Å, при этом большая часть составляла около 2,5 Å, а дистальная часть — 4–8 Å. Построение модели выполнялось на картах, прошедших постобработку с помощью DeepEMhancer 0,13 45 . В качестве входных данных использовались полукарты, в то время как нормализация и маскирование/шумоподавление входных объемов выполнялись автоматически в режиме tightTarget, что давало карты со значительно улучшенной интерпретируемостью. Окончательная модель, построенная на картах DeepEMhancer, была окончательно проверена с использованием экспериментальных карт.
В качестве входных данных использовались полукарты, в то время как нормализация и маскирование/шумоподавление входных объемов выполнялись автоматически в режиме tightTarget, что давало карты со значительно улучшенной интерпретируемостью. Окончательная модель, построенная на картах DeepEMhancer, была окончательно проверена с использованием экспериментальных карт.
Построение и уточнение атомной модели
Первоначальная модель была создана на основе структуры MuA (PDB: 4FCY) с использованием автоматизированного сервера моделирования гомологии белковой структуры SWISS-MODEL 9.0072 46 . Это было твердое тело, встроенное в карту с помощью ChimeraX 47 . Подгонка модели была проверена и дополнительно подобрана в COOT 48 , а менее упорядоченные N- и C-концевые области были удалены. Впоследствии модель мономера shTnsB Alphafold 49 также использовалась для включения некоторых из этих областей в карту. Далее модель была перестроена de novo в COOT и уточнена с использованием phenix. real_space_refine 50 , а сервер гибкой настройки молекулярной динамики Namdinator использовался для ускорения и улучшения модели для сопоставления соответствия и уточнения модели как на ранней, так и на поздней стадии. этапы доработки 51 .
real_space_refine 50 , а сервер гибкой настройки молекулярной динамики Namdinator использовался для ускорения и улучшения модели для сопоставления соответствия и уточнения модели как на ранней, так и на поздней стадии. этапы доработки 51 .
Анализ транспозиции и мутагенез in vivo
Активность транспозиции CAST определяли с использованием векторов pHelper, pTarget и pDonor, полученных от Addgene (кат. № 127922, 127926 и 127924). Мутанты shTnsB (рис. 7) были созданы в векторе pHelper с использованием набора для клонирования In-Fusion от Takara и праймеров, содержащих желаемые точечные мутации. Анализы in vivo проводили в соответствии с Saito et al. 52 . Вкратце, по 12 нг pHelper, pTarget и pDonor были преобразованы в 30 мкл TransforMax EC100 pir+ Electrocompetent 9.0080 E. coli (Люциген). Клетки восстанавливали в течение 1 ч и высевали на среду LB, содержащую ампициллин, канамицин и хлорамфеникол. Клетки соскребали через 24 часа после посева, лизировали в 15 мкл буфера для лизиса колоний (TE с 0,1% Triton X-100), кипятили в течение 5 минут, разбавляли 30 мкл воды и вращали при 4000× г в течение 10 минут для осаждения. обломки. Супернатант использовали для последующего анализа.
обломки. Супернатант использовали для последующего анализа.
Анализ активности кПЦР
Частоту вставок относительно концентрации целевой плазмиды определяли с помощью кПЦР (TaqMan Fast Advanced Master Mix, Applied Biosytems) с набором праймеров/зонда (5′-6-FAM, Int ZEN и набор праймеров/зондов (5′-6-FAM, Int ZEN, 3′-Iowa Black, IDT), соответствующих продукту транспозиции (прямой праймер: 5′-ggttgagaagtcatttaataaggccactgttaaacg-3′, обратный праймер: 5′-aacgctgatgggtcacgacg- 3′, зонд: 5′-ctgtcgtcggtgacagattaatgtcattgtgac-3′) (рис. 7). Каждая реакция (11 мкл) содержала 2 мкл экстрагированных нуклеиновых кислот, 900 нМ прямого праймера, 900 нМ обратного праймера и 250 нМ зонда. Флуоресценцию измеряли с использованием прибора LightCycler 480 (Roche). Данные анализировали методом 2 –∆∆Ct с нормализацией по pTarget Ct.
Сводка отчета
Дополнительную информацию о дизайне исследования можно найти в Сводке отчета об исследовании природы, связанной с этой статьей.
Доступность данных
Реагенты, полученные в ходе этого исследования, доступны по запросу с заполненным Соглашением о передаче материалов. Атомные координаты и крио-ЭМ карты, включенные в это исследование, были депонированы в банке данных белков и банке данных электронной микроскопии под кодами доступа: PDB 8AA5, EMD-1529.4 и EMD-15344. Исходные данные qPCR и несрезанные гели предоставляются с этой статьей. Исходные данные приводятся вместе с настоящей статьей.
Список литературы
Макарова К.С. и др. Эволюционная классификация систем CRISPR-Cas: взрыв класса 2 и производные варианты. Нац. Преподобный Микробиолог. 18 , 67–83 (2020).
Артикул КАС пабмед Google Scholar
Дудна, Дж. А. Перспективы и проблемы терапевтического редактирования генома. Природа 578 , 229–236 (2020).

Артикул ОБЪЯВЛЕНИЯ КАС пабмед ПабМед Центральный Google Scholar
Фор, Г. и др. CRISPR-Cas в мобильных генетических элементах: контрзащита и не только. Нац. Преподобный Микробиолог. 17 , 513–525 (2019).
Артикул КАС пабмед Google Scholar
Петерс Дж. Э., Макарова К. С., Шмаков С., Кунин Е. В. Рекрутирование систем CRISPR-Cas Tn7-подобными транспозонами. Проц. Натл акад. науч. США 114 , E7358–E7366 (2017 г.).
Артикул ОБЪЯВЛЕНИЯ КАС пабмед ПабМед Центральный Google Scholar
Кунин Е.В., Макарова К.С. Происхождение и эволюция систем CRISPR-Cas. Филос. Транс. Р. Соц. Лонд. Б биол. науч. 374 , 20180087 (2019).
Артикул КАС пабмед ПабМед Центральный Google Scholar
«>Шмаков С. и др. Разнообразие и эволюция систем CRISPR-Cas класса 2. Нац. Преподобный Микробиолог. 15 , 169–182 (2017).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Klompe, S.E., Vo, PLH, Halpin-Healy, T.S. & Sternberg, S.H. Системы CRISPR-Cas, кодируемые транспозонами, обеспечивают прямую интеграцию ДНК под управлением РНК. Природа 571 , 219–225 (2019).
Hickman, A.B. et al. Неожиданное структурное разнообразие в рекомбинации ДНК: рестрикционная эндонуклеазная связь. Мол. Cell 5 , 1025–1034 (2000).
Артикул КАС пабмед Google Scholar
«>Stellwagen, AE & Craig, NL. Анализ мутантов с усилением функции АТФ-зависимого регулятора транспозиции Tn7. Дж. Мол. биол. 305 , 633–642 (2001).
Артикул КАС пабмед Google Scholar
Сарновский Р. Дж., Мэй Э. У. и Крейг Н. Л. Транспозаза Tn7 представляет собой гетеромерный комплекс, в котором активность разрыва и соединения ДНК распределена между различными генными продуктами. EMBO J. 15 , 6348–6361 (1996).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Питерс, Дж. Э. Tn7. Микробиолог. Спектр.
 2 , 1–20 (2014).
2 , 1–20 (2014).Кудувалли, П. Н., Рао, Дж. Э. и Крейг, Н. Л. Структура ДНК-мишени играет решающую роль в транспозиции Tn7. EMBO J. 20 , 924–932 (2001).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Райс, П. А., Крейг, Н. Л. и Дайда, Ф. Комментарий к «Вставка ДНК под управлением РНК с CRISPR-ассоциированными транспозазами». Наука , https://doi.org/10.1126/science.abb2022 (2020).
Стрекер, Дж., Ладха, А., Макарова, К.С., Кунин, Е.В. и Чжан, Ф. Ответ на комментарий к «РНК-управляемая вставка ДНК с CRISPR-ассоциированными транспозазами». Наука , https://doi.org/10.1126/science.abb2920 (2020).
Холдер, Дж. В. и Крейг, Н. Л. Архитектура посттранспозиционного комплекса Tn7: сложная нуклеопротеиновая структура. Дж. Мол. биол.
 401 , 167–181 (2010).
401 , 167–181 (2010).Артикул КАС пабмед ПабМед Центральный Google Scholar
Kaczmarska, Z. et al. Структурная основа распознавания концов транспозонов объясняет центральные особенности систем транспозиции Tn7. Мол. Сотовый , https://doi.org/10.1016/j.molcel.2022.05.005 (2022).
Дэвис Д. Р., Горышин И. Ю., Резникофф В. С. и Реймент И. Трехмерная структура промежуточного соединения синаптического комплекса Tn5. Наука 289 , 77–85 (2000).
Артикул ОБЪЯВЛЕНИЯ КАС пабмед Google Scholar
Ghanim, G.E., Kellogg, E.H., Nogales, E. & Rio, D.C. Структура комплекса транспозаза-ДНК P-элемента выявляет необычные структуры ДНК и контакты GTP-ДНК. Нац. Структура Мол. биол. 26 , 1013–1022 (2019).
Артикул КАС пабмед ПабМед Центральный Google Scholar
«>Montaño, S.P., Pigli, Y.Z. & Rice, P.A. Структура транспозосом μ проливает свет на эволюцию рекомбиназы DDE. Природа 491 , 413–417 (2012).
Артикул ОБЪЯВЛЕНИЯ пабмед ПабМед Центральный Google Scholar
Пуджани, А., Рубинштейн, Дж. Л., Флит, Д. Дж. и Брубейкер, М. А. cryoSPARC: алгоритмы для быстрого неконтролируемого определения структуры крио-ЭМ. Нац. Методы 14 , 290–296 (2017).
Артикул КАС пабмед Google Scholar
Маннервик М. Целевые гены гомеодоменовых белков. Bioessays 21 , 267–270 (1999).

Артикул КАС пабмед Google Scholar
Хикман, А. Б. и Дайда, Ф. Механизмы транспозиции ДНК. Микробиологический спектр. 3 , 0034–2014 (2015). МДНА3-.
Артикул Google Scholar
Siguier, P., Gourbeyre, E. & Chandler, M. Известные известные, известные неизвестные и неизвестные неизвестные в прокариотической транспозиции. Курс. мнение микробиол. 38 , 171–180 (2017).
Артикул КАС пабмед Google Scholar
Allingham, J.S., Pribil, P.A. & Haniford, D.B. Все три остатка каталитической триады транспозазы Tn 10 DDE функционируют в связывании ионов двухвалентных металлов. Дж. Мол. биол. 289 , 1195–1206 (1999).
Артикул КАС пабмед Google Scholar
«>Холм, Л. и Лааксо, Л. М. Обновление сервера Дали. Рез. нуклеиновых кислот . 44 , W351–W355 (2016 г.).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Xu, H. E. et al. Кристаллическая структура комплекса парный домен Pax6 человека и ДНК показывает специфическую роль линкерной области и карбоксиконцевого субдомена в связывании ДНК. Гены Дев. 13 , 1263–1275 (1999).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Benler, S.
 et al. Гены-карго Tn7-подобных транспозонов включают огромное разнообразие защитных систем, мобильных генетических элементов и генов устойчивости к антибиотикам. mBio 12 , e0293821 (2021).
et al. Гены-карго Tn7-подобных транспозонов включают огромное разнообразие защитных систем, мобильных генетических элементов и генов устойчивости к антибиотикам. mBio 12 , e0293821 (2021).Артикул пабмед Google Scholar
Querques, I., Schmitz, M., Oberli, S., Chanez, C. & Jinek, M. Выбор и ремоделирование целевых участков с помощью систем CRISPR-транспозонов типа V. Природа 599 , 497–502 (2021).
Артикул ОБЪЯВЛЕНИЯ КАС пабмед ПабМед Центральный Google Scholar
Xiao, R. et al. Структурные основы распознавания ДНК-мишени с помощью CRISPR-Cas12k для РНК-управляемой транспозиции ДНК. Мол. Сотовый 81 , 4457–4466.e4455 (2021 г.).
Артикул КАС пабмед Google Scholar
«>Халпин-Хили, Т. С., Кломпе, С. Э., Штернберг, С. Х. и Фернандес, И. С. Структурная основа нацеливания ДНК с помощью системы CRISPR-Cas, кодируемой транспозонами. Природа 577 , 271–274 (2020).
Артикул ОБЪЯВЛЕНИЯ КАС пабмед Google Scholar
Jia, N., Xie, W., de la Cruz, M.J., Eng, E.T. и Patel, D.J. Структурно-функциональное понимание начального этапа интеграции ДНК с помощью комплекса CRISPR-Cas-Transposon. Сотовые Res. 30 , 182–184 (2020).
Артикул КАС пабмед ПабМед Центральный Google Scholar
«>Холодий Г.Ю. и др. Четыре гена, два конца и res-область участвуют в транспозиции Tn5053: парадигма нового семейства транспозонов, несущих либо мероперон, либо интегрон. Мол. микробиол. 17 , 1189 (1995).
Артикул КАС пабмед Google Scholar
Николас Э. и др. Семейство репликативных транспозонов Tn3. Микробиолог. Спектр. 3 , 0060–2014 (2014). МДНК3.
Google Scholar
Петровский С. и Станисич В. А. Tn502 и Tn512 являются охотниками за res-сайтами, что свидетельствует о независимой от резольвазы транспозиции в случайные сайты.
 J. Бактериол. 192 , 1865–1874 (2010).
J. Бактериол. 192 , 1865–1874 (2010).Артикул КАС пабмед ПабМед Центральный Google Scholar
Минахина С., Холодий Г., Миндлин С., Юрьева О., Никифоров В. Транспозоны семейства Tn5053 являются охотниками за рез-сайтами, распознающими рез-сайты плазмид, занятые родственными резольвазами. Мол. микробиол. 33 , 1059 (1999).
Артикул КАС пабмед Google Scholar
Chen, Q. et al. Структурная основа бесшовного вырезания и специфического нацеливания транспозазой piggyBac. Нац. коммун. 11 , 3446 (2020).
Артикул ОБЪЯВЛЕНИЯ КАС пабмед ПабМед Центральный Google Scholar
Пунджани, А. и Флит, Д. Дж. Трехмерный анализ изменчивости: выявление непрерывной гибкости и дискретной неоднородности крио-ЭМ с одной частицей.
 Дж. Структура. биол. 213 , 107702 (2021).
Дж. Структура. биол. 213 , 107702 (2021).Артикул КАС пабмед Google Scholar
Вилас, Дж. Л. и др. MonoRes: автоматическая и точная оценка локального разрешения для карт электронной микроскопии. Структура 26 , 337–344.e334 (2018).
Артикул КАС пабмед Google Scholar
Sanchez-Garcia, R. et al. DeepEMhancer: решение для глубокого обучения для крио-ЭМ объемной постобработки. Комм. биол. 4 , 874 (2021).
Артикул пабмед ПабМед Центральный Google Scholar
Bienert, S. et al. Репозиторий SWISS-MODEL — новые возможности и функциональность. Рез. нуклеиновых кислот. 45 , Д313–д319 (2017).
Артикул КАС пабмед Google Scholar
«>Эмсли П., Локамп Б., Скотт В. Г. и Коутан К. Особенности и развитие Coot. Acta Кристаллогр. Д. биол. Кристаллогр. 66 , 486–501 (2010).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Бэк, М. и др. Точное предсказание белковых структур и взаимодействий с использованием трехдорожечной нейронной сети. Наука 373 , 871–876 (2021).
Артикул ОБЪЯВЛЕНИЯ КАС пабмед ПабМед Центральный Google Scholar
Adams, P.D. et al. PHENIX: комплексная система на основе Python для решения макромолекулярной структуры.
 Acta Кристаллогр. Д. биол. Кристаллогр. 66 , 213–221 (2010).
Acta Кристаллогр. Д. биол. Кристаллогр. 66 , 213–221 (2010).Артикул КАС пабмед ПабМед Центральный Google Scholar
Kidmose, R. T. et al. Namdinator — автоматическая молекулярно-динамическая гибкая подгонка структурных моделей к крио-ЭМ и кристаллографическим экспериментальным картам. IUCrJ 6 , 526–531 (2019).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Сайто, М. и др. Двойной режим CRISPR-ассоциированного самонаведения транспозонов. Cell 184 , 2441–2453.e2418 (2021).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Рыбарски Дж. Р., Ху К., Хилл А. М., Уилке К. О. и Финкельштейн И. Дж. Метагеномное открытие CRISPR-ассоциированных транспозонов. Проц. Натл акад. науч. США , https://doi.org/10.1073/pnas.2112279118 (2021).
Чой, К.Ю., Ли, Ю., Сарновский, Р. и Крейг, Н.Л. Прямое взаимодействие между субъединицами TnsA и TnsB контролирует гетеромерную транспозазу Tn7. Проц. Натл акад. науч. США 110 , E2038–E2045 (2013 г.).
Strecker, J. et al. Вставка ДНК под управлением РНК с CRISPR-ассоциированными транспозазами. Наука 364 , 48–53 (2019).
Артикул ОБЪЯВЛЕНИЯ Google Scholar
Peterson, G. & Reznikoff, W. Мутации активного сайта транспозазы Tn5 указывают на положение донорской основной ДНК в синаптическом комплексе. Дж. Биол. хим. 278 , 1904–1909 (2003).
Артикул КАС пабмед Google Scholar
Park, J. U. et al. Структурная основа для выбора сайта-мишени в системах транспозиции ДНК, управляемых РНК. Наука 373 , 768–774 (2021).
Артикул ОБЪЯВЛЕНИЯ КАС пабмед ПабМед Центральный Google Scholar
Skelding, Z., Queen-Baker, J. & Craig, N.L. Альтернативные взаимодействия между транспозазой Tn7 и белком, связывающим ДНК-мишень Tn7, регулируют иммунитет и транспозицию мишени. EMBO J. 22 , 5904–5917 (2003).
Артикул КАС пабмед ПабМед Центральный Google Scholar
Goddard, T.D. et al. UCSF ChimeraX: решение современных задач визуализации и анализа. Науки о белках. 27 , 14–25 (2018).
Артикул КАС пабмед Google Scholar
Скачать ссылки
Благодарности
Мы благодарим Датский национальный центр крио-ЭМ в CFIM в Копенгагенском университете и особенно Тиллманн Пейп за поддержку во время сбора крио-ЭМ данных. Мы также благодарим отдел экспрессии белков CPR за помощь в экспрессии и очистке белков. ФТК является участником Копенгагенской программы докторантуры по биологическим наукам (NNF19SA0035440). Г.М. является частью Центра белковых исследований (CPR) Фонда Ново Нордиск, который финансируется Фондом Ново Нордиск (грант NNF14CC0001). Эта работа также была поддержана грантом NNF0024386, грантом NNF17SA0030214 и грантами Distinguished Investigator (NNF18OC0055061) для GM, который является членом Кластера интегративной структурной биологии (ISBUC) в Копенгагенском университете.
Мы также благодарим отдел экспрессии белков CPR за помощь в экспрессии и очистке белков. ФТК является участником Копенгагенской программы докторантуры по биологическим наукам (NNF19SA0035440). Г.М. является частью Центра белковых исследований (CPR) Фонда Ново Нордиск, который финансируется Фондом Ново Нордиск (грант NNF14CC0001). Эта работа также была поддержана грантом NNF0024386, грантом NNF17SA0030214 и грантами Distinguished Investigator (NNF18OC0055061) для GM, который является членом Кластера интегративной структурной биологии (ISBUC) в Копенгагенском университете.
Информация об авторе
Примечания автора
Стефано Стелла
Текущий адрес: Twelve Bio ApS, Ole Maaløes Vej 3, 2200, Копенгаген, Дания
Авторы и филиалы
Группа структурной молекулярной биологии, Фонд Ново Нордиск, Центр белковых исследований, Факультет здоровья и медицинских наук of Copenhagen, 2200, Копенгаген, Дания
Франсиско Тенхо-Кастаньо, Николас Софос, Луиза С.
 Штуцке, Андерс Фульсанг, Стефано Стелла и Гильермо Монтойя
Штуцке, Андерс Фульсанг, Стефано Стелла и Гильермо МонтойяОтделение по очистке и характеристике белков, Исследовательский центр Ново Нордиск, Факультет Копенгагенского университета здоровья и медицинских наук, 2200, Копенгаген, Дания
Blanca López-Méndez
Авторы
- Francisco Tenjo-Castaño
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
- Nicholas Sofos
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
- Blanca López-Méndez
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Академия
- Luisa S. Stutzke
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
- Anders Fuglsang
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
- Stefano Stella
Посмотреть публикации автора
Вы также можете искать этого автора в PubMed Google Scholar
- Гильермо Монтойя
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Contributions
G. M. задумал исследование, F.T.C. и Г.М. поставил биохимические эксперименты. ФТК и С.С. установили протокол экспрессии и очистки. ФТК и AF охарактеризовали свойства связывания белок-ДНК. ФТК и Л.С. создали мутантов и провели эксперименты по транспозиции. ФТК и Б.Л.М. выполнил анализ SEC-MALS, F.T.C. подготовили сетки ЭМ и собрали крио-ЭМ изображения с Н.С. ФТК и Н.С. выполнил крио-ЭМ обработку данных, построил модели и Г.М. приступил к крио-ЭМ карте и анализу структуры с их помощью. Общие результаты обсуждались и оценивались со всеми авторами. Г.М. координировал и руководил проектом и написал рукопись с участием всех авторов.
M. задумал исследование, F.T.C. и Г.М. поставил биохимические эксперименты. ФТК и С.С. установили протокол экспрессии и очистки. ФТК и AF охарактеризовали свойства связывания белок-ДНК. ФТК и Л.С. создали мутантов и провели эксперименты по транспозиции. ФТК и Б.Л.М. выполнил анализ SEC-MALS, F.T.C. подготовили сетки ЭМ и собрали крио-ЭМ изображения с Н.С. ФТК и Н.С. выполнил крио-ЭМ обработку данных, построил модели и Г.М. приступил к крио-ЭМ карте и анализу структуры с их помощью. Общие результаты обсуждались и оценивались со всеми авторами. Г.М. координировал и руководил проектом и написал рукопись с участием всех авторов.
Автор, ответственный за переписку
Переписка с Гильермо Монтойя.
Декларации этики
Конкурирующие интересы
G.M. и S.S. являются соучредителями и членами Twelve Bio BoD. Остальные авторы заявляют об отсутствии конкурирующих интересов.
Рецензирование
Информация о рецензировании
Nature Communications благодарит анонимных рецензентов за их вклад в рецензирование этой работы. Доступны отчеты рецензентов.
Доступны отчеты рецензентов.
Дополнительная информация
Примечание издателя Springer Nature остается нейтральной в отношении юрисдикционных претензий в опубликованных картах и институциональной принадлежности.
Дополнительная информация
Дополнительная информация
Файл рецензирования
Описание дополнительных дополнительных файлов
САМОЧКА ПРИМЕНЕНИЯ 1
9666666666.0003 Исходные данные
Исходные данные
Права и разрешения
Открытый доступ Эта статья находится под лицензией Creative Commons Attribution 4.0 International License, которая разрешает использование, совместное использование, адаптацию, распространение и воспроизведение на любом носителе или в любом формате. , при условии, что вы укажете первоначальных авторов и источник, предоставите ссылку на лицензию Creative Commons и укажите, были ли внесены изменения. Изображения или другие сторонние материалы в этой статье включены в лицензию Creative Commons для статьи, если иное не указано в кредитной строке материала. Если материал не включен в лицензию Creative Commons статьи, а ваше предполагаемое использование не разрешено законом или выходит за рамки разрешенного использования, вам необходимо получить разрешение непосредственно от правообладателя. Чтобы просмотреть копию этой лицензии, посетите http://creativecommons.org/licenses/by/4.0/.
Изображения или другие сторонние материалы в этой статье включены в лицензию Creative Commons для статьи, если иное не указано в кредитной строке материала. Если материал не включен в лицензию Creative Commons статьи, а ваше предполагаемое использование не разрешено законом или выходит за рамки разрешенного использования, вам необходимо получить разрешение непосредственно от правообладателя. Чтобы просмотреть копию этой лицензии, посетите http://creativecommons.org/licenses/by/4.0/.
Перепечатки и разрешения
Об этой статье
vk-mem-erupt — библиотека Rust gfx // Lib.rs
2 выпуска
0.2.4 25 окт. 2021 г. 0.2.3 23 июля 2021 г.
# 172 в графических API
Массачусетский технологический институт/Апач 305 КБ
4. 5К SLoC
5К SLoC
Это форк vk-mem-rs Грэма Вихлидала, но с использованием извержения вместо ash для привязки Vulkan. Таким образом, вся работа над этим ящиком выполняется Грэмом Вихлидалом и командой, стоящей за библиотекой VMA. Я просто модифицировал ящик таким образом, чтобы его можно было использовать вместе с креплениями извержения Вулкана.
- вк-мем: (https://github.com/gwihlidal/vk-mem-rs)
- извержение: (https://gitlab.com/Friz64/erupt)
Этот крейт предоставляет слой FFI и идиоматические оболочки ржавчины для превосходной библиотеки AMD Vulkan Memory Allocator (VMA) C/C++.
- Документация
- Примечания к выпуску
- ВМА GitHub
- Документация VMA
- Открытие графического процессора Объявление
- Открытое обновление графического процессора
Проблема
Выделение памяти и создание ресурсов (буфера и изображения) в Vulkan затруднено (по сравнению со старыми графическими API-интерфейсами, такими как D3D11 или OpenGL) по нескольким причинам:
- Требуется много шаблонного кода, как и все остальное в Vulkan, потому что это низкоуровневый и высокопроизводительный API.

- Существует дополнительный уровень косвенности: VkDeviceMemory выделяется отдельно от создания VkBuffer/VkImage, и они должны быть связаны вместе. Привязку нельзя изменить позже — ресурс необходимо создать заново.
- Необходимо запросить у драйвера поддерживаемые кучи памяти и типы памяти. Разные IHV обеспечивают разные его типы.
- Рекомендуется выделять большие блоки памяти и назначать их части определенным ресурсам.
Особенности
Этот ящик может помочь разработчикам игр управлять выделением памяти и созданием ресурсов, предлагая некоторые функции более высокого уровня:
- Функции, помогающие выбрать правильный и оптимальный тип памяти на основе предполагаемого использования памяти.
- Требуемые или предпочтительные характеристики памяти выражаются с использованием описания более высокого уровня по сравнению с флагами Vulkan.
- Функции, которые выделяют блоки памяти, резервируют и возвращают пользователю их части (VkDeviceMemory + offset + size).

- Библиотека отслеживает выделенные блоки памяти, используемые и неиспользуемые диапазоны внутри них, находит наиболее подходящие неиспользуемые диапазоны для новых выделений, соблюдает все правила выравнивания и детализации буфера/изображения.
- Функции, которые могут создавать образ/буфер, выделять для него память и связывать их вместе — все за один вызов.
Дополнительные характеристики:
- Кросс-платформенный
- Окна
- Линукс
- macOS (MoltenVK)
- Хорошо протестированный и задокументированный API
- Базовая библиотека включает ряд коммерческих игр.
- Обширная документация (включая полные описания алгоритмов в репозитории VMA)
- Поддержка пользовательских пулов памяти:
- Создание пула с желаемыми параметрами (например, фиксированный или ограниченный максимальный размер)
- Выделите из него память.
- Поддержка линейной или партнерской стратегии распределения
- Создайте пул с линейным алгоритмом и используйте его для гораздо более быстрого выделения и освобождения памяти в режиме «свободно сразу», в стеке, двойном стеке или кольцевом буфере.

- Подробная статистика:
- Глобально, для кучи памяти и для каждого типа памяти.
- Объем используемой памяти
- Объем неиспользуемой памяти
- Количество выделенных блоков
- Количество распределений
- и т. д.
- Аннотации отладки:
- Свяжите строку с именем или непрозрачным указателем на ваши собственные данные при каждом выделении.
- Дамп JSON:
- Получить строку в формате JSON с подробной картой внутреннего состояния, включая список выделений и промежутков между ними.
- Преобразуйте этот дамп JSON в изображение, чтобы визуализировать свою память. См. инструменты/VmaDumpVis.
- Поддержка отображения памяти:
- Внутренний подсчет ссылок.
- Поддержка постоянно отображаемой памяти; просто выделите соответствующий флаг, и вы получите доступ к сопоставленному указателю.
- Поддержка распределения дефрагментации:
- Вызовите одну функцию и позвольте библиотеке перемещать данные, чтобы освободить некоторые блоки памяти и сделать ваши выделения более компактными.

- Поддержка потерянных выделений:
- Выделите память с соответствующими флагами и позвольте библиотеке удалить выделения, которые не используются для многих кадров, чтобы освободить место для новых.
- Поддержка несогласованного выделения памяти и сброса:
-
nonCoherentAtomSize учитывается автоматически.
- Поддержка попытки обнаружить неправильное использование отображаемой памяти:
- Включить инициализацию всей выделенной памяти с помощью битового шаблона для обнаружения использования неинициализированной или освобожденной памяти.
- Включить проверку магического числа до и после каждого выделения памяти для обнаружения выхода за пределы памяти.
Планируемые функции
- Обширные модульные тесты и примеры.
- Уже проведены некоторые модульные тесты, но не полное покрытие
- Пример не написан - вероятно, образец VMA будет портирован на
ash и vk_mem
- Запись и воспроизведение выделений для углубленного анализа использования памяти, переходов ресурсов и т.
 д.
д.- Проверка правильности, измерение производительности и сбор статистики.
Пример
Основное использование этого ящика очень простое; дополнительные функции являются необязательными.
После создания экземпляра vk_mem::Allocator для создания буфера требуется очень мало кода:
// Создать буфер (только GPU, в данном примере 16 КБ)
пусть create_info = vk_mem::AllocationCreateInfo {
использование: vk_mem::MemoryUsage::GpuOnly,
..По умолчанию::по умолчанию()
};
пусть (буфер, распределение, информация_распределения) = распределитель
.создать_буфер(
&ash::vk::BufferCreateInfo::builder()
.размер(16 * 1024)
.usage(ash::vk::BufferUsageFlags::VERTEX_BUFFER | ash::vk::BufferUsageFlags::TRANSFER_DST)
.строить(),
&create_info,
)
.развернуть();
// Делаем что-то с буфером! (тип ash::vk::Buffer)
// Уничтожаем буфер
allocator.destroy_buffer(buffer, &allocation). unwrap();
unwrap();
С этим вызовом одной функции ( vk_mem::Allocator::create_buffer ):
-
ash::vk::Buffer ( VkBuffer ) создан. -
ash::vk::DeviceMemory ( VkDeviceMemory ) блок выделяется при необходимости. - К этому буферу привязана неиспользуемая область блока памяти.
-
Создается vk_mem::Allocation , представляющий память, назначенную этому буферу. Его можно запросить для таких параметров, как дескриптор памяти Vulkan и смещение.
РасплавленныйВК
Для MoltenVK на macOS вам необходимо установить правильные переменные среды. Что-то вроде:
экспорт SDK_PATH=/path/to/vulkansdk-macos-1.1.106.0
экспортировать DYLD_LIBRARY_PATH=$SDK_PATH/macOS/lib
экспортировать VK_ICD_FILENAMES=$SDK_PATH/macOS/etc/vulkan/icd.d/MoltenVK_icd.json
экспортировать VK_LAYER_PATH=$SDK_PATH/macOS/etc/vulkan/explicit_layer.d
грузовой тест
Использование
Добавьте это к вашему Cargo. toml
toml :
[зависимости]
вк-мем = "0.2.3"
и добавьте это в корень вашего ящика:
внешний крейт vk_mem;
Компиляция с использованием MinGW W64
Vulkan Memory Allocator требует потоков C++11.
MinGW W64 не поддерживает их по умолчанию, поэтому вам нужно переключиться на сборку posix.
Например, в Debian вам нужно запустить следующее:
альтернативы обновления --set x86_64-w64-mingw32-gcc /usr/bin/x86_64-w64-mingw32-gcc-posix
альтернативы обновления --set x86_64-w64-mingw32-g++ /usr/bin/x86_64-w64-mingw32-g++-posix
альтернативы обновления --set i686-w64-mingw32-gcc /usr/bin/i686-w64-mingw32-gcc-posix
альтернативы обновления --set i686-w64-mingw32-g++ /usr/bin/i686-w64-mingw32-g++-posix
Лицензия
Лицензия под любой из
- Лицензия Apache, версия 2.0 (LICENSE-APACHE или http://www.apache.org/licenses/LICENSE-2.0)
- Лицензия MIT (LICENSE-MIT или http://opensource.org/licenses/MIT)
на ваш выбор.
Кредиты и особая благодарность
- Адам Савицки - AMD (автор библиотеки C/C++)
- Майк Кляйн (Автор пепла - Vulkan ржавчины крепления)
- Йохан Андерссон (взносы)
- Патрик Миноуг (взносы)
- Лейл Конвей (взносы)
- благодарностей (взносы)
- Хенрик Ридгорд (Вклады)
- msiglreith (Вклады)
- Максим Павленко (Вклад)
- Брэндон Поллак (взносы)
Взнос
Если вы прямо не укажете иное, любой преднамеренно отправленный вклад
для включения вами в этот крейт, как определено в лицензии Apache-2.0,
иметь двойную лицензию, как указано выше, без каких-либо дополнительных условий.
Пожертвования всегда приветствуются; загляните в систему отслеживания проблем, чтобы узнать, какие известные улучшения задокументированы.
Кодекс поведения
Вклад в крейт вк-мем организован на условиях
Участник Covenant, сопровождающий vk-mem, @gwihlidal, обещает
вмешаться, чтобы поддержать этот кодекс поведения.
Зависимости
~6,5 МБ
~149K SLoC
VK Modelle Binding Mercedes Benz O 405 K81060
Чтобы иметь возможность использовать TRUCKMO Truck Models — Your Truck Models spezialist в полном объеме, мы рекомендуем активировать Javascript в вашем браузере.
- Закрыть меню
€16,88 *
Цены без учета НДС плюс стоимость доставки
Готов к отправке сегодня,
Срок поставки ок. 1-3 рабочих дня
1-3 рабочих дня
Просмотрено
Функциональный
Активный
Неактивный
Функциональные файлы cookie необходимы для функционирования интернет-магазина. Эти файлы cookie присваивают вашему браузеру случайный уникальный идентификатор, чтобы обеспечить беспрепятственный процесс совершения покупок при нескольких просмотрах страниц.
Сеанс:
Сеансовые файлы cookie сохраняют ваши данные о покупках при нескольких просмотрах страниц и поэтому необходимы для вашего личного опыта покупок.
Список желаний:
Файл cookie позволяет сделать список желаний доступным для пользователя во время сеансов. Это означает, что список желаний остается на месте даже в течение нескольких сеансов браузера.
Назначение устройства:
Назначение устройства помогает магазину обеспечить наилучшую выкладку для текущего размера активной витрины.
Токен CSRF:
Файл cookie токена CSRF способствует вашей безопасности. Усиливает защиту форм от нежелательных хакерских атак.
Токен входа:
Токен входа используется для распознавания пользователей в сеансах. Этот файл cookie не содержит никаких личных данных, но обеспечивает персонализацию в течение нескольких сеансов браузера.
Исключение кэша:
Файлы cookie исключений кэша позволяют пользователям читать отдельное содержимое независимо от кэш-памяти.
Файлы cookie Активная проверка:
Этот файл cookie используется веб-сайтом, чтобы узнать, разрешены ли файлы cookie браузером пользователя сайта.
Настройки файлов cookie:
Этот файл cookie используется для хранения настроек файлов cookie пользователя сайта в течение нескольких сеансов браузера.
Информация об источнике:
Файл cookie сохраняет исходную страницу пользователя и первую посещенную страницу для дальнейшего использования.
Активированные файлы cookie:
Сохраняет файлы cookie, которые пользователь уже принял в первый раз.
Полоса:
Этот файл cookie используется платежной системой для повышения безопасности при обработке платежей на веб-сайте.
Маркетинг
Активный
Неактивный
Маркетинговые файлы cookie используются для целенаправленного и индивидуального отображения рекламы на веб-сайте в течение нескольких просмотров страниц и сеансов браузера.
Файлы cookie AddThis:
AddThis кнопки общего доступа, инструменты таргетинга и предложения контента.
Активный
Неактивный
Интеграция AddThis:
AddThis включается только в том случае, если этот файл cookie был принят.
Активный
Неактивный
Отслеживание
Активный
Неактивный
Отслеживающие файлы cookie помогают оператору магазина собирать и оценивать информацию о поведении пользователей на своем веб-сайте.
Google Analytics:
Google Analytics используется для анализа трафика веб-сайта. В результате можно создавать и считывать статистику активности на сайте.
Активный
Неактивный
Диспетчер тегов Google:
Файл cookie управляет интеграцией Диспетчера тегов Google. Дополнительные скрипты и инструменты отслеживания интегрируются на страницу через Диспетчер тегов Google.
Активный
Неактивный
Служба
Активный
Неактивный
Сервисные файлы cookie используются для предоставления пользователю дополнительных предложений, например. живые чаты на сайте. Информация, полученная с помощью этих служебных файлов cookie, также может обрабатываться в целях анализа страницы.
живые чаты на сайте. Информация, полученная с помощью этих служебных файлов cookie, также может обрабатываться в целях анализа страницы.
Видео YouTube:
Видео YouTube включаются только в том случае, если этот файл cookie был принят.
Активный
Неактивный
Этот веб-сайт использует файлы cookie, чтобы предоставить вам наилучшие функциональные возможности.
Dr V K Bind - Лучшие врачи хирурги-ортопеды в Чунаре
АДРЕС
Госпиталь Навдживан График работы OPD, Чунар:
Дней Время начала Время окончания Понедельник 9:00 утра 11:00 17:00 19:00 вторник 9:00 11:00 17:00 19:00 Среда 9:00 11:00 17:00 19:00 Четверг 9:00 11:00 17:00 19:00 Пятница 9:00 11:00 17:00 19:00 Суббота 9:00 11:00 17:00 19:00
О докторе
Доктор В. К. Бинд является одним из врачей-ортопедов в Чунаре . Доктор В. К. Бинд является консультантом по специальности , хирургом-ортопедом, специалистом в больнице Навдживан .
Больница Навдживан расположена по адресу: г. ХХХХХХ ХХХХХХ ХХХХХХХ 000000 ОПД доктора.
V K Bind находится в больнице с понедельника по субботу.
К. Бинд является одним из врачей-ортопедов в Чунаре . Доктор В. К. Бинд является консультантом по специальности , хирургом-ортопедом, специалистом в больнице Навдживан .
Больница Навдживан расположена по адресу: г. ХХХХХХ ХХХХХХ ХХХХХХХ 000000 ОПД доктора.
V K Bind находится в больнице с понедельника по субботу.
Написать отзыв
★
★
★
★
★
отзывов
WhatsApp
Звоните
Свяжитесь со своей российской аудиторией с помощью VK Messenger
ПРИЛОЖЕНИЯ ДЛЯ ОБМЕНА СООБЩЕНИЯМИ
Свяжитесь со своей российской аудиторией с помощью самого
популярного приложения для обмена сообщениями в стране.
ПОПРОБУЙ
66,6 млн
мобильных активных пользователей Янг, 9 лет1198 городских пользователей
5 миллиардов
сообщений ежедневно 80+ поддерживаемых языков
Используйте другой подход для привлечения клиентов
в разных уголках мира с помощью нашего VK Messenger API Мощный API
Общайтесь со своими клиентами по всему миру. WayMore создал простой, безопасный и надежный API для VK Messenger, который поможет вам улучшить вашу коммуникационную игру.
Активное участие
Отправляйте и получайте мультимедийные сообщения, содержащие текст, изображения, кнопки, смайлики, наклейки, видео- и аудиоконтент или файлы любого типа. Общайтесь любым доступным способом.
Круглосуточная поддержка
Мы очень серьезно относимся к поддержке. Мы предоставляем нашим клиентам круглосуточную службу поддержки 365 дней в году по всему миру. Ничего меньшего недостаточно.
Ничего меньшего недостаточно.
Более высокие скорости доставки
Убедитесь, что ваше сообщение достигнет адресата и цели. Оптимизируйте скорость доставки, чтобы ваши сообщения отправлялись, доставлялись и читались с возможностью аварийного переключения на другие каналы обмена сообщениями.
Доступно аварийное переключение
Никогда не упускайте возможности связаться со своей аудиторией. Если ваши сообщения не прочитаны, вы можете использовать интеллектуальные решения WayMore для переключения на другой канал.
Инсайты в режиме реального времени
Обратные звонки доставки для победы. Они помогут вам лучше понять рентабельность инвестиций и клиентов.
Получите больше с мессенджером ВКонтакте
Обеспечьте успешную поддержку клиентов
Будьте там, где ваши клиенты. Покажите им, что вы будете рядом, несмотря ни на что, и поощряйте двустороннюю связь между ними и вашей службой поддержки по каналу, который они используют чаще всего.
Увеличьте свой
клиентская база Превратите своих существующих клиентов в промоутеров с помощью социальных приглашений по каналам для мгновенного общения.
Оптимизация коэффициентов конверсии
Взаимодействуйте со своими клиентами с помощью персонализированных рекламных сообщений, которые содержат не только текст. Используйте видео, гифки, изображения или ссылки и обязательно привлеките их внимание.
Общайтесь с
вашей аудиторией Держите своих клиентов в курсе. Отправляйте важную информацию и напоминания о событиях, на которые подписались люди, используя VK Messenger API.
Активируйте качественных лидов
Взаимодействуйте с большим количеством людей и генерируйте качественных лидов, используя API VK Messenger и его популярность в нескольких странах.
Улучшить качество обслуживания клиентов
Превзойти ожидания. Доставляйте согласованные персонализированные сообщения и повышайте качество обслуживания клиентов. Мы можем помочь вам начать работу в кратчайшие сроки. Не теряйте больше времени.
Мы можем помочь вам начать работу в кратчайшие сроки. Не теряйте больше времени.
Вы в 4 шагах от создания кампании в мессенджере ВКонтакте
1. Войдите в свою учетную запись Waymore.
Если у вас нет учетной записи, зарегистрируйтесь сейчас.
2. Пополните свой счет
Пополнить счет с помощью Visa, Mastercard, PayPal.
3. Начните свою кампанию
Нажмите «Общение» и «Мессенджер ВКонтакте» в левом меню главной панели
и начните свою кампанию в мессенджере ВКонтакте.
4. Зарегистрируйте идентификатор отправителя и используйте сервис рассылок VK messenger для рассылки своей рассылки.
Просто заполните форму со всеми необходимыми данными (информация о компании, информация о кампании, информация о сообщении, функции мультимедийного брендинга).
Мощный маркетинг.
Простое ценообразование. Никаких обязательных договоров, никаких скрытых платежей.
Мы всегда будем честны с нашими расходами.
